Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Boletín de la Sociedad Argentina de Botánica
versión On-line ISSN 1851-2372
Bol. Soc. Argent. Bot. vol.53 no.2 Córdoba jun. 2018
Genética y Evolución - Genetics and Evolution
Tamaño Del Genoma, Características Cariotípicas Y Tamaño Del Polen En Especies Argentinas De Spergula (Caryophyllaceae)
MARTA CAROLINA BREM1*, JUAN PABLO COULLERI1, CAROLA REGINA VOLPONI2 y MARÍA SILVIA FERRUCCI1
1 Instituto de Botánica del Nordeste, Consejo Nacional de Investigaciones Científicas y Técnicas –Universidad Nacional del Nordeste, Sargento Cabral 2131, Corrientes, Capital, Argentina.
2 Comisión de Investigaciones Científicas, prov. de Buenos Aires, Facultad de Ciencias Naturales y Museo de la Universidad Nacional de La Plata, Avenida 122 y 60, La Plata, Buenos Aires, Argentina.
*Autor correspondiente: martacarolinabrem@gmail.com
Resumen: El objetivo de este trabajo fue estimar el contenido de ADN (2C) en cuatro especies de Spergula que habitan Argentina y analizar la variación del tamaño del genoma (Cx) en relación con las características cariotípicas y palinológicas. Se analizó por primera vez el contenido de ADN de cinco taxones y el tamaño del genoma de cuatro, que variaron muy poco entre las especies, desde Cx= 0,458 pg (S. salina, 4x) a Cx= 0,856 pg (S. arvensis, 2x). Se registraron dos nuevos recuentos cromosómicos para el género y el tamaño del polen varió de pequeños a medianos. Los coeficientes de regresión sugieren que el número de cromosomas varía de manera inversamente proporcional al tamaño del genoma, mientras que el tamaño del genoma presenta una baja relación con la longitud cromosómica total (LCT) del genoma diploide, aunque muestra una alta relación con respecto a la longitud cromosómica promedio (LCP). En tanto, el tamaño del polen varía independientemente con respecto al tamaño del genoma. La variación cariotípica detectada podría deberse a cambios numéricos y estructurales durante la evolución del género, mientras que el tamaño del polen podría estar relacionado con presiones de selección asociadas a estrategias de dispersión.
Palabras clave: Spergula, Argentina, contenido de ADN, nivel de ploidía, tamaño cromosómico, tamaño del polen.
Summary: Genome size, karyotypic features and pollen size in Argentinian species of Spergula (Caryophyllaceae). The objective of this work was to estimate the DNA content (2C) in four species of Spergula that inhabit Argentina and to analyze the variation of genome size (Cx) in relation to karyotypic and palynological features. The DNA content of fve taxa and the genome size of four, which varied very little between species, were analyzed for the frst time from Cx= 0.458 pg (S. salina, 4x) to Cx= 0.856 pg (S. arvensis, 2x). Two new chromosomal counts were recorded for the genus and the pollen size varied from small to medium. The regression coefficients suggest that the chromosome number varies inversely proportional in regards to the genome size, while the genome size presents a low relation with the total chromosome length (TCL) of the diploid genome, although it shows a high relation with respect to the average chromosome length (ACL). Meanwhile, the pollen size varies independently regarding to the genome size. The detected karyotypic variation could be due by numerical and structural changes during the genus evolution, while the pollen size could be related with selection pressures associated with dispersal strategies.
Key words: Spergula, Argentina, DNA content, ploidy level, chromosome size, pollen size.
Introducción
Spergula L. pertenece a la familia Caryophyllaceae Juss., subfamilia Paronychoideae (A. L. Juss.) Meisn., son hierbas anuales, raramente perennes. Comprende alrededor de 70 especies de distribución casi cosmopolita, cuyos centros de diversidad se localizan en la región del Mediterráneo y en Chile (Pedersen, 1984). En Argentina, Spergula se halla representada por 12 especies, dos subespecies y dos variedades (Zuloaga et al., 2008; Brem, 2017), éstas son mayoritariamente halóftas, comunes en pastizales y algunas de alta montaña (Pedersen, 1984; Volponi, 1994).
Spergularia (Pers.) Pers. ex J. Presl & C. Presl es un género muy afín a Spergula, su estatus taxonómico ha sido controvertido debido a la presencia de caracteres superpuestos o graduales con Spergula (Pedersen, 1984; González, 2010; Brem, 2017); tal es así que actualmente varias especies de Spergularia se circunscriben a Spergula, como S. bocconii (Scheele) Pedersen, S. diandra (Guss.) Murb. y S. salina (J. Presl & C. Presl) D. Dietr., entre otras.
La poliploidía es una alteración cromosómica en la cual un organismo posee más de dos juegos cromosómicos completos, pudiendo ser 2n= 14 (diploide), 28 (tetraploide), 42 (hexaploide), 56 (octoploide), etc., donde n representa el número de cromosomas en los gametos. Los números somáticos se basan en el número de cromosomas gaméticos de las especies diploides, que se conoce como el número cromosómico básico (x); en una especie diploide n= x, pero en una especie poliploide n es un múltiplo de x. La inferencia del número básico de una serie poliploide es un paso importante para la comprensión del número cromosómico en la formulación de una hipótesis evolutiva (Kenyon, 1997). La evolución del número cromosómico en Caryophyllaceae fue investigada por Fernandes & Leitão (1971), estos autores suponen que Caryophyllaceae desciende de Phytolaccaceae vía Molluginaceae con un número básico principalmente de x= 9, considerando a éste ancestral para la familia. A partir de este número básico ocurre una serie descendente en Paronychoideae de x= 5 y en Alsinoideae de x= 6; y una serie ascendente hasta x= 12 en Caryophylloideae. Respecto a las subfamilias,
Paronychoideae es la menos variable, caracterizada por los números básicos x= 8 y x= 9, y menos frecuente x= 7 en algunas especies de Paronychia Mill. y Chaetonychia cymosa (L.) Sweet, o incluso x= 5 en Illecebrum vertidllatum L., la principal causa de variación es la poliploidía (Ratter, 1976; Bittrich, 1993).
La cantidad de ADN en un genoma nuclear gamético no replicado se conoce como el "valor 1C" o "valor C", comúnmente llamado tamaño del genoma (Soltis et al, 2003). Esta magnitud es igual al contenido de ADN nuclear 2C dividido por el nivel de ploidía (Bennett et al., 1998). Sin embargo, la terminología presenta dificultades en su uso en organismos de diferentes niveles de ploidía, por ello Greilhuber et al. (2005) proponen una terminología unificada que describe el tamaño del genoma con facilidad y sin ambigüedad. Estos autores sugieren el término completo "tamaño del genoma monoploide" y el término abreviado "valor Cx" para el contenido de ADN del genoma monoploide en poliploides y no poliploides, sosteniendo que el valor C y el valor Cx son congruentes.
La estimación del contenido de ADN ha facilitado los estudios de evolución y especiación en numerosos grupos de plantas debido a que tanto el número cromosómico como el tamaño del genoma varían considerablemente en las plantas con flores (Soltis et al, 2003). En las angiospermas, el contenido de ADN nuclear (2C) varía entre 1 y 125 pg por núcleo diploide (Bennett & Leitch, 2012), dicha variabilidad llevo a Leitch et al. (2005) a proponer 5 categorías para tamaño del genoma: muy pequeños (<1,4 pg), pequeños (<3,5 pg), intermedios (>3,5 a <14 pg), grandes (>14 pg) y muy grandes (>35 pg). El tamaño del genoma ha sido estimado en aproximadamente 8510 especies (Bennett & Leitch, 2012), entre las cuales sólo se conoce para Spergula el valor C de S. arvensis correspondiente a 1C = 1,05 pg (Bennett et al., 1998).
Respecto al conocimiento citogenético en Spergula, este es muy escaso, solo registrándose los siguientes números cromosómicos: Spergula arvensis L. 2n= 18 (Arohonka, 1982; Sidhu & Bir, 1983; Runemark, 1996); S. bocconii 2n= 36 (Dalgaard, 1985; Runemark, 1996); S. diandra: n= 9 (Humphries et al, 1978; Kaur & Singhal, 2012 ), n= 18 (Kaur & Singhal, 2012); y S. salina: 2n= 18 (Probatova, 2006), 2n= 36 (Petrova, 1995; Lifante et al, 1992).
En relación al estudio palinológico en Spergula, los granos de polen de S. arvensis han sido los más estudiados. Se destacan, los trabajos realizados por Sáenz (1980) quien analiza el polen de esta especie de tres provincias de España, y lo caracteriza como suboblatos, exarrugados, con aperturas dispuestas globalmente, y de exina muy delgada, con gránulos esferoidales y diminutos poros homogéneos. Taia (1994) describe para S. arvensis granos subprolatos, 4-6-8 colpados, con presencia de membrana y téctum granulado. Erdtman (1966) referencia para la especie granos 6 colpados, mientras que Perveen & Qaiser (2006) describe la especie sobre material de Pakistán, con granos 3-4-10 colpados. Para Argentina, Volponi (2013) analiza 5 especies de Spergula: S. arvensis, S. laevis (Cambess.) D. Dietr., S. platensis (Cambess.) Shinners var. balansae (R. Rossbach) Pedersen, S. platensis (Cambess.) Shinners var. platensis Phil., S. ramosa (Cambess.) D. Dietr. ssp. ramosa y S. villosa Pers., las cuales agrupa bajo el Tipo Spergula ramosa. Este tipo polínico incluye granos 3-colpados (algunos granos en Spergula arvensis son 4- colpados); predominantemente pequeños; prolato- esferoidales a oblato-esferoidales, de ámbito subcircular; colpos largos, de 1,8 - 4 µm de ancho; exina de 0,5 - 1,3 µm de espesor, adelgazada hacia las aperturas, algo más gruesa en los polos, granos tectados microequinados, imperforados o puncteados.
Recientes estudios han encontrado una fuerte relación positiva entre el tamaño del genoma y el tamaño de las células, dejando abierta la posibilidad de que el tamaño del genoma puede determinar en parte, o correlacionarse, con el tamaño de polen (Beaulieu et al., 2008; Knight et al., 2010). En concordancia, existen estudios que sugieren que el tamaño del polen aumenta con la ploidía (Gould, 1957; Johansen & von Bothmer, 1994; Knigth et al., 2010).
La falta de conocimiento sobre el tamaño del genoma, los escasos datos cromosómicos y su relación con el tamaño del polen en Spergula nos llevó a plantear los siguientes objetivos: (1) estimar el tamaño del genoma; (2) estudiar la relación entre el tamaño del genoma y las características cariotípicas; y (3) analizar la relación entre la variación del tamaño del genoma y el tamaño del polen.
maTeriales y méTodos
Material Biológico
El material analizado en la estimación del contenido de ADN, los recuentos cromosómicos y la estimación tamaño del polen se presenta en la Tabla 1. Los ejemplares correspondientes fueron depositados en el herbario del Instituto Botánica del Nordeste (CTES). Las muestras palinológicas se obtuvieron de ejemplares de los herbarios BAB, CTES y SI.
Estimación del tamaño del genoma
El contenido de ADN (2C) fue estimado usando un citómetro PA fow (Partec GmbH, Germany) en núcleos aislados de tejido foliar. Las muestras fueron analizadas a partir de material fresco o de material conservado en sílica-gel a -10ºC. Una hoja completa fue molida finamente con hoja de afeitar en 0,5 mL de buffer de extracción (CyStain® UV Precise P), y se fltró a través de una malla de nylon de 40 µm para separar la suspensión de núcleos del material grueso. Posteriormente, éstos fueron teñidos con una solución de ioduro de propidio, luego de una hora de incubación las muestras fueron examinadas.
Se llevaron a cabo tres estimaciones de ADN para cada muestra (5.000 núcleos por análisis), siendo representada ésta por un individuo para cada taxón (Tabla 1), y se utilizó como estándar interno a Paspalum dilatatum Chirú (2n= 6x= 60= 3,57 pg) (Vaio et al., 2007). El contenido de ADN nuclear (2C) desconocido se calculó mediante la fórmula: 2C contenido de ADN de la muestra= (Media del pico de la muestra/Media del pico del estándar) × 2C contenido de ADN del estándar. Las medias y los desvíos estándar fueron estimados en cada medida del contenido de ADN de la muestra.
Los valores Cx, que representan el contenido de ADN de un genoma monoploide no replicado con el número cromosómico x (Greilhuber et al., 2005), se calcularon dividiendo la cantidad de ADN nuclear 2C por el nivel de ploidía.
Características cariotípicas
Números cromosómicos. - Los recuentos cromosómicos fueron realizados a partir de meristemas radicales de semillas germinadas en cajas de Petri. Los ápices radicales fueron pretratados con 8-hidroxiquinoleína 0,002 M durante cuatro horas a temperatura ambiente, fijados en una mezcla de 5:1 (etanol: ácido láctico) por 24 horas (Fernández, 1973) y luego teñidos mediante la técnica de Feulgen (Jong, 1997). Posteriormente los meristemas fueron aplastados en una gota de orceína lacto-propiónica; los preparados se hicieron permanentes con Euparal (Asco Laboratories, Manchester, UK). Las microfotografías fueron tomadas a través de un microscopio Zeiss Axioplan con una cámara Canon Powershot A640 de las cinco mejores metafases por individuo (Tabla 1) y se realizaron los conteos cromosómicos.
Tamaño cromosómico. -Debido al reducido tamaño de los cromosomas resultó imposible la confección de idiogramas, aunque se pudo obtener los parámetros: longitud cromosómica total del genoma diploide (LCT) y longitud cromosómica promedio (LCP). Para ello, fueron medidos los cromosomas de cinco metafases completas con un estado de condensación similar utilizando el software ImageJ (Rasband, 1997-2016).
Tamaño del polen
Se estudió el polen de 3 ejemplares de cada especie, los granos de polen se prepararon utilizando la técnica de acetólisis (Erdtman, 1960) y se montaron en gelatina glicerinada. Posteriormente los preparados palinológicos permanentes se depositaron en la Palinoteca de la Universidad Nacional del Nordeste (PAL- CTES). Las mediciones se llevaron a cabo sobre un total de 20 granos por muestra de flores en antesis con microscopio óptico Leica DM LB2. Además, el estudio se complementó con microscopio electrónico de barrido (MEB) en un equipo JEOL 5800 LV de la Secretaría General de Ciencia y Técnica de la Universidad Nacional del Nordeste operando a 15 k V. Para este análisis los granos de polen fueron montados sobre un disco de aluminio de 5 cm de diámetro, luego se metalizaron con un delgado baño de oro-paladio y se obtuvieron fotomicrografías.
Análisis estadístico
Para evaluar la relación entre el tamaño del genoma y las características cariotípicas se llevó a cabo un análisis de regresión lineal, utilizando el valor Cx como variable independiente y número cromosómico, valores de LCT y LCP como variables dependientes. Además, se realizó otro análisis similar para conocer la correspondencia entre el tamaño del genoma y el tamaño del polen (longitud del eje polar), siguiendo el criterio propuesto por Knight et al. (2010), considerando al grano de polen como "unicelular". El promedio de la longitud del eje polar fue utilizado en el análisis de regresión.
Los análisis estadísticos fueron llevados a cabo utilizando el programa InfoStat (Di Rienzo et al., 2011).
Resultados
Tamaño del genoma
El valor Cx y el valor 2C con los errores estándar de las especies estudiadas se detallan en la Tabla 1. El tamaño del genoma varía muy poco entre las especies estudiadas (Fig. 1), correspondiendo el valor más pequeño a Spergula salina 4x (Cx= 0,458 pg) mientras que el valor más alto corresponde a S. arvensis (Cx= 0,856 pg).
Números cromosómicos
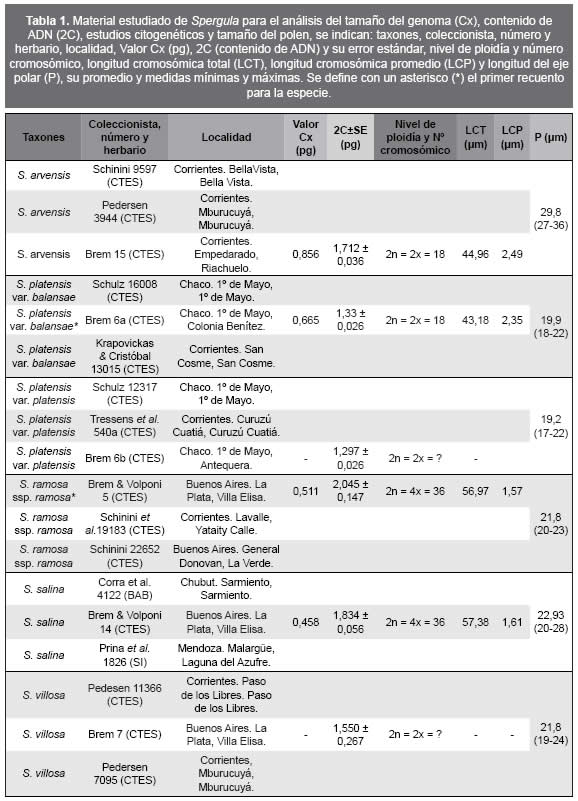
En la Tabla 1 se detallan los números cromosómicos obtenidos a partir del análisis mitótico. Entre los recuentos de las cuatro especies de Spergula, dos registran número cromosómico diploide de 2n= 2x= 18 (S. arvensis y S. platensis var. balansae) mientras que las otras dos presentan una dotación tetraploide de 2n= 4x= 36 (S. ramosa ssp. ramosa y S. salina), siendo el número básico x= 9, ilustrándose las correspondientes metafases mitóticas en la Fig. 2.
Tamaño cromosómico
La longitud cromosómica total del genoma diploide (LCT) varió de 57,38 µm (S. salina) a 43,18 µm (S. platensis var. balansae) mientras que la longitud cromosómica promedio (LCP) varió de 2,49 µm (S. arvensis) a 1,57 µm (S. ramosa spp. ramosa) Tabla 1.
Relación tamaño del genoma (Cx) y número cromosómico
El análisis de regresión lineal entre el tamaño del genoma y el número cromosómico (Fig. 3) mostró una relación lineal negativa y significativa (R2= 0,79; p<0,05), el diagrama de dispersión muestra que las variables son inversamente proporcionales.

Fig. 1. Histogramas de ADN obtenidos con citometría de fujo: A: S. platensis var. balansae. B: S. platensis var. platensis. C: S. ramosa ssp. ramosa. D: S. salina. E: S. villosa. El eje "x" representa la fuorescencia de yoduro de propidio (PI) (es decir, la cantidad relativa de ADN) y el eje "y" representa el número de núcleos medidos (× 100). M, muestra medida en cada histograma; Es, estándar interno.
Casi el 80% de la variabilidad del número de cromosomas se explica a través del tamaño del genoma, a mayor número cromosómico menor tamaño del genoma y viceversa.
Relación tamaño del genoma (Cx) y longitud cromosómica total del genoma diploide (LCT)
El análisis de regresión lineal entre el tamaño del genoma y la longitud cromosómica total del genoma diploide (Fig. 4) mostró una relación lineal positiva significativa (R2= 0,13; p<0,050) pero muy baja, la pendiente muestra que las variables guardan poca relación entre sí.
Relación tamaño del genoma y longitud cromosómica promedio (LCP)
El análisis de regresión lineal entre el tamaño del genoma y la longitud cromosómica promedio (Fig. 5) mostró una relación lineal positiva y significativa (R2= 0,84; p<0,05), el diagrama de dispersión muestra que las variables son directamente proporcionales. Los Resultados indican que la longitud cromosómica es dependiente del tamaño del genoma en las especies estudiadas. Las especies diploides (S. arvensis y S. platensis var. balansae) poseen cromosomas de mayor longitud y mayor contenido de ADN respecto de las especies poliploides (S. ramosa ssp. ramosa y S. salina) (Tabla 1).
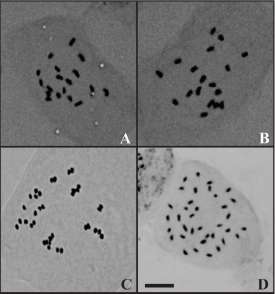
Fig. 2. Fotomicrografías de metafases mitóticas. A: Spergula arvensis. B: S. platensis var. balansae. C. S. ramosa ssp. ramosa. D: S. salina. Escala: 10µm.

Fig. 3. Análisis de regresión lineal mostrando una relación lineal negativa y significativa entre el tamaño del genoma (Valor Cx) y el número cromosómico en cuatro taxones de Spergula.
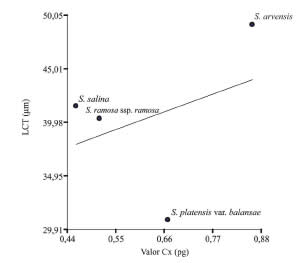
Fig. 4. Análisis de regresión lineal mostrando una relación lineal muy baja positiva y significativa entre el tamaño del genoma (Valor Cx) y el longitud cromosómica total del genoma diploide (LCT) en cuatro taxones de Spergula.

Fig. 5. Análisis de regresión lineal mostrando una relación lineal positiva y significativa entre el tamaño del genoma (Valor Cx) y la longitud cromosómica promedio (LCP) en cuatro taxones de Spergula.
Relación tamaño del genoma y tamaño del polen El tamaño del polen varía entre 17 a 36 micras (Tabla 1, Fig. 6), los granos medianos corresponden a S. arvensis, granos medianos a pequeños en S. salina mientras que las restantes especies poseen granos pequeños.
El análisis de regresión lineal entre el tamaño del genoma y el tamaño del polen (Fig. 7) mostró una relación lineal baja positiva, no significativa (R2= 0,48; p>0,05).
Discusión
En las Angiospermas, la variación del contenido nuclear de ADN ocurre en todos los niveles taxonómicos incluso en especies muy relacionadas entre sí (Price, 1988). Sobre la base de la información disponible acerca del tamaño del genoma, la familia Caryophyllaceae se caracteriza por presentar genomas muy pequeños a intermedios cuyo valor Cx oscila entre 0,42 -4,33 pg (Bennett & Leitch, 2012).
Respecto a Spergula, existe un solo registro del tamaño del genoma y corresponde a S. arvensis, 1C= 1,05 pg (Bennett et al., 1998), el cual es muy similar al aquí obtenido. La totalidad de los genomas analizados en este trabajo presentan una tendencia muy conservadora a mantener su tamaño muy pequeño (i.e., Cx <1,4 pg), tendencia que se condice con aquellas reconstruidas por Leitch et al. (1998) y Soltis et al. (2003) en las angiospermas ancestrales. Sin embargo, Coulleri et al. (2014) han enfatizado que la evolución del genoma más que ser conservadora presenta una naturaleza dinámica, en la cual algunos linajes que poseen grandes tamaños de genoma se caracterizan por tasas más lentas de diversificación y mayores tasas de extinción que los linajes con tamaño de genoma medio a muy pequeño. Por lo tanto, y de acuerdo Leitch et al. (1998) y Soltis et al. (2003), la selección natural podría favorecer la reducción del tamaño del genoma, hecho que se evidencia en que la mayoría de las angiospermas tienen

Fig. 6. Microfotografías obtenidas con microscopio electrónico de barrido (MEB) y microscopio óptico (MO). MEB: A: Vista ecuatorial de S. arvensis. B: Vista ecuatorial y C: Vista subpolar de S. platensis var. balansae. F: Vista ecuatorial de S. platensis var. platensis. G: Vista polar de S. ramosa ssp. ramosa. J: Vista subpolar de S. salina. K y N: Vista ecuatorial de S. villosa. MO: D: Vista ecuatorial y E: Vista subecuatorial de S. arvensis. H: Vista subecuatorial e I: Vista ecuatorial de S. platensis var. balansae. L: Vista ecuatorial y M: Vista polar de S. platensis var. platensis. O: Vista ecuatorial de S. ramosa ssp. ramosa. P: Vista polar de S. salina. Q: Vista ecuatorial de S. villosa: Escala= 12µm.
genomas pequeños o muy pequeños al igual que los presentados por las especies objeto de este estudio.
En poliploides, se espera que el valor 2C sea mayor al de sus progenitores diploides de manera proporcional a su nivel de ploidía, sin embargo el valor Cx se espera que sea similar (Leitch & Bennett, 2004). Esta expectativa se observa en algunas series poliploides, especialmente las recién formadas (Urdampilleta et al., 2016), pero hay ejemplos que sugieren que los valores Cx en poliploides particulares son menores que lo esperado (Leitch & Bennett, 2004; Zenil-Ferguson et al., 2016; Simonin & Roddy, 2018), dada la tendencia a la reducción del tamaño del genoma favorecida por la selección natural.
Evolución cromosómica
El aporte realizado en relación a los recuentos cromosómicos más lo conocido para el género constituye casi el 43% del total de las especies de Spergula que habitan la Argentina. La variabilidad en el numero cromosómico registrado a partir de nuestros Resultados detallan la existencia de algunos poliploides, característica frecuente en especies de Paronychioideae (Bittrich, 1993) que explica en parte la evolución del número cromosómico en el género Spergula. En la familia se registran cambios numéricos en relación a la evolución cariotípica, es decir variaciones en el número de cromosomas, ya sea por la presencia o ausencia de cromosomas (aneuploidía) tal como fue demostrado en Silene por Melzheimer (1974), o por aumento en el juego completo de cromosomas (poliploidía). Numerosos autores sugieren que los poliploides muestran un mejor rendimiento y más bajas tasas de extinción en ambientes nuevos, extremos o cambiantes (Hagerup, 1932; Stebbins, 1970; Brochmann et al., 2004; Wood et al., 2009; Soltis et al. 2014). De hecho, S. salina y S. ramosa ssp. ramosa, son ejemplos de poliploides eficientes con distribución amplia, capaces de conquistar ambientes muy variables (Brem, 2017).
En cuanto a los cambios estructurales, si bien no pudieron ser determinadas las características morfológicas de los cromosomas, nuestros Resultados evidencian una ligera variación en la longitud cromosómica total (LCT). Autores como Flavell (1986) y Uozu et al. (1997) proponen que la presencia de variaciones en secuencias repetitivas dispersas o dispuestas en bloques se evidencia en los cambios en la LCT y en el tamaño del genoma. Esto pareciera indicar una compensación entre el número cromosómico vs. el tamaño del cromosoma y del genoma, ya que las especies tetraploides analizadas (S. ramosa ssp. ramosa y S. salina) muestran cromosomas y tamaño del genoma más pequeño comparativamente con las especies diploides (S. arvensis y S. platensis var. balansae). Dicha tendencia ésta en concordancia con la reducción del tamaño del genoma en poliploides señalado por Leitch & Bennett (2004), Zenil-Ferguson et al. (2016) y Simonin & Roddy (2018); los primeros autores sugieren que la selección natural opera en favor de: aquellos individuos eficientes en mecanismos de eliminación del ADN en relación a la idea del "nucleotipo" (Bennett, 1987); la reducción de los costos bioquímicos inherentes a la síntesis, replicación y transcripción de ADN asociados al ADN extra; y a la mejora en el apareamiento entre homólogos y posterior estabilización del poliploide (Feldman et al., 1997), dando lugar a la reducción del genoma a gran escala en poliploides.
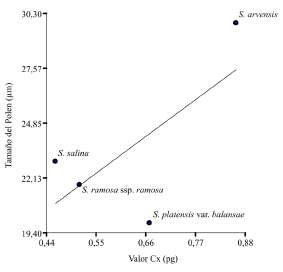
Fig. 7. Análisis de regresión lineal mostrando una relación lineal baja positiva y no significativa entre el tamaño del genoma (Valor Cx) y el tamaño del polen (P) en cuatro taxones de Spergula.
Relación entre el tamaño del polen y el tamaño del genoma
El tamaño del polen de las especies analizadas son coincidentes, en su mayoría, con lo sugerido por Erdtman (1966) para la tribu Sperguleae (Paronychioideae) y por Punt & Hoen (1995), Perveen & Qaiser (2006) y Volponi (2013) para el género.
Dada la fuerte tendencia positiva entre el tamaño celular y el tamaño del genoma reportado por Beaulieu et al. (2008), especulamos que el tamaño del genoma podría estar correlacionado en parte con el tamaño del polen. En Caryophyllaceae, un grano de polen consiste en una célula vegetativa y dos células generativas; o sea el polen es tricelular (Saito et al., 2002). Para este análisis, como se citó anteriormente seguimos a Knight et al. (2010), quienes se refrieron al polen como "unicelular". Nuestros resultados mostraron una relación débil entre el tamaño del genoma y el tamaño del polen (Fig. 6), esto podría deberse a que el polen no es metabólicamente activo después de la dehiscencia o liberación, sino más bien hasta su momento de imbibición y germinación, cuando alcanza el volumen máximo en la etapa de la formación del tubo polínico, tal como lo advirtió Knight et al. (2010) en varias familias de plantas con semillas. Entonces, es posible que esta correlación débil surgiera a partir del análisis en una etapa incorrecta del desarrollo del polen. Por otra parte, estudios similares realizados en Sapindaceae (Coulleri et al., 2014) explican la débil relación entre ambos parámetros a través de la selección natural actuando a favor de granos pequeños en relación con estrategias de dispersión, teniendo un grano pequeño mayor probabilidad de transporte a un estigma receptivo tanto por el viento como por insectos, que granos grandes. Ésta fuerte selección natural puede oscurecer cualquier relación entre el tamaño del polen y el tamaño del genoma tal como lo propusieron Leitch et al. (2005).
El mecanismo de dispersión del polen en Spergula es principalmente entomófla mediada por sírfdos, que toman el néctar de las fores adoptando el aspecto de himenópteros (abejas y avispas) con las que fácilmente se confunden (New, 1961; Bittrich, 1993; Brem, 2017). Así entonces, teniendo en cuenta lo planteado por Leitch et al. (2005), la selección natural en Spergula podría estar favoreciendo en gran medida un pequeño tamaño del polen en relación a la estrategia de dispersión.
Conclusiones
En este trabajo se reportan por primera vez dos recuentos cromosómicos (S. platensis var. balansae y S. ramosa ssp. ramosa), uno de los cuales resultó ser un poliploide. Además, se confrmó el número cromosómico en S. arvensis y se determinó que el citotipo tetraploide de S. salina habita en la República Argentina.
Por otro lado, se obtuvieron para Spergula las primeras estimaciones del contenido de ADN (2C) de cinco taxones y el tamaño del genoma (Cx) de cuatro, los cuales constituyen una novedad para la familia.
Se demostró la relación inversamente proporcional entre el número cromosómico y el tamaño del genoma, lo cual indicaría una tendencia evolutiva a la reducción del tamaño del genoma en las especies poliploides.
El análisis de la relación entre el tamaño del genoma y el tamaño del polen demostró que estas características varían de manera independiente entre sí en el género, probablemente debido a presiones de selección en contra de granos de polen grandes como estrategia de dispersión.
Agradecimientos
Los autores desean agradecer a la Dra. Myriam Carolina Peichoto, al Dr. Pablo Ortega-Baes y al Dr. Gabriel Bernardello, por la lectura crítica y las sugerencias realizadas sobre el manuscrito preliminar. Este trabajo ha sido subsidiado por la Agencia Nacional de Promoción Científica y Tecnológica (PICTO-UNNE, 2011-0202) y por el Consejo Nacional de Investigaciones Científicas y Técnicas (CONICET) y la Universidad Nacional del Nordeste (SGCyT PI15-P001).
Bibliografía
AROHONKA, T. 1982. Chromosome counts of vascular plants of the island Seili in Nauvo, southwestern Finland. Turun Yliopiston Biologian-Laitoksen Julkaisuja 3: 1-12. [ Links ]
BEAULIEU, J. M., I. J. LEITCH, S. PATEL, A. PENDHARKAR & C. A. KNIGHT. 2008. Genome size is a strong predictor of cell size and stomatal density in angiosperms. New Phytol. 179: 975-986. [ Links ]
BENNETT, M. D. 1987. Variation in genomic form in plants and its ecological implications. New Phytol. 106: 177-200. [ Links ]
BENNETT, M. D., I. J. LEITCH & L. HANSON. 1998. DNA amounts in two samples of angiosperm weeds. Ann. Bot. 82: 121-134. [ Links ]
BENNETT, M. D. & I. J. LEITCH. 2012. Plant DNA C-values database (release 6.0, Dec. 2012) http:// www.kew.org/cvalues/ [ Links ]
BITTRICH, V. 1993. Caryophyllaceae. En Kubitzki, K., Bittrich, V. , Rohmer, J. (eds.), The Families and Genera of Vascular Plants 2: 212-221. [ Links ]
BREM, M. C. 2017. Estudios biodistemáticos en las especies argentinas del género Spergula L. (Caryophyllaceae). Tesis doctoral. Universidad Nacional del Nordeste, Corrientes.
BROCHMANN, C., A. K. BRYSTING, I. G. ALSOS, L. BORGEN, H. H. GRUNDT, A. C. SCHEEN & R. ELVEN. 2004. Polyploidy in arctic plants. Biol. J. Linnean Soc. 82: 521-536.
COULLERI, J. P., J. D. URDAMPILLETA & M. S. FERRUCCI. 2014. Genome size evolution in Sapindaceae at subfamily level: a case study of independence in relation to karyological and palynological traits. Bot. J. Linn. Soc. 174: 589-600.
DALGAARD, V. 1985. Chromosome studies in fowering plants from Madeira. Willdenowia 137-156.
DI RIENZO, J. A., F. CASANOVES, M. G. BALZARINI, L. GONZÁLEZ, M. TABLADA & C. W. ROBLEDO. 2011. InfoStat, versión 2011. Grupo InfoStat, ficA, Universidad Nacional de Córdoba, Argentina. URL http://www.infostat.com.ar
ERDTMAN, G. 1960. The acetolysis method. Svensk Bot. Tidskr. 54: 561-564.
ERDTMAN, G. 1966. Pollen morphology and plant taxonomy. Angiosperms. Hafiner Publ Co, New York & London.
FELDMAN, M., B. LIU, G. SEGAL, S. ABBO, A. A. LEVY & J. M. VEGA. 1997. Rapid elimination of low-copy DNA sequences in polyploid wheat: a possible mechanism for differentiation of homoeologous chromosomes. Genetics 147: 1381-1387.
FERNANDES, A. & M. T. LEITÃO. 1971. Contribution a la connaissance cytotaxinomique des Spermatophyta du Portugal. III. Caryophyllaceae. Bol. Soc. Brot. 45: 167-176.
FERNÁNDEZ, A. 1973. El ácido láctico como fijador cromosómico. Bol. Soc. Argent. Bot. 15: 287-290.
FLAVELL, R. B. 1986. Repetitive DNA and chromosome evolution in plants. Phil. Trans. R. Soc. Lond. B. 312: 227-242.
GONZÁLEZ, G. L. 2010. Sobre el género Spergula L. [incl. Spergularia (Pers.) Pers. ex J. Presl & C. Presl, nom. cons.] (Caryophyllaceae) y sus especies en la Península Ibérica e Islas Baleares. Lagascalia 30: 7-18.
GOULD, F. W. 1957. Pollen size as related to polyploidy and speciation in the Andropogon saccharoides - A. barbinodis complex. Brittonia 9: 71-75.
GREILHUBER, J., J. DOLEZEL, M. A. LYSAK & M. D. BENNETT. 2005. The origin, evolution and proposed stabilization of the terms 'genome size' and 'C-value' to describe nuclear DNA contents. Ann. Bot. 95: 255-260.
HAGERUP, O. 1932. Über polyploidie in beziehung zu klima, ökologie und phylogenie. Hereditas 16: 19-40.
HUMPHRIES, C. J., B. G. MURRAY, G. BOCQUET & K. N. VASUDEVAN. 1978. Chromosome numbers of phanerogams from Morocco and Algeria. Bot. Not. 131: 391-406.
JOHANSEN, B. & R. VON BOTHMER. 1994. Pollen size in Hordeum L.: correlation between size, ploidy level, and breeding system. Sex. Plant Reprod.7: 259-263.
JONG, J. 1997. Laboratory manual of plant cytological techniques. Royal Bot. Garden, Edinburgh.
KAUR, D. & V. K. SINGHAL. 2012. Phenomenon of cytomixis and intraspecific polyploidy (2x, 4x) in Spergularia diandra (Guss.) Heldr. & Sart. in the Cold Desert Regions of Kinnaur District (Himachal Pradesh). Cytologia 77: 163-171.
KENYON, J. C. 1997. Models of reticulate evolution in the coral genus Acropora based on chromosome numbers: parallels with plants. Evolution 51: 756-767.
KNIGHT, C. A., R. B. CLANCY, L. GÖTZENBERGER, L. DANN & J. M. BEAULIEU. 2010. On the relationship between pollen size and genome size. J. Bot. 2010: 1-7.
LEITCH, I. J. & M. D. BENNETT. 2004. Genome downsizing in polyploid plants. Biol. J. Linn. Soc. 82: 651-663.
LEITCH, I. J., M. W. CHASE & M. D. BENNETT. 1998. Phylogenetic analysis of DNA C-values provides evidence for a small ancestral genome size in fowering plants. Ann. Bot. 82: 85-94.
LEITCH, I. J., D. E. SOLTIS, P. S. SOLTIS & M. D. BENNET. 2005. Evolution of DNA amount across land plants (Embryophyta). Ann. Bot. 95: 207-217.
LIFANTE, Z. D., T. LUQUE & C. S. BÁRBARA. 1992. Chromosome numbers of plants colIected during Iter Mediterraneum II in Israel. Bocconea 3: 229-250.
MELZHEIMER, V. 1974. Bemerkungen zur cytologie einiger arten der gattung Silene L. von der Balkan- Halbinsel. Candollea 29: 337-343.
NEW, J. K. 1961. Spergula arvensis L. Journal of Ecology 49: 205-215.
PEDERSEN, T. M. 1984. Caryophyllaceae. En Correa, M. N. (ed.), Flora Patagónica, Colec. Cient. I.N.T.A. 8: 253-267.
PERVEEN A. & M. QAISER. 2006. Pollen fora of Pakistan-LI-Caryophyllaceae. Pak. J. Bot. 38: 901-915.
PETROVA, A. 1995. Mediterranean chromosome number reports 5 (415-434). Fl. Medit. 5: 279-288.
PRICE, H. J. 1988. Nuclear DNA content variation within angiosperm species. Evol. Trend. Plant. 2: 53-60.
PROBATOVA, N. S. 2006. Chromosome numbers of plants of the Primorsky Territory, the Amur River basin and Magadan region. Bot. Zhurn. (Moscow & Leningrad) 91: 491-509.
PUNT W. & P.P. HOEN. 1995. Caryophyllaceae. Rev. Palaeobot. Palynol. 88: 83-272.
RASBAND, W. S. 1997-2016. ImageJ, U. S. National Institutes of Health, Bethesda, Maryland, USA, https://imagej.nih.gov/ij/
RATTER, J. A. 1976. Cytogenetic studies in Spergularia: IX. Summary and conclusions. Notes Roy. Bot. Gard. Edinburgh 34: 411-428.
RUNEMARK, H. 1996. Mediterranean chromosome number reports 6 (590-678). Fl. Medit. 6: 223-243.
SÁENZ, C. 1980. Polen de la fora de Doñana (Huelva, España). Lazaroa 2: 191-270.
SAITO, C., N. NAGATA, A. SAKAI, K. MORI, H. KUROIWA & T. KUROIWA. 2002. Angiosperm species that produce sperm cell pairs or generative cells with polarized distribution of DNA-containing organelles. Sex. Plant Reprod. 15: 167-178.
SIDHU, M. & S. S. BIR. 1983. Karyological studies on weeds on cultivable lands in Punjab, India. Trop. Life Sci. Res. 1: 1-13.
SIMONIN, K. A. & A. B. RODDY. 2018. Genome downsizing, physiological novelty, and the global dominance of flowering plants. PLoS Biol. 16: e2003706.
SOLTIS, D. E., P. S. SOLTIS, M. D. BENNETT & I. J. LEITCH. 2003. Evolution of genome size in the angiosperms. Am. J. Bot. 90: 1596-1603.
SOLTIS, D. E., C. J. VISGER & P. S. SOLTIS. 2014. The polyploidy revolution then. and now: Stebbins Revisited. Am. J. Bot. 101: 1057-1078.
STEBBINS, G. L. 1970. 1970. Variation and evolution in plants: progress during the past twenty years. In: Hecht M. K. & W. C. Steere (Eds.), Essays in Evolution and Genetics in Honor of Theodosius Dobzhansky. A Supplement to Evolutionary Biology, pp. 173-208. Appleton-Century-Crofts, N.Y.
TAIA, W. K. 1994. On the pollen morphology of some Egyptian Caryophyllaceae. J. King Saud Univ. 6: 149-165.
UOZU, S., H. IKEHASHI, N. OHMIDO, H. OHTSUBO, E. OHTSUBO & K. FUKUI. 1997. Repetitive sequences: cause for variation in genome size and chromosome morphology in the genus Oryza. Plant Mol. Biol. 35: 791-799.
URDAMPILLETA, J. D., J. P. COULLERI & M. S. FERRUCCI. 2016. Insights into the Andean genera Bridgesia and Guindilia (Sapindaceae): an integrated approach. Syst. Biodivers. 14: 583-598.
VAIO, M., C. MAZZELLA, V. PORRO, P. SPERANZA, B. LOPEZ-CARRO, E. ESTRAMIL & G. A. FOLLE. 2007. Nuclear DNA content in allopolyploid species and synthetic hybrids in the grass genus Paspalum. Plant Syst. Evol. 265: 109-121.
VOLPONI, C. R. 1994. Caryophyllaceae. En R. Kiesling. (ed.), Flora de San Juan 1: 175-178.
VOLPONI, C. R. 2013. Caryophyllaceae. En PIRE, S. M., L. M. ANZÓTEGUI & G. A. CUADRADO (eds.) Flora Polínica del Nordeste Argentino I V: 51-58 EUDENE, Corrientes, Argentina.
WOOD, T. E., N. TAKEBAYASHI, M. S. BARKER, I. MAYROSE, P. B. GREENSPOON & L. H. RIESEBERG. 2009. The frequency of polyploid speciation in vascular plants. Proc. Natl. Acad. Sci. U.S.A. 106: 13875-13879.
ZENIL-FERGUSON, R., J. M. PONCIANO & J. G. BURLEIGH. 2016. Evaluating the role of genome downsizing and size thresholds from genome size distributions in angiosperms. Am. J. Bot. 103: 1175-1186.
ZULOAGA, F. O., O. MORRONE & M. BELGRANO. 2008. Caryophyllaceae. En ZULOAGA, F. O., O. MORRONE & M. BELGRANO (eds.), Catálogo de las Plantas Vasculares del Cono Sur Vol. 2. Monogr. Syst. Bot. Missouri Bot. Gard. 107: 1894-1896.
Recibido el 1 de julio de 2017, aceptado el 6 de abril de 2018. Editora: Viviana Solís Neffa.














