Introducción
Solanaceae Juss. cuenta con alrededor de 98 géneros y 2700 especies (Olmstead & Bohs, 2007; Olmstead et al., 2008; Sarkinen et al., 2013). La familia es cosmopolita y Sudamérica es su centro de diversificación (Hunziker, 2001, Olmstead & Bohs, 2007; Olmstead et al., 2008). Incluye representantes de gran valor económico, etnobotánico, científico y ornamental, además de otras que revisten interés como especies venenosas y de uso medicinal (Hunziker, 2001; Bohs & Olmstead, 2001). Solanum L. es el mayor y más diverso género de la familia (inclusive, uno de los más ricos dentro de Angiospermas), con aproximadamente, 1100-1400 especies válidas (Hunziker, 2001; Bohs, 2005; Sarkinen et al., 2013). Cerca de un tercio de estas especies pertenecen al subgénero Leptostemonum (Dunal) Bitter, conocidas como ”Solanum espinosos". La monofilia del subgénero ha sido comprobada por varios estudios de filogenia molecular (Olmstead & Palmer, 1997; Levin et al., 2006; Sarkinen et al., 2013). El grupo incluye plantas comestibles, así como también malezas (Levin et al., 2006; Knapp et al., 2017).
Solanum elaeagnifolium Cav. es una de estas malezas que merece especial atención. Es una planta invasora en distintas partes del mundo (Knapp et al., 2004; Stanton et al., 2009, 2012; Zhu et al., 2013), sobre todo en áreas secas (Knapp et al., 2017) y muestra un alto grado de variación morfológica a lo largo de su área de distribución, particularmente en la forma de la hoja y en el número de acúleos (Boyd et al., 1984; Knapp et al., 2004; Stanton et al., 2009, 2012; Zhu et al., 2013; Knapp et al., 2017). Se trata de una hierba perenne cubierta por numerosos tricomas peltados de aspecto blanquecino/plateado, con un tallo que puede crecer hasta 1 m de altura y hojas simples con márgenes enteras o lobuladas (Christodoulakis et al., 2009; Burrows et al., 2013; Knapp et al., 2017).
Los tricomas de S. elaeagnifolium son muy peculiares y son de dos tipos: glandulares con cabeza uni-pluricelular esférica y un pie bicelular; y eglandulares estrellados (Bruno et al., 1999). Los tricomas estrellados son menos numerosos en el hipofilo que en el epifilo. Tienen una estructura compleja y varían en tamaño (diámetro y altura) y en el número de células. Todos poseen un pie verticalmente ascendente que mide aproximadamente entre 20 y 60 pm de longitud y una célula central con pared celular secundaria gruesa y lignificada, rodeada de 10-18 células no lignificadas (rayos), que presentan un arreglo radial, formando una roseta alrededor de la célula central y están unidos por sus bases (Cosa et al., 1998; Christodoulakis et al., 2009; Burrows et al., 2013). Estos rayos miden entre 120 y 300 pm de longitud; el pie es biseriado y tiene 2 o 4 células de paredes gruesas (Cosa et al., 1998). En cuanto a los estomas foliares, son de tipo anomocítico, anisocítico y paracítico (Cosa et al., 1998) y se encuentran en ambas superficies de la hoja (Prabhakar 2004; Christodoulakis et al., 2009), aunque más abundantes en el hipofilo (Cosa et al., 1998).
Las flores de S. elaeagnifolium son pentámeras (ocasionalmente flores tetrámeras) con corolas estrelladas, lóbulos deltoides a triangulares y tejido interpetalar abundante. Las anteras son de forma cónica alargada, dehiscentes por un poro apical, suelen ser heteromórficas, con tres de las cinco ligeramente más largas que el resto, y en general son ligeramente curvadas. Al igual que en la mayoría de las especies del género, el androceo de S. elaeagnifolium se caracteriza por la formación de un cono anteral, una estructura adaptada a la “buzz pollination”, es decir, la polinización por abejorros que provocan la salida del polen por la vibración de sus músculos del vuelo (Coleman & Coleman, 1982; Knapp et al., 2017). El estilo es largo, ligeramente curvado, pubescente en la parte basal con tricomas estrellados (Knapp et al., 2017).
Varios rasgos contribuyen con el potencial de invasión de S. elaeagnifolium: es capaz de dispersarse a grandes distancias, es resistente a la sequía, puede resultar poco palatable y tóxica a los herbívoros (Christodoulakis et al., 2009) y produce compuestos alelopáticos (Mkula, 2006). Se propaga por medio de semillas, pero también asexualmente mediante raíces gemíferas (Fernández & Brevedan, 1972; Stanton et al., 2012). Debido a sus tallos subterráneos, las plantas forman densas colonias, a menudo en áreas perturbadas. Las raíces de S. elaeagnifolium han sido caracterizadas como “tuberizadas” (Cosa et al., 1998), presentando hasta 20 capas corticales. Solo las partes subterráneas verticales se engrosan, las horizontales no son tuberizadas ni engrosadas (Knapp et al., 2017). Estudios previos en S. elaeagnifolium demostraron la existencia de una serie euploide basada en x= 12 para poblaciones que crecen espontáneamente en Argentina (Moscone, 1992; Acosta et al., 2005; Scaldaferro et al., 2012; Chiarini, 2014; Chiarini et al., 2019). Tres citotipos fueron encontrados: diploide (2n=2x=24) de amplia distribución, tetraploide (2n=4x=48) de distribución oeste y sur de Argentina, y hexaploide (2n=6x=72) que predomina en el centro del país y se extiende hacia el Este (Scaldaferro et al., 2012). Varios indicios apuntan a que se trataría de autopoliploides y no de alopoliploides (Chiarini 2014; Knapp et al., 2017). Es necesario señalar que la poliploidía reviste importancia por muchos aspectos: la interacción entre genomas duplicados daría origen a cambios positivos en la expresión génica, alteraciones en las interacciones planta/ animal y dimorfismo sexual, entre otros aspectos (Wendel, 2000; Soltis et al., 2009). Los poliploides también suelen presentar diferente rango geográfico respecto de sus progenitores diploides, debido a que probablemente están pre-adaptados a hábitats cuyos recursos resultan limitados para sus progenitores (Lewis, 1980; Scaldaferro et al., 2012). Solanum elaeagnifolium es una especie altamente polimórfica y con diversidad cromosómica, no obstante, no existen estudios que consideren los rasgos morfoanatómicos en relación con el polimorfismo y el nivel de ploidía.
Solanum elaeagnifolium presenta también una marcada diversidad a nivel genético: un estudio filogeográfico realizado con secuencias del cloroplasto (Chiarini et al., 2018) detectó la presencia de tres linajes bien distanciados dentro de la especie. De estos linajes, solo dos están presentes en Argentina e incluyen poblaciones poliploides, con hexaploides en solo uno de ellos. La marcada diferencia entre las poblaciones de Argentina y las del resto del mundo fueron notadas también por Gopurenko et al. (2014), quienes se centraron en poblaciones de Australia, mientras que Zhu et al. (2012) advierten un alto grado de variación genética en poblaciones de S. elaeagnifolium en Australia.
Teniendo en cuenta todos estos antecedentes, se planteó el objetivo de estudiar variables morfológicas y anatómicas (de epidermis, flor y semilla) en especímenes de S. elaeagnifolium con distintos niveles de ploidía, con el fin de determinar si existen diferencias entre ellas que puedan estar relacionadas con los linajes genéticos hallados por Chiarini et al. (2019).
Específicamente, se propuso: 1) determinar el número cromosómico y el nivel de ploidía de poblaciones de Solanum elaeagnifolium, 2) describir y cuantificar los siguientes caracteres anatómicos de epidermis: morfología de los estomas; número de células epidérmicas propiamente dichas por mm2 (ppdd); número de estomas por mm2; tamaño de los estomas (pm2); número de rayos de los tricomas eglandulares; área de tricomas eglandulares (pm2), analizar masa de semillas y dimensiones de órganos florales (corola, estambres y pistilos), y relacionar las mediciones obtenidas en distintas poblaciones con los niveles de ploidía, los linajes genéticos y variables climáticas.
Materiales y Métodos
Material Vegetal. Para cada muestra (= población), la cual cuenta con un ejemplar de herbario depositado en el Museo Botánico de Córdoba (CORD), se intentó conocer el linaje genético (rojo, azul o amarillo, de acuerdo a Chiarini et al., 2019), el nivel de ploidía, la georreferencia (longitud y latitud) y tener suficientes réplicas de las mediciones de las semillas, las flores y variables de la epidermis en base al material que fue extraído de ellas, aunque esto no fue posible en la totalidad de los casos (Anexo 1). Una parte de las muestras contó con semillas colectadas en áreas naturales de Argentina y otros países, las cuales se hicieron germinar en cápsulas de Petri, obteniéndose así material para los estudios cromosómicos y para generar plántulas que fueron mantenidas en cultivo en los invernaderos del IMBIV, y que luego se usaron en los estudios de epidermis.
Preparados cromosómicos.
Se utilizaron raíces pretratadas con solución acuosa saturada de paradiclorobenceno durante 2 horas a temperatura ambiente. Posteriormente, se fijaron en una mezcla 3:1 de alcohol etílico absoluto: ácido acético glacial (preparada en el momento) durante 24 horas y se conservaron en freezer hasta su uso. Se efectuó la digestión enzimática de las raíces con Pectinex ® durante 30 minutos a 37°C. Los ápices radicales se aplastaron con ácido acético 45%, y se congelaron con nitrógeno líquido. Para la tinción se empleó una solución Giemsa al 2% preparada en el momento (Guerra, 1983). Posteriormente los preparados se montaron con Entellan®.
Los preparados fueron observados y fotografiados utilizando el microscopio Zeiss Axiophot con cámara digital Leica DFC300FX. El análisis de las imágenes se realizó con el software libre ImageJ (Rasband, 1997).
Anatomía de la epidermis
Las hojas fueron fijadas en FAA (formaldehído, alcohol, ác. acético). Se realizaron extendidos utilizando la técnica de "peeling" (D'Ambrogio de Argüeso, 1986), se tiñeron con azul astral por cinco minutos y safranina alcohólica por un minuto. Luego de enjuagar con agua, se realizó el montaje con glicerina acuosa al 50% y se procedió al sellado con pintura de uñas (Zarlavsky, 2014). Los estomas se clasificaron según Dilcher (1974) y Prabhakar (2004). La descripción de los tricomas se basó en el trabajo de Inamdar & Gangadhara (1977). Para el análisis cuantitativo, se determinó el número de células epidérmicas propiamente dichas y de estomas por mm2, el tamaño de los estomas en pm2 como así también el número de rayos de los tricomas estrellados y el área de los mismos. Para estos recuentos y mediciones se utilizó un ocular reticulado. El índice estomático se calculó según la siguiente fórmula (Stace, 1965):
I.E. = (frecuencia de estomas / frecuencia de estomas + frecuencia de células epidérmicas propiamente dichas) x 100
De cada muestra se tomaron tres individuos al azar que constituyeron las réplicas del diseño, y como pseudoréplicas, se tomaron tres hojas de cada individuo y cinco secciones de cada hoja.
Masa de semillas
De cada población se tomaron 10 semillas al azar, se pesaron en balanza de precisión y luego se promedió para obtener la masa de una semilla individual. Este procedimiento se replicó entre 3 y 10 veces para cada población.
Mediciones de las flores
Del material herborizado se extrajeron flores que fueron hidratadas, fotografiadas en la lupa Olympus SZX16 y posteriormente se midió la longitud de las cinco anteras, de los lóbulos corolinos y del pistilo, utilizando el software ImageJ. En el caso de las anteras, por ser estas desiguales entre sí en longitud, se calculó además un Coeficiente de Variación (CV) entre las cinco anteras de una misma flor.
Análisis estadístico
Las variables obtenidas fueron comparadas estadísticamente en entorno R 3.6.1 (R Core Team, 2019).
La diferencia multivariada entre niveles de ploidía para la longitud de la corola, longitud de pistilo, longitud promedio y coeficiente de variación (CV) de los estambres, número de rayos de los tricomas, área de los tricomas y masa de las semillas se exploró mediante un PERMANOVA, dada la heterogeneidad y falta de normalidad de los datos (Anderson et al., 2017). Para las variables número de células epidérmicas propiamente dichas por mm2, número de estomas por mm2 y tamaño de los estomas (pm2) no se obtuvieron datos suficientes como para poder comparar los niveles de ploidía. Debido a que el PERMANOVA no es tan robusto como otras técnicas multivariadas con respecto a la dispersión de los datos (Anderson et al., 2017), se puso a prueba la heterocedasticidad entre niveles de ploidía mediante la función betadisper del paquete vegan (Oksanen et al., 2019).
Para la representación visual de la disimilitud global en estos rasgos, se empleó un método de ordenamiento de escalado multidimensional no- métrico (NMDS, por sus siglas en inglés) (Minchin, 1987). Dado que el NMDS emplea rangos, es el método elegido para respuestas no-lineales (Oksanen, 2015). Se empleó la función metaMDS del paquete vegan en los valores de los rasgos transformados a logaritmo por las diferencias de escala entre las variables. Previamente, y debido a que no fue posible obtener datos de todos los rasgos para la totalidad de ejemplares para los que se contaba con el dato de nivel de ploidía, se optó por imputar los valores perdidos, lo cual se realizó con la función imputePCA del paquete miss MDA (Josse & Husson, 2016). Esta función realiza una serie de análisis de componentes principales de forma iterativa a la vez que imputa los valores perdidos mediante un valor inicial aleatorio. Este proceso se repite hasta alcanzar la solución con la menor diferencia entre la matriz ajustada y la observada (Josse & Husson, 2016). La matriz completa con los valores observados e imputados de los rasgos fue la empleada en el análisis de ordenamiento.
Se realizó un segundo análisis NMDS incluyendo esta vez los niveles de ploidía, para evaluar el poder explicativo de cuatro parámetros ambientales asociados a cada espécimen: temperatura promedio anual, precipitación promedio anual, coeficiente de variación de precipitaciones (estacionalidad pluvial) y desvío estándar de la temperatura (estacionalidad térmica) además de la longitud y latitud, dado que estas variables probaron ser explicativas en trabajos previos (Scaldaferro et al., 2012).
Los parámetros ambientales se obtuvieron de la base Worldclim versión 2 (http://www. worldclim.org/; Fick & Hijmans, 2017), previa georreferenciación utilizando la plataforma Google Earth 7.1.8.3036 (32-bit) de ejemplares de herbario de S. elaeagnifolium depositados en el Museo Botánico de Córdoba (CORD). Para la extracción de los valores se empleó el paquete raster (Hijmans, 2019) y el paquete sp (Pebesma & Bivand, 2005; Bivand et al., 2013) se utilizó para proyectar las coordenadas de cada espécimen.
Para el ajuste de los NMDS se utilizaron diagramas de Shepard, los cuales grafican el ajuste lineal o monotónico entre las distancias de ordenamiento y las distancias originales (Borcard et al., 2011).
Para el análisis del efecto del nivel de ploidía y el linaje sobre las variables estomáticas (número de células epidérmicas ppdd., número y área de estomas e índice estomático) se emplearon modelos lineales generalizados mixtos mediante el paquete lme4 (Bates et al., 2015) en entorno R 3.6.1 (R Core Team, 2019). Para el número de estomas y el número de células epidérmicas se empleó la función glmer y se utilizó distribución de Poisson, con ploidía (tres niveles: diploide, tetraploide, hexaploide) o linaje (tres niveles: azul, rojo y amarillo) como factores fijos y la hoja muestreada anidada dentro de individuo y dentro del ejemplar de herbario como factores aleatorios en ambos casos. En el caso del índice estomático se empleó la función lmer del mismo paquete, distribución normal, y la misma arquitectura de factores aleatorios, pero la estimación de la significancia se realizó con lmerTest (Kuznetsova et al., 2017).
Dado el gran desbalance de datos para las comparaciones en el número de semillas y el número de rayos del tricoma se optó por emplear un método no paramétrico gráfico, basado en bootstrap. Este método, llamado "gráficos de estimación de efecto" permite visualizar la magnitud de efecto y su grado de incertidumbre en el mismo gráfico (Ho et al., 2019) dentro del paquete dabestr para R.
Se exploraron las correlaciones entre las variables mediante un análisis no paramétrico utilizando la prueba de Spearman. En este análisis, los niveles de cada una de las variables a relacionar están codificados de forma ordinal, es decir, mediante rangos (el número de orden del valor de cada observación de la variable dentro del conjunto de observaciones) (Hauke & Kossowski, 2011).
Resultados
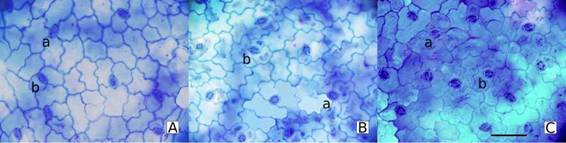
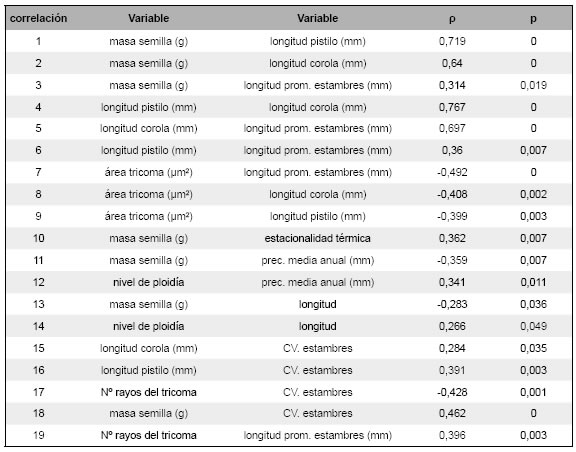
Fig. 1: Fotomicrografías de epidermis. A: Citotipo diploide. B: Citotipo tetraploide. C: Citotipo hexaploide. Todas las fotografías a la misma escala:50 pm. Abreviaturas=a: estomas anomocíticos, b: estomas anisocíticos.
Recuentos cromosómicos
Se obtuvieron preparados cromosómicos de 40 poblaciones. El número diploide (2n=2x=24) fue hallado en 19 poblaciones y el número tetraploide (2n=2x=48) en 12 poblaciones, mientras que en 9 poblaciones se encontró el número hexaploide (2n=2x=72) (Anexo 2). Estos datos se sumaron en los diferentes análisis a los datos de ploidía informados en trabajos previos (Scaldaferro et al., 2012; Chiarini et al., 2019). La distribución geográfica de las nuevas poblaciones analizadas coincide con el de las previas (Fig. 2 de Scaldaferro et al., 2012): el citotipo diploide ocupa áreas en el centro y oeste de Argentina, la distribución más occidental del citotipo tetraploide coincide con una diagonal árida (Bruniard, 1982), en la provincia biogeográfica de Monte (Cabrera & Willink, 1982), y el citotipo hexaploide se ubica en la región centro y este del país.
Variables anatómicas
Estomas. La morfología de los estomas de S. elaeagnifolium no se modifica con la ploidía (Fig. 1), presentándose en los tres niveles numerosos estomas anisocíticos, esto es, estomas completamente rodeados por sólo tres células subsidiarias, variables en posición y forma, con una de ellas claramente más pequeña, y estomas anomocíticos, es decir, sin células subsidiarias, rodeados por células edpidérmicas ppdd., variables en posición, forma y tamaño (Prabhakar, 2004). En algunos preparados se detectaron estomas paracíticos, estomas completamente rodeados por una o dos células subsidiarias cuyas paredes están orientadas hacia los polos de las células oclusivas, coincidiendo con los hallazgos previos (Cosa et al., 1998; Bruno et al., 1999; Christodoulakis et al., 2009).
Número de células epidérmicasppdd/mm 2 y número de estomas/mm 2 . El número de estomas por mm2 resultó ser diferente entre los distintos niveles de ploidía (Tabla 1, Fig. 2), siendo el citotipo tetraploide el que presentó menor número de estomas/mm2 (frecuentemente entre 7 y 8 por área de ocular reticulado) (Anexo 1). El número de células epidérmicas ppdd/mm2 y el índice estomático no presentaron diferencias significativas al compararse entre ploidías (Anexo 3).
Tamaño de los estomas (pm 2 ). El tamaño varió entre 103 y 478 pm2 (Anexo 1). Los valores medios se muestran en la Tabla 1. Esta variable no presentó diferencias significativas al compararse entre los distintos niveles de ploidía.
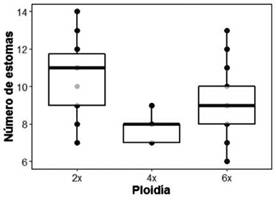
Fig. 2: Gráfico de cajas comparando el número de estomas entre los niveles de ploidía. Las medias fueron significativamente diferentes entre los tres niveles (z > |-2.23 |; P< 0.02).
Tricomas. Son pelos estrellados formados por un pie multicelular, más rayos laterales
unidos en su base (i.e. peltados, Fig. 3) y un rayo central reducido a un punto. En la mayoría de los casos, el número de rayos laterales es 16, pero se encontraron muestras con hasta 26 rayos, lo que hace que los hexaploides sean marginal pero significativamente distintos (Figs. 3-4). El área de estos tricomas varió entre las 61 pm2 y las 584 pm2 (Anexo 1), mientras que los valores medios para cada ploidía oscilaron entre 192-234 pm2 (Tabla 1). Aunque el área de los tricomas no mostró diferencias significativas entre niveles de ploidías, sí contribuyó a la discriminación de grupos en términos de similitud/ disimilitud a nivel global (ver PERMANOVA y Fig. 5).

Fig. 3: Fotomicrografías de tricomas estrellados. A: Citotipo diploide (Juliani 34). B: Citotipo tetraploide (Barboza 2319). C: Citotipo hexaploide (Moscone 180). D: Tricoma con la menor cantidad de rayos laterales (8) (Barboza 1944, diploide). E: Tricoma con la mayor cantidad de rayos laterales (26) (Moscone 109, hexaploide). F: Grupo de tres tricomas ilustrando la diferencia en tamaños. A- C presentan 16 rayos laterales. Escala= 200 pm.
Tabla 1 Medidas de resumen de las variables morfológicas estudiadas de acuerdo al nivel de ploidía: diploides (2x), tetraploides (4x) y hexaploides (6x). sd = desvío estándar. N = tamaño de la muestra. CV= coeficiente de variación.
Para ninguna de las variables epidérmicas (tanto de estomas como de tricomas) fue posible obtener datos suficientes como para comparar entre los tres linajes (rojo, amarillo y azul, Chiarini et al., 2019). Sí fue posible comparar los linajes amarillo y azul entre sí para la mayoría de las variables, pero las diferencias resultaron no significativas en todos los casos (Anexo 3).
Semillas. Las semillas de S. elaeagnifolium son de color tostado pálido, aplanadas, reniformes o lenticulares y se encuentran en un número promedio de alrededor de 60 dentro de cada fruto maduro, el cual es una baya de color amarillo (Chiarini & Barboza, 2007; Knapp et al., 2017). Los datos de masa presentaron una gran dispersión y elevada varianza (Tabla 1, Fig. 6), especialmente en el citotipo diploide. El test no paramétrico indicó que la media de la masa de las semillas de los tetraploides es marginalmente superior respecto de las otras dos ploidías.
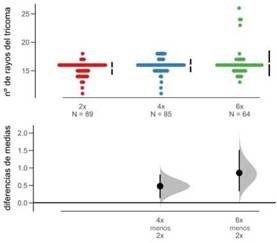
Fig. 4: Estimación de efectos para el valor medio de la diferencia en el número de rayos del tricoma entre ploidías de S. elaeagnifolium respecto a los diploides. En el gráfico superior se visualizan las observaciones y las líneas negras representan los valores medios. En el inferior se visualizan las curvas para la distribución de las diferencias resultado del remuestreo (N=100 bootstraps) y el intervalo de confianza. Los hexaploides fueron los únicos que difieren respecto a diploides y tetraploides (IC: 0.136; 1.52). Solo se muestra el gráfico respecto a los diploides.
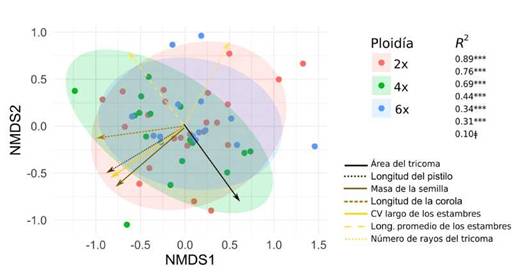
Fig. 5: Análisis de ordenamiento por rangos (NMDS) para longitud de la corola, longitud de pistilo, longitud promedio y coeficiente de variación (CV) de los estambres, número de rayos del tricoma y masa de la semilla, en relación a los tres niveles de ploidía de S. elaeagnifolium. El valor de R2 indica el ajuste entre el rasgo considerado y los ejes de ordenamiento. Valores significativos indican que ese rasgo contribuye a discriminar especímenes en los ejes. ** P=0.001; # P=0.08.
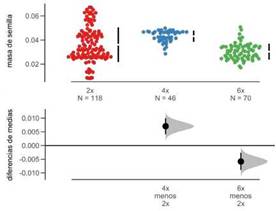
Fig. 6: Estimación de efectos para el valor medio de la diferencia en la masa de las semillas entre ploidías de S. elaeagnifolium respecto a los diploides. En el gráfico superior se visualizan las observaciones y las líneas negras representan los valores medios. En el gráfico inferior se visualizan las curvas para la distribución de las diferencias resultado del remuestreo (N=100 bootstraps) y el intervalo de confianza. En cada caso la diferencia entre tetraploides y diploides (IC: 0.00407; 0.00998) y entre hexaploides y diploides (IC:-0.0089;- 0.0027) resultó significativa. La masa de las semillas de los tetraploides supera la de los hexaploides (IC: -0.015; -0.011; gráfico no mostrado).
Flor. Los valores medios de las variables longitud de la corola, de los estambres y del pistilo (Tabla 1) no mostraron diferencias significativas entre los niveles de ploidía. Sin embargo, estas variables permitieron discriminar grupos de observaciones en términos de similitud / disimilitud a nivel global (ver PERMANOVA y Figs. 5, 7). Las cinco anteras de cada flor, que presentaron una longitud de entre 7.8 y 25.3 mm con una media de 16.1 mm (Anexo 1), en algunos individuos resultaron del mismo largo, mientras que en otros, dos de las anteras eran entre 0,1 y 18 % más cortas (5,5 % en promedio más cortas, ver Anexo 3, Tabla 1).
Con respecto a los linajes, no se registraron diferencias significativas en cuanto a la masa de semillas, mientras que para los rasgos florales solo hubo datos suficientes para comparar los linajes amarillo y azul, los cuales no resultaron ser distintos (Anexo 3), excepto para la variable longitud de la corola.
Análisis estadísticos
PERMANOVA. Este análisis permitió apreciar la variabilidad de los rasgos a nivel global y la utilidad de estos para separar grupos. En este sentido, la ploidía explicó el grado de similitud entre los ejemplares de manera significativa (R2=0,26; P=0,001). NMDS. En un primer análisis, se evaluó la interdistancia para cada observación con las variables número de rayos de tricomas, área de tricomas, masa de la semilla, longitud del pistilo, longitud de la corola, longitud promedio de los estambres y coeficiente de variación de la longitud de los estambres en su conjunto. Todas las variables mencionadas, excepto el número de rayos de los tricomas, contribuyeron a la discriminación entre niveles de ploidía. Es decir, estas variables son significativas para explicar las similitudes que tienen a nivel global las observaciones entre sí en los dos ejes resumen (NMDS1 y NMDS2) en relación a los tres niveles de ploidía (Figs. 5, 7). En el caso particular del citotipo tetraploide (mostrado en verde, Figs. 5, 7) la longitud promedio de los estambres y el área de los tricomas son las variables que mejor permiten diferenciar al grupo en términos de similitud / disimilitud global.
En un segundo análisis (NMDS de Rasgos morfológicos+variables ambientales y coordenadas geográficas) se encontró que la variable precipitación media anual es la única variable ambiental que contribuye a la discriminación entre los tres niveles de ploidía (Fig. 7).
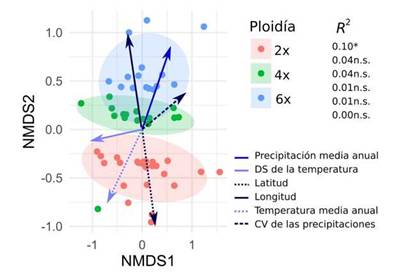
Fig. 7: Análisis de ordenamiento por rangos (NMDS) para el nivel de ploidía, el longitud de la corola, longitud de pistilo, longitud promedio y coeficiente de variación de los estambres, número de rayos del tricoma y masa de la semilla, y ajuste con parámetros ambientales asociados a cada espécimen estudiado de en S. elaeagnifolium. El valor de R 2 indica el ajuste entre el rasgo considerado y los ejes de ordenamiento. Valores significativos indican que efectivamente ese rasgo contribuye a discriminar especímenes en los ejes. CV: Coeficiente de variación, DS: Desvío estándar. * P=0.05; n.s.= no significativo. El valor de R2 para el coeficiente de variación de las precipitaciones es menor a 0,001.
Correlaciones. En la Tabla 2 se muestran las 11 variables que están significativamente correlacionadas de acuerdo a los resultados del análisis de correlaciones de Spearman (p<0.05). El resto de las variables no están correlacionadas y no se muestran en la tabla. Ninguna de las variables analizadas mostró diferencias al compararse por linajes.
Discusión
La incorporación de variables morfo- anatómicas a estudios citogenéticos en especies con poblaciones poliploides y altamente polimórficas (como en el caso de S. elaeagnifolium) aporta información novedosa y original, ya que hay pocos estudios que consideran estas perspectivas en conjunto, es decir, cuya finalidad es explicar la variabilidad observada en relación a los niveles de ploidía.
Se sabe que la poliploidía se comporta como una fuente de variabilidad genética, al alterar la estructura genómica de los organismos de manera directa, o indirectamente, al interactuar con otros factores, fisiológicos y ambientales (Lewis, 1980; Stebbins, 1985). La poliploidía puede considerarse en sí misma como un factor explicativo del polimorfismo morfológico, ya que uno de sus efectos notables es el aumento en el tamaño de las células, que puede verse reflejado en un aumento en la talla de la planta o de determinados órganos como las flores, los granos de polen o las semillas (Gould, 1957; Stebbins, 1971; Pegtel, 1999). Cambios en el volumen celular modifican la relación superficie-volumen, lo cual altera la tasa de procesos fisiológicos y metabólicos que dependen de sistemas regulatorios (Alcántar Vázquez, 2014). Siendo así, se espera que la mayoría de los rasgos morfológicos estén correlacionados positivamente con los niveles de ploidía. Sin embargo, lo hallado en este trabajo es que en S. elaeagnifolium, los rasgos morfo-anatómicos de las poblaciones estudiadas no responden linealmente a los efectos de la poliploidización.
Si bien el análisis de comparación de medias mostró que la mayoría de las variables no difieren entre citotipos, esto no implica que estas mismas variables, en conjunto (y sumadas a las variables climáticas) sean útiles para discriminar grupos de observaciones entre sí.
Por otro lado, se puede considerar que el conjunto de caracteres mostrados por un poliploide puede traer consigo una mayor adaptación al ambiente que ocupa o bien facilitar el cambio a un nicho ecológico distinto. Es decir, los cambios adaptativos originados como consecuencia de la poliploidización, pueden causar una diferenciación ecológica entre citotipos o niveles de ploidía (Mable, 2003; Comai, 2005; Otto, 2007).
Es importante precisar el papel discriminante de la variable precipitación media anual. Ya que esta es la única variable ambiental evaluada que discrimina entre niveles de ploidía, es la más relevante para la explicación de las diferencias observadas. La correlación significativa y positiva entre el nivel de ploidía y la precipitación corrobora el resultado de Scaldaferro et al. (2012), con los tetraploides distribuidos sobre la diagonal árida y los hexaploides en áreas más húmedas.
Las variables anatómicas de la epidermis no presentaron diferencias significativas entre los citotipos, excepto por el número de estomas, en donde los tetraploides arrojaron una media marginalmente inferior. Tal como documentan Gabriel et al. (2011), quienes evalúan la resistencia genética a estrés hídrico en variedades de Solanum tuberosum en invernadero, un número y un área estomática bajos ayudan a la resistencia y a la recuperación frente a un estrés hídrico. Altas densidades estomáticas están asociadas a elevadas conductancias estomáticas, elevadas tasas de transpiración y maximización de la difusión de dióxido de carbono durante períodos óptimos de fotosíntesis (Beaulieu et al., 2008, Burrows et al.,
. Asimismo, puede considerarse que la gran varianza del número de estomas en los diploides de S. elaeagnifolium, estaría en relación con la amplia distribución de este citotipo.
El tamaño de los estomas es un factor clave en el proceso de aclimatación al estrés hídrico, al existir una relación inversa entre el tamaño del estoma y la resistencia a la sequía (Strasburger et al., 1986; Aasaman et al., 2001). Esta variable tampoco presenta diferencias significativas al compararse entre ploidías. Sin embargo, el valor promedio es menor para tetraploides que para hexaploides. Este dato junto con el del número de estomas, podría sugerir que las diferencias en los valores de estas variables anatómicas se deben a cambios adaptativos al estrés hídrico. Si bien los valores medios no arrojaron diferencias significativas, el creciente aumento del área estomática entre niveles de ploidía (diploides < tetraploides < hexaploides) podría ser también una consecuencia directa de la poliploidización (Alcántar Vázquez,
. Los datos obtenidos no fueron suficientes para evaluar un efecto combinado del nivel de ploidía y el área geográfica.
Con respecto a los tricomas, se les atribuye un posible papel en la economía del agua al influenciar la capa límite de difusión de agua de la superficie transpirante de una hoja (Johnson, 1975; Cadena Iñiguez et al., 2001), así como también disminución de la tasa de evapotranspiración, enfriamiento o aislamiento de las hojas reflejando la radiación solar incidente y/o disipando el calor absorbido y protegiendo a la hoja contra el daño de los rayos UV (Johnson, 1975; Woodman & Fernandes, 1991; Roy et al., 1999; Barp et al., 2006; Pierce, 2007; Burrows et al., 2013). El tamaño de los tricomas es una característica que suele variar en respuesta al ambiente en el cual crece la planta (Johnson, 1975; García et al., 2000). En S. elaeagnifolium, Burrows et al. (2013) desechan la participación de los tricomas en la absorción de líquido y consideran como posibles funciones la de defensa contra la herbivoría y la disminución de la absorción de radiación. En nuestro caso, los tetraploides, son los que presentan mayores áreas de tricomas, y aunque si bien la diferencia entre ploidías no es estadísticamente significativa, esta variable es útil para explicar las similitudes que tienen las muestras a nivel global. Sería necesario un análisis relacionando área de los tricomas con la insolación y radiación para una mejor explicación del patrón observado.
Los tricomas peltados, como los de S. elaeagnifolium, tendrían un papel en la compensación entre la fotoprotección y la adquisición de agua de rocío (Pierce, 2007). Esto podría ser una explicación para la diferencia en el número de rayos del tricoma observada entre ploidías, ya que el citotipo hexaploide, en donde se hallaron tricomas con rayos supernumerarios, ocupa áreas del país con mayor precipitación media anual. Al mismo tiempo, debe considerarse el efecto de la poliploidía en la organogénesis. En Arabidopsis, los tricomas de las hojas tienen una morfología ramificada característica (Orr- Weaver, 2015), en donde el aumento de la ploidía da como resultado ramas adicionales, mientras que la reducción disminuye el número de ramas. Al estar los tricomas asociados con la resistencia a los insectos, es probable que la ramificación sea relevante y que la ploidía afecte esta función. En efecto, en S. elaeagnifolium, la cubierta densa de tricomas solapados tiene un efecto repelente contra herbívoros pequeños (Christodoulakis et al., 2009;
Burrows et al., 2013). Entre los enemigos naturales de S. elaeagnifolium en la región se cita al coleóptero Gratiana lutescens (Siebert, 1975; Vigna et al., 1981) que se alimenta vorazmente de las hojas. Se sabe que en S. sisymbriifolium las hojas mantenidas al sol desarrollan mayor cantidad de tricomas, los cuales impiden la alimentación de larvas de otro coleóptero, Gratiana spadicea (Boligon, 2007). Sin embargo, no hay estudios específicos sobre la interacción entre S. elaeagnifolium y los insectos antes mencionados, la cual debería ser revisada teniendo en cuenta el número de tricomas y de rayos y los niveles de ploidía.
Con respecto a la masa de las semillas, existen investigaciones sobre su compleja relación con el tamaño del genoma. Beaulieu et al., (2007) sostienen que en Angiospermas (incluido el Orden Solanales) las divergencias en el tamaño del genoma se correlacionan positivamente con las de la masa de semillas. Existe evidencia de que en algunas especies la masa de las semillas escala con la poliploidía (Stebbins, 1971; Bretagnolle et al., 1995; Beaulieu et al., 2007; García Osuna et al., 2015). En S. elaeagnifolium, los tetraploides muestran una semilla significativamente más pesada que los otros citotipos, pero la masa no se incrementa proporcionalmente con la ploidía. La masa, en cambio, podría estar afectada por factores tales como el número de frutos por planta. Esto, junto con la cantidad y peso de las semillas, se modifican en respuesta tanto a las condiciones ambientales que afectan la disponibilidad de recursos como al genotipo de la planta (Vaughton & Ramsey, 1998; Barthlott & Hunt, 2000; Vargas et al., 2003; Garrido S. et al., 2005; De Malach et al., 2019). En las muestras analizadas se halló una correlación negativa entre la precipitación media anual y la masa de las semillas, y una correlación positiva entre la estacionalidad y la masa de las semillas (Tabla 2). Murray et al. (2004) mencionan a la precipitación como una variable que puede influenciar la masa de las semillas, en relación con gradientes latitudinales y longitudinales. El aumento del gasto metabólico a temperaturas más altas puede estar relacionado con la persistencia de las semillas en el suelo. Los recursos de semillas se agotan más rápido cuando hace calor, y por lo tanto, en regiones tropicales y áridas, una masa de semillas más grande podría haber evolucionado en compensación (Murray et al., 2004). Las características de los bancos de semillas podrían explicar gran parte de la persistencia de algunas especies invasoras, como es el caso de S. elaeagnifolium (Mekki, 2007). La distribución más occidental del citotipo tetraploide coincide con la diagonal árida (Bruniard, 1982), en la provincia biogeográfica de Monte (Cabrera & Willink, 1982), tal como fuera notado previamente por Scaldaferro et al., (2012). Las semillas de los tetraploides serían más pesadas, no tanto por efecto de la poliploidía, sino mas bien por efecto de compensación de recursos. Esta interpretación también está sustentada en las correlaciones positivas entre la masa de las semillas y la longitud geográfica, mientras más al Oeste, más pesada la semilla (Tabla 2).
Con respecto a las variables medidas en la flor, sus valores medios no presentaron diferencias significativas entre ploidías. No obstante, existe una relación significativa entre los ejes NMDS1 y NMDS2 y estas variables florales. Dichas variables están además correlacionadas positivamente con la masa de las semillas. Existen evidencias de una relación entre masa de semilla y peso seco de la corola (Sakai & Sakai, 1994), que involucra mecanismos ecológicos y fisiológicos de polinización (Garrido J.L. et al., 2005), pero tales aspectos no han sido abordados aún para los distintos citotipos de S. elaeagnifolium.
Las variables florales presentaron también una correlación positiva entre ellas. Las diferencias de las distintas dimensiones entre los citotipos son alométricas: siempre hay una misma proporción entre las dimensiones de las anteras y el resto de las partes de la flor. Dentro de Solanum sect. Basarthrum, existe una correlación entre cantidad de polen y radio de la corola (Mione & Anderson, 1992), lo que sugiere que el tamaño de la corola habría evolucionado en relación a la función masculina, o que las flores más grandes producen más polen, mientras que la longitud del estilo estaría en relación con el tamaño del grano de polen (Plitmann & Levin, 1983; Vonhof & Harder, 1995).
La organización modular debe ser considerada al analizar las relaciones entre dimensiones de las partes de un organismo. Los módulos son subconjuntos de rasgos integrados (es decir, tienden a variar de manera coordinada) que varían de forma relativamente independiente de otros subconjuntos similares (Klingenberg, 2014). En el caso de S. elaeagnifolium, considerando la flor como un módulo, se hallaron relaciones isométricas entre las dimensiones de la corola y el pistilo, de la corola y los estambres y de los estambres y el pistilo (Tabla 2), pero también relaciones alométricas (cuanto mayor la corola más desiguales los estambres). Esto sería un indicio de que esta especie, a lo largo de su vasto rango de distribución, ajusta su biología floral de acuerdo a presiones del ambiente, que pueden ser bióticas (polinizadores) o abióticas (temperatura, precipitaciones, condiciones del suelo).
La heterantería (o heterandria, presencia de estambres notablemente desiguales en una misma flor) es una peculiaridad que ocurre en varios clados de Solanum. Las diferencias en el tamaño del estambre pueden afectar la longitud del filamento, la longitud de la antera o ambas (Levin et al., 2006; Bohs et al., 2007; Vallejo-Marin et al., 2009; Del Vitto & Petenatti, 2015). Su función presumiblemente sería depositar polen en una parte de la abeja donde no le es fácil quitárselo. Otra posibilidad es que el/los estambres largos funcionen como una plataforma de aterrizaje para los insectos visitantes, que luego trabajan las anteras restantes para el polen (Levin et al., 2007; Bohs et al., 2007; Vallejo-Marin et al., 2009; Del Vitto & Petenatti, 2015). En el caso de S. elaeagnifolium, la variación de longitud entre estambres, aporta a la diferenciación global. Los tetraploides, que habitan al oeste en la región de Monte, presentan flores con estambres más semejantes entre sí que los de las flores de los hexaploides y diploides. Si bien los visitantes florales has sido documentados en poblaciones de S. elaeagnifolium en Mendoza y San Luis (Jensen-Haarup, 1908; Jorgensen, 1909; Petenatti & Del Vitto, 1991), en Estados Unidos (Linsley & Cazier, 1963; Buchmann & Cane, 1989), y en áreas invadidas en Grecia (Tscheulin & Petanidou, 2013), estos estudios no contemplan las ploidías ni las medidas de los estambres.
Por otro lado, el coeficiente de variación de la longitud de los estambres está positivamente correlacionado con la longitud de los lóbulos corolinos y con la longitud del pistilo. Esto podría estar en relación con el tipo de insectos polinizadores. El cono anteral de Solanum presenta modificaciones de acuerdo a la especie de himenóptero que lo visita (Coleman & Coleman, 1982; Bohs et al., 2007; Del Vitto & Petenatti,
. En particular, en el clado Elaeagnifolium las anteras no son fuertemente conniventes, y sólo abejas grandes que puedan contactar tanto el estilo como las anteras son polinizadores eficientes, mientras que abejas pequeñas como halíctidos son "ladrones" de polen (Knapp et al., 2017). Por otro lado, mientras el tamaño de la abeja se incrementa, también aumenta el "buzz ratio" o frecuencia de vibración de los músculos del vuelo (De Luca et al., 2019). Dado que S. elaeagnifolium es una especie con distribución amplia, los polinizadores podrían variar de acuerdo a la región geográfica y consecuentemente esto podría afectar la forma de su cono anteral. Por el momento, solo se conoce un elenco de visitantes florales de S. elaeagnifolium (Knapp et al., 2017). Serán necesarios estudios más detallados, abarcando diploides y poliploides de toda el área de S. elaeagnifolium en Argentina, para establecer si existe una diferenciación mediada por polinizadores.
Conclusiones
En las muestras estudiadas, la relación entre el nivel de ploidía o el linaje genético y las características morfológicas de las plantas es apenas significativa (en cuanto número de estomas, masa de semilla, número de rayos del tricoma) o nula (resto de las variables).
El número de estomas por mm2 resultó ser diferente entre ploidías. Los datos de masa de semillas presentaron una gran dispersión y elevada varianza, siendo la media marginalmente superior en los tetraploides respecto de las otras dos ploidías. Las diferencias de medias en estas dos variables son difícilmente explicables como un efecto de la duplicación de la dotación cromosómica.
En la mayoría de los casos el número de rayos laterales de los tricomas estrellados es 16, pero se encontraron entre los hexaploides muestras con un número mayor, que los hace marginal pero significativamente distintos.
En ningún rasgo se comprobó que sus valores medidos variaran de manera proporcional al aumento de ploidía, como ha sido visto en otras especies con series poliploides. Ninguna de las variables analizadas mostró diferencias al compararse por linajes.
Los valores encontrados podrían explicarse, más que por un efecto de la ploidía creciente, por causas tales como múltiples orígenes independientes de la poliploidía, plasticidad fenotípica y presiones del ambiente (bióticas y abióticas). Ensayos con materiales de estas mismas poblaciones pero en invernadero, con condiciones controladas, podrían arrojar luz sobre tales presiones ambientales.
Se necesitan más estudios y la incorporación de otras variables para determinar si hay otras características (por ejemplo, datos de ADN nuclear, fito-histoquímicos, de preferencia de nichos ecológicos) que distinguen los citotipos y/o los linajes genéticos.












 uBio
uBio