Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Revista industrial y agrícola de Tucumán
versão On-line ISSN 1851-3018
Rev. ind. agric. Tucumán vol.95 no.2 Las Talitas dez. 2018
Desarrollo del evento transgénico de caña de azúcar TUC 87-3RG resistente a glifosato
Aldo Noguera*, Ramón Enrique*, Santiago Ostengo**, Maria Francisca Perera**, Josefina Racedo**, Diego Costilla***, Silvia Zossi***, María Inés Cuenya**, María Paula Filippone**, Björn Welin** y Atilio Pedro Castagnaro**
* Instituto de Tecnología Agroindustrial del Noroeste. Los autores contribuyen de manera equivalente al presente trabajo.
Argentino (ITANOA).
**ITANOA, Consejo Nacional de Investigaciones Científicas (CONICET).
***Estación Experimental Agroindustrial Obispo Colombres (EEAOC). noguera@eeaoc.org.ar
RESUMEN
La variedad comercial de caña de azúcar RA 87-3 se transformó mediante biobalística con una construcción genética que porta el gen epsps de la cepa CP4 de Agrobacterium tumefaciens, y el gen nptII que confieren resistencia a glifosato y resistencia a kanamicina/geneticina, respectivamente. Las líneas transformadas fueron multiplicadas en invernadero y se evaluó la resistencia al herbicida utilizando diferentes concentraciones de glifosato (3, 4 y 8 l/ha). Las líneas resistentes al herbicida (RH) se evaluaron en campo para confirmar la resistencia a glifosato (3 l/ha) y realizar un análisis preliminar del comportamiento de estas con respecto al cultivar parental. Todas las líneas transformadas mantuvieron la resistencia al herbicida pero muchas mostraron cambios fenotípicos. Las diez líneas RH que resultaron muy parecidas a la variedad RA 87-3 se analizaron fenotípica y genéticamente utilizando los nueve descriptores morfológicos obligatorios propuestos por la International Union for the Protection of New Varieties of Plants (UPOV) y con 339 marcadores moleculares, respectivamente. Seis líneas de las anteriores presentaron cambios morfológicos y genéticos menores y fueron seleccionadas para ensayos en campo durante dos ciclos vegetativos (caña planta y soca 1), en dos áreas de producción de Argentina. Las seis líneas RH presentaron características agronómicas, industriales y de composición química indistinguibles respecto del cultivar parental. La herencia estable del gen CP4 epsps fue confirmada mediante ensayos de RT-qPCR y Southern blot en diferentes generaciones clonales (caña planta y soca 1). Los estudios confirmaron la utilidad de la transformación genética como una herramienta complementaria a la mejora clásica, y destaca la ventaja del uso de los descriptores de UPOV junto con los marcadores moleculares para una selección temprana de eventos transgénicos que tengan un alto parecido con el genotipo parental. Tomando en cuenta los resultados, una de las seis líneas estudiadas se seleccionó para una posible liberación comercial, la cual debe ser sometida a evaluación por parte de los entes regulatorios (CONABIA, SENASA y DNMA).
Palabras clave: Saccharum spp., Glifosato, gen CP4 epsps, resistencia a herbicida.
ABSTRACT
Development of the transgenic sugarcane event TUC 87-3RG resistant to glyphosate
The commercial variety of sugarcane RA 87-3 was transformed by biobalistic with a genetic construct that carries the epsps gene of the CP4 strain of Agrobacterium tumefaciens, and with the nptII gene that confer resistance to glyphosate and resistance to kanamycin / geneticin, respectively. Transformed lines were multiplied in greenhouse and resistance to the herbicide was evaluated using different concentrations of glyphosate (3, 4 and 8 l / ha). Resistant lines to the herbicide (RH) were evaluated in the field to confirm resistance to glyphosate and to carry out a preliminary analysis of their behavior with respect to the parental cultivar. All transformed lines maintained resistance to the herbicide but many showed phenotypic changes. Ten RH lines that were very similar to the RA 87-3 variety were analyzed phenotypically and genetically using the 9 compulsory morphological markers proposed by The International Union for the Protection of New Varieties of Plants (UPOV) and with 339 molecular markers, respectively. Six lines of the previous ones presented minor morphological and genetic changes and were selected for field trials during two vegetative cycles (cane plant and ratoon 1), in two production areas of Argentina. The six RH lines presented agronomic, industrial and chemical composition characteristics indistinguishable from the parental cultivar. The stable inheritance of the CP4 epsps gene was confirmed by RT-qPCR and Southern blot assays in different clonal generations (growth cycles). Our studies confirmed the usefulness of genetic transformation as a complementary tool to classical improvement and highlight the advantage of the use of UPOV descriptors together with molecular markers for an early selection of transgenic events that have a high similarity with the parental genotype. Taking into account the results, one of the six lines evaluated was proposed for a possible commercial release, under the requirements of regulatory affairs offices (CONABIA, SENASA and DNMA).
Key words: Saccharum spp., glyphosate, CP4 epsps gene, herbicide resistance.
INTRODUCCIÓN Y ANTECEDENTES
La caña de azúcar (Saccharum spp.) es el principal cultivo productor de azúcar y representa más del 70% la producción mundial (Dillon et al., 2007). Adicionalmente, hay un interés creciente en la expansión y el mejoramiento de este cultivo debido a que es una importante fuente de biomasa para la producción de energía, además de ser una materia prima promisoria para la obtención de polímeros y químicos con valor agregado. Debido a la extrema complejidad del genoma de la caña de azúcar (Grivet et al., 2001) y a que el mejoramiento clásico es lento y laborioso, en muchos casos, los caracteres de interés agronómico e industrial no se encuentran en el germoplasma cultivado ni en especies emparentadas, resultando prácticamente imposible la introgresión de dichos caracteres a través de cruzamientos sexuales.
Consecuentemente, la transformación genética es una herramienta valiosa que permite superar estas limitaciones, y posibilita la introducción de caracteres de interés para lograr incrementos de rendimiento y disminución de costos tendientes a una producción más sustentable. Varios genes han sido introducidos por ingeniería genética en forma exitosa en caña de azúcar, por ejemplo, el gen bar que confiere resistencia a glufosinato de amonio (Enriquez-Obregón et al., 1998; Gallo Mehager and Irvine 1996; Falco et al. 2000; Leibbrandt and Snyman 2001); el gen cry1A que otorga resistencia a insectos (Rapulana and Bouwer, 2013) y el gen de la isomaltulosa sintasa para la producción del disacárido isomaltulosa (Basnayake et al., 2012). Sin embargo, hasta el presente solo se han informado dos eventos transgénicos de caña de azúcar aprobados para su comercialización, una variedad tolerante a la sequía en Indonesia en 2013 (James, 2013) y un evento resistente a insectos liberado al cultivo comercial en Brasil en el 2017 (Mano, 2017).
Para llevar a cabo el proceso de desregulación comercial de un cultivo transgénico en Argentina, los ensayos en campo son esenciales para evaluar adecuadamente el impacto potencial de este en los ambientes agrícolas y en la seguridad alimentaria. Se requieren también estudios moleculares y bioquímicos para verificar la expresión estable de los genes introducidos en la planta, además de evaluar la composición química y las características agronómicas.
Estos dos últimos aspectos son especialmente importantes en cultivos como la caña de azúcar, donde se conoce que la transformación genética y los procesos de regeneración in vitro pueden provocar importantes cambios epigenéticos y genéticos, lo que genera una descendencia muy heterogénea con cambios fenotípicos notables (Gallo- Meagher and Irvine, 1996; Arencibia et al., 1999; Arencibia et al., 2000; Sala et al., 1999; Gilbert et al, 2009). Por lo tanto, para una posible liberación comercial es de suma importancia obtener líneas transformadas con herencia estable para el carácter(es) introducido(s), con un rendimiento agronómico e industrial comparable al cultivar elite parental.
El desarrollo de cultivos resistentes al glifosato sigue siendo una de las aplicaciones comerciales más importantes de la ingeniería genética vegetal (Green, 2009b) y hasta el momento hay siete cultivos resistentes al glifosato liberados comercialmente en el mundo, incluidos alfalfa, colza, col, algodón, maíz, soja y remolacha azucarera (Green, 2009a). En 2017 se plantaron cultivos modificados genéticamente (GM) en más de 189,8 millones de hectáreas en el mundo, y los cultivos GM resistentes a herbicidas (RH) representaron el 47% del área total de cultivos GM. El mismo año se plantaron en Argentina aproximadamente 23 millones de hectáreas con cultivos transgénicos RH, de los cuales el 77% corresponde a la producción de soja (ISAAA 2017).
Mediante estudios realizados en caña de azúcar se han estimado pérdidas de hasta un 70% de rendimiento debido a las malezas, dependiendo de la gravedad de la infestación (Gupta, 1960; Ibrahim, 1984). En Argentina, más de 300 especies de malezas afectan el cultivo de caña de azúcar y las pérdidas de rendimientos se han estimado en hasta 50% en algunas áreas de producción (Olea et al., 2009). Los altos costos inherentes al control de las malezas y la creciente preocupación por el medio ambiente han llevado a la búsqueda de alternativas más baratas y más efectivas, como el uso de herbicidas biodegradables de amplio espectro como el glifosato y el glufosinato de amonio.
Por otro lado, es importante destacar que para introducir cualquier material transgénico al cultivo comercial, es fundamental llevar a cabo un proceso denominado desregulación, el cual es un requisito indispensable en todos los países donde se comercializará dicho material transgénico. En nuestro país existe, desde el año 1991, un sólido sistema regulatorio de Organismos Vegetales Genéticamente Modificados (OVGM), de manera que cada evento debe atravesar un proceso de evaluación de bioseguridad para poder llegar al mercado. Los pasos del sistema regulatorio son: i) evaluación agroecológica, conducida por la Comisión Nacional Asesora en Biotecnología Agropecuaria (CONABIA); ii) evaluación de la inocuidad alimentaria, realizada por el Servicio Nacional de Sanidad y Calidad Agroalimentaria (SENASA) y iii) evaluación de la conveniencia comercial de la aprobación del evento para su producción y comercialización, lo cual está a cargo de la Dirección Nacional de Mercados Agroalimentarios (DNMA).
En el presente trabajo se demuestra que la transformación de la caña de azúcar mediante bombardeo con microproyectiles permite recuperar líneas transformadas genéticamente muy similares a la variedad élite parental. Se obtuvo una línea RH a partir de un número relativamente pequeño de eventos de transformación, la cual presentó además características fenotípicas, agronómicas e industriales similares a la variedad de origen, considerado esto un prerrequisito para una posible liberación comercial.
MATERIALES Y MÉTODOS
Material vegetal y condiciones de cultivo in vitro
Se prepararon callos embriogénicos de la variedad elite parental RA 87-3 para realizar el bombardeo con microproyectiles recubiertos con el ADN de interés, como se describe en Vellicce et al. (2011).
Construcción genética y transferencia de genes
El plásmido pEA1 contiene los genes sintéticos CP4 epsps y nptII. Ambos genes se fusionaron a promotores constitutivos fuertes para favorecer la expresión en plantas monocotiledóneas, el promotor de ubiquitina de maíz para el gen nptII y el promotor de actina de arroz para el gen CP4 epsps. El gen CP4 epsps fue fusionado con la secuencia codificante del péptido de tránsito de cloroplasto (cTP4) de Petunia hybrida. La construcción genética completa fue proporcionada por Biosidus AG SA. Para el bombardeo de callos embriogénicos se utilizaron micropartículas de tungsteno (M10, con un diámetro promedio de 0,8 μm), sobre las cuales se precipitaron 3,2 μl del plásmido pEA1. El bombardeo se realizó utilizando un acelerador de micropartículas en condiciones de vacío con una presión de helio de 1200 psi, desarrollado en el Centro de Tecnología Canavieira (CTC) según el modelo descrito por Finer et al. (1992). Las condiciones de bombardeo fueron optimizadas utilizando el plásmido pFN1 que contiene el gen reportero de `-glucuronidasa (gusA) (Vellicce et al., 2011).
Condiciones de selección, regeneración y micropropagación
La selección de las plantas transformadas se realizó utilizando un medio de cultivo que contenía 45 mg l-1
de geneticina según Bower et al. (1996) con modificaciones. Los brotes seleccionados se regeneraron en medio suplementado con 0,7 mg de BAP l-1 sin 2,4-D con un fotoperíodo de 16 h luz y 8 h de oscuridad. Posteriormente, los brotes regenerados se micropropagaron y enraizaron de acuerdo con un protocolo previamente optimizado para la variedad de caña de azúcar RA 87-3 (Noguera et al., 2010) y se aclimataron bajo condiciones controladas en invernadero (2500 lux y 80-100% de humedad).
Ensayos de resistencia a herbicidas en condiciones de invernadero
Para evaluar la resistencia al glifosato de las líneas transformadas, las plantas se rociaron con una solución de glifosato a distintas concentraciones (3, 4 y 8 l/ha) de una formulación comercial (Helm 48% p/v, Helm Argentina) y las plantas sobrevivientes se evaluaron a una, dos y cuatro semanas posteriores a la aplicación del herbicida.
Detección de plantas transgénicas por PCR
A partir de 200 mg de hojas molidas con N2 provenientes de 88 líneas regeneradas resistentes a la geneticina y de plantas control no transformadas, se realizó la extracción de ADN genómico utilizando el método descripto por Aljanabi (1999). Con la finalidad de detectar las líneas genéticamente transformadas, se amplificó mediante PCR un fragmento de 561 pb del gen epsps usando los cebadores EPA-F (5'- GCTCGATTTCGGCAATGC-3') y EPA-R (5'- GTTTGATGACTTCGATGTCGG-3'). La reacción de amplificación por PCR se realizó durante 30 ciclos de acuerdo con las siguientes condiciones: desnaturalización a 95ºC durante 30 s, hibridación a 64ºC durante 30 s y elongación a 72ºC durante 60 s.
Determinación del contenido de clorofila
Se colectaron muestras a diferentes tiempos (0, 1, 2 y 4 semanas) de las líneas transgénicas en invernadero (G) y de plantas no transformadas tratadas con 3 l/ha de glifosato. La extracción del contenido total de clorofila se realizó a partir de un disco de hoja de caña de azúcar de aproximadamente 15 mg con 500 μl de solución de acetona al 80%. Las muestras se incubaron durante una hora a 4ºC y luego se diluyeron 10 veces para la cuantificación espectrofotométrica. El contenido total de clorofila (a y b) se midió con un espectrofotómetro UVVisible Agilent 8453 (EE.UU.), siguiendo el método descrito por Lichtenthaler (1987).
PCR cuantitativa en tiempo real
Para la extracción del ARN total se colectaron hojas jóvenes de las líneas transgénicas 28 y 37 (RH) y de la variedad parental no transformada RA 87-3, provenientes de cuatro ciclos consecutivos de crecimiento (invernadero (G); caña planta, P; soca uno, R1 y soca dos, R2). El ARN total se aisló de acuerdo con las instrucciones del fabricante (SV Total RNA Isolation Kit, Promega, EE.UU.). La síntesis del ADNc se realizó con la transcriptasa reversa SuperScriptII (Invitrogen) a partir de 1 μg de ARN total tratado con DNasa y oligo-dT15 como cebadores. El ADNc sintetizado se usó para realizar la PCR cuantitativa en tiempo real (RT-qPCR). Las reacciones realizaron en presencia del colorante SYBR green (Biodynamics, Argentina) y se monitorearon con el sistema de PCR en tiempo real Mini Opticon (BioRad, EE.UU.). En el estudio se usaron los siguientes cebadores: EPS4-F (5'- CGCGCATCACCGGCCTTCTGG-3') y EPS4-R (5'- CCGCCATTGCCGACGCCATC-3'). Como gen de referencia se usó un fragmento de actina de caña de azúcar y se amplificó usando el cebador directo ACT2-F (5'-AATGGTCAAGGCTGGTTTTG-3') y el cebador inverso ACT2-R (5'-CCTCTCTTGGACTGTGCCTC-3'). Todos los amplicones tienen un tamaño de aproximadamente 125 pb. La mezcla de reacción contenía 1x SYBR Green PCR MasterMix (Mezcla Real®, Biodynamics, Argentina); 0,4 μM de los cebadores directo y reverso y 1 μl de ADNc (dilución 1: 5) en un volumen total de 15 μl. La amplificación por PCR se realizó durante 39 ciclos de acuerdo con las siguientes condiciones: desnaturalización a 95ºC durante 15 s, hibridación a 60ºC durante 30 s y extensión a 68ºC durante 40 s. Las hojas provenientes de plantas no transgénicas de RA 87-3 sirvieron como muestra de referencia, y los resultados se normalizaron frente a la amplificación del gen de actina. La expresión relativa de los transcriptos se calculó utilizando los valores de ciclo umbral (Ct) obtenidos de cada muestra de la siguiente manera: expresión relativa = 2-, siendo Ct = muestra Ct - actina Ct y Ct = muestra Ct - Muestra de Ct ref (Livak and Schmittgen, 2001). Los valores promedio se calcularon a partir de muestras ensayadas por triplicado.
Resistencia al glifosato y características de crecimiento fenotípico en el campo
Se pulverizaron plantas de caña de azúcar (P) de tres meses de edad con una solución 3 l/ha de glifosato de una formulación comercial (Helm 48% p/v, Helm Argentina) y se evaluaron los resultados 15 y 30 días después de la aplicación del herbicida. Diez plantas de caña de azúcar (10 meses de edad) de cada una de las líneas transgénicas (6, 15, 18, 22, 23, 27, 28, 29, 36 y 37) fueron evaluadas en cuanto a sus caracteres cualitativos y cuantitativos siguiendo las pautas propuestas por la UPOV (2005) para la diferenciación de variedades de Saccharum L. En la evaluación fenotípica se utilizaron los nueve caracteres obligatorios exigidos por la UPOV para identificar las variedades de caña de azúcar.
Caracterización genotípica mediante marcadores moleculares
El ADN total se extrajo por el método de CTAB descripto anteriormente (Aljanabi, 1999) a partir de tejido molido con N2 de las siguientes líneas resistentes al glifosato: 6, 15, 18, 22, 23, 27, 28, 29, 36 y 37, y de la variedad parental RA 87-3 micropropagada in vitro y de manera convencional, como control de genotipo. También se incluyeron otras tres variedades de caña de azúcar de la colección de germoplasma de la EEAOC (LCP 85-384, TUC 97-7 y TUC 95-24) para validar la utilidad de la técnica molecular. Para caracterizar los genotipos se utilizaron nueve combinaciones de cebadores para generar marcadores moleculares TRAP (de sus siglas en inglés de Target Region Amplified Polymorphism). Los cebadores sentido (Alwala, 2006) fueron diseñados a partir de cuatro genes asociados con el metabolismo de la sacarosa, y los cebadores arbitrarios antisentido se obtuvieron de Li and Quirós (2001). La mezcla de reacción para la amplificación contenía: 1X tampón Taq ADN polimerasa; 2,5 mM MgCl2, 0,1U Taq ADN polimerasa; 0,16 μM de ambos cebadores (Invitrogen, Life Technologies); 88 μM de cada dATP, dTTP y dGTP; 72 μM de dCTP; 1,4 μM de Cy5.5-dCTP (GE Health care Life Sciences) y 100 ng de ADN. Los parámetros de amplificación fueron: 1 ciclo a 94ºC durante 4 min; 35 ciclos a 94ºC durante 45 s, 45ºC durante 45 s y 72ºC durante 1 min; y 1 ciclo a 72ºC durante 7 min. Los productos de amplificación se separaron mediante electroforesis en geles de poliacrilamida en condiciones desnaturalizantes en el equipo Li-cor DNA Analyzer 4300. Los geles se analizaron usando el software SagaMX AFLP® (LI-COR), considerando todas las bandas amplificadas y evaluadas de manera dominante.
Los datos de marcadores moleculares y también los de caracteres morfológicos se transformaron en matrices binarias de 0 o 1. La similitud se calculó a partir de las matrices de datos moleculares, morfológicos o la combinación de ambas empleando el coeficiente de Jaccard (Sneath and Sokal, 1973a). Los análisis de conglomerados se llevaron a cabo utilizando el análisis de similitud de McQuitty (UPGMA) (Sneath and Sokal, 1973b). El módulo FIND se utilizó para identificar todos los árboles que fueron compilados por el módulo CONSEN para evaluar la solidez de la topología del árbol. Todos los cálculos se llevaron a cabo utilizando el software InfoStat (Di Rienzo et al., 2009).
Análisis de Southern blot
Se amplificaron por PCR un fragmento de 428 pb del gen epsps y un fragmento de 598 pb del gen nptII a partir de pEA1. Dichos fragmentos fueron utilizados como sondas para hibridar con el ADN genómico extraído de hojas molidas con N2 de plantas resistentes al glifosato y plantas no transformadas, en dos edades de cultivo (P y R1). Los ADN genómicos (aproximadamente 20 μg por muestra) y el plásmido pEA1 (10 μg) fueron digeridos con las enzimas de restricción MfeI o Bpu10I, separados por electroforesis mediante un gel de agarosa al 0,8% y luego transferidos a una membrana de nylon (Hybond+, Amersham). Las membranas se hibridaron con los fragmentos del gen epsps y nptII marcados previamente con 32P-dCTP durante toda la noche a 65ºC, utilizando un kit (Prime-a-Gene, Promega). A continuación, las membranas se lavaron tres veces con diferentes concentraciones de buffer de citrato sódico salino (SSC) (2X; 1X y 0,5 X) suplementados con SDS al 0,1% y fueron expuestas a una pantalla de fósforo (GE) para ser escaneadas, luego de 24 h de exposición, utilizando un equipo PhosphorImager Storm 845 (GE).
Comparación de caracteres agronómicos y químicos entre las seis líneas resistentes al herbicida y su variedad parental RA 87-3
Seis líneas transgénicas RH (15, 18, 22, 27, 28 y 37) y la variedad parental RA 87-3 no transformada fueron evaluadas en dos localidades ubicadas en áreas de producción independientes del noroeste de Argentina (provincias de Tucumán y Salta). Los ensayos se implantaron, en cada sitio, según un diseño de bloques completos al azar (RCBD) con tres repeticiones, donde cada unidad experimental estuvo conformada por una parcela de tres surcos de siete metros de largo. Se plantaron dos tratamientos de la variedad parental RA 87-3, uno con caña semilla proveniente de vitroplantas y otro con caña semilla propagada en campo de manera convencional, ya que informes anteriores han mostrado que en algunos casos el material vegetal propagado in vitro muestra diferencias de crecimiento en comparación con las plantas propagadas en campo (Arencibia et al., 1999; Arencibia et al., 2000; Sala et al., 1999). En las edades de corte de caña planta y soca 1 se evaluaron los siguientes caracteres agronómicos: rendimiento de caña por parcela (kg de caña/parcela), altura, peso y diámetro del tallo. Estos últimos se evaluaron a partir de una muestra de diez tallos tomados al azar del surco central de cada parcela. Estas mismas muestras se procesaron en laboratorio para realizar las siguientes determinaciones químicas: contenido de proteína, fibra en tallo y contenido de sacarosa en jugo. Las proteínas totales se determinaron mediante el método Kjeldahl, siguiendo los procedimientos AOAC (2009), utilizando un destilador Buchi B-324. La evaluación del contenido de fibra detergente ácida (FDA) y fibra detergente neutra (FDN) se realizó utilizando un analizador de fibra ANKOM 2000, de acuerdo con la metodología ANKOM (2006a, 2006b). La determinación del contenido de sacarosa se realizó mediante cromatografía líquida de alta resolución (HPLC), utilizando una bomba y un inyector automático Alliance. La columna utilizada fue Sugar Pak (Waters), utilizando como solución de EDTA cálcica 0,05 M en fase móvil a un caudal de 0,5 ml/min y detector de índice de refracción (Waters). Para cada variable, los datos se analizaron estadísticamente mediante ANOVA y prueba de LSD de Fisher (nivel de significación 5%).
RESULTADOS Y DISCUSIÓN
Transformación y selección de líneas transgénicas resistentes al glifosato
Se bombardearon mil setecientos veintiocho (1728) callos de la variedad comercial de caña de azúcar RA 87-3 con el plásmido pEA1 que contiene el gen sintético CP4 epsps, que confiere resistencia al glifosato, y el gen nptII de E. coli utilizado para la selección de células transformadas con el antibiótico kanamicina/geneticina. Ochenta y ocho (88) plántulas fueron seleccionadas como resistentes a la geneticina, pero solo 43 líneas sobrevivieron el proceso completo de regeneración in vitro. De estas plantas, 36 resultaron PCR positivas (eficiencia de transformación del 2,1%) para el gen CP4 epsps (Figura 1).
Figura 1: Análisis molecular por PCR de las líneas transgénicas. 1-13 líneas evaluadas luego de la selección; c, plantas no transformadas utilizadas como control negativo; +, dilución 1/1000 del plásmido pEA1 utilizado como control positivo; M, marcador molecular 100bp. La flecha negra indica un producto de PCR de 561 pb correspondiente a un fragmento del gen epsps.
Esta eficiencia es ligeramente más alta que la informada anteriormente en caña de azúcar (1,6%) por Vickers et al., (2005).
Con el fin de determinar la resistencia frente al glifosato, todas las plantas transformadas, incluidas las siete plantas PCR-negativas, se micropropagaron y se evaluaron en condiciones de invernadero (G). De las 43 líneas, 17 resultaron resistentes al glifosato (3l/ha), mientras que cinco líneas mostraron resistencia intermedia y 21 líneas no mostraron resistencia al herbicida a las concentraciones probadas. Muchos reportes afirman que la micropropagación in vitro de la caña de azúcar puede causar plantas menos vigorosas y productivas (Gilbert et al., 2009; Vickers et al., 2005; Basnayake et al., 2012; Hoy et al., 2003). En un reciente estudio en Australia, se compararon siete variedades transgénicas cultivadas in vitro con respecto a la variedad propagada en campo y se observó que estas eran más pequeñas con tallos más delgados que aquellas propagadas convencionalmente en campo. Sin embargo, ya en el segundo año de crecimiento, los rendimientos y los fenotipos fueron indistinguibles entre ambos tipos de propagación. La experiencia de nuestro programa de propagación in vitro en la EEAOC confirma esta observación, ya que el primer año de plantación de plántulas propagadas in vitro puede en algunos casos dar lugar a tallos más cortos y delgados; sin embargo, durante la propagación subsiguiente en campo, tales diferencias desaparecen (datos no mostrados). Por la razón antes mencionada, todas las líneas transgénicas se micropropagaron bajo un esquema optimizado que minimiza la variación somaclonal, y luego se propagaron en campo durante al menos dos años, previo a la evaluación de los rasgos agronómicos e industriales.
Pruebas de resistencia al glifosato en campo y evaluación de características fenotípicas de las líneas transformadas
Las 17 líneas resistentes al glifosato, seleccionadas en invernadero, fueron plantadas en un ensayo en campo para confirmar la resistencia al herbicida y evaluar preliminarmente el crecimiento y el fenotipo de las mismas. Todas las líneas evaluadas mantuvieron la resistencia al glifosato observada, pero varias de ellas mostraron una clara diferencia de crecimiento en comparación con el genotipo parental, similar a lo observado en el invernadero. Diez líneas transgénicas, seleccionadas por su parecido fenotípico con la variedad parental, se analizaron morfológicamente utilizando los nueve descriptores obligatorios propuestos por la UPOV. Cuando se analizaron dichos descriptores ninguna de las diez líneas evaluadas mostró una identidad del 100% con la variedadparental RA 87-3; pero varias de ellas solo difirieron encaracterísticas fenotípicas menores como la coloración de los tejidos vegetales (Tabla 1).
Tabla 1 Caracterización de diez líneas transgénicas y de la variedad parental RA 87-3 utilizando los descriptores morfológicos de la UPOV.
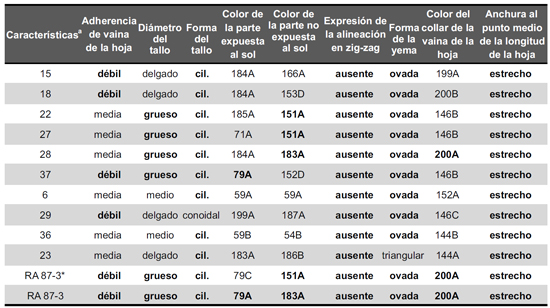
Cuatro de las líneas RH evaluadas (6, 23, 29 y 36) presentaron cambios en más del 50% de los descriptores analizados con respecto a la variedad parental y, por lo tanto, fueron descartadas. Las seis líneas restantes (15, 18, 22, 27, 28 y 37) que mostraron identidad en al menos cinco de los nueve descriptores se seleccionaron para un análisis posterior como posibles candidatas para una liberación comercial. Las dos líneas destacadas en este estudio fueron las líneas 28 y 37 que presentaron seis y siete coincidencias en los descriptores de la UPOV respecto a la línea parental, respectivamente. Se ha demostrado anteriormente que los descriptores morfológicos de la UPOV combinados con marcadores moleculares pueden utilizarse en caña de azúcar para estimar la diversidad genética, elegir cruzamientos para identificar líneas superiores y proteger los derechos de propiedad intelectual (Perera et al., 2012).
Niveles de expresión del gen CP4 epsps en líneas transgénicas tratadas con glifosato y su relación con el contenido de clorofila
Se utilizó PCR cuantitativa en tiempo real (RTqPCR) para medir los niveles de expresión del gen CP4 epsps en las seis líneas resistentes al glifosato (15, 18, 22, 27, 28 y 37) y evaluar una posible relación con la resistencia al herbicida. Se encontró que los niveles de expresión génica relativa aumentaron de 15 a 25 veces en las seis líneas examinadas, en comparación con las plantas control no transformadas. Los niveles de expresión de CP4 epsps más elevados se observaron en las líneas 18, 28 y 37 (Figura 2a).
Para cuantificar y visualizar la resistencia al glifosato, se tomaron muestras de hojas (G) de las seis líneas transformadas y de plantas control no transformadas (RA 87-3), luego del tratamiento con glifosato (3 l/ha).
El contenido de clorofila relacionado con la capacidad de resistencia de las líneas fue similar en todas las concentraciones de glifosato a las 0, 1, 2 y 4 semanas después de la aplicación. Si bien este contenido fue similar al de las plantas no transformadas no tratadas, se observaron diferencias sustanciales con respecto a las plantas no transformadas tratadas con herbicida (Figura 2b-e). Las plantas tratadas no transformadas perdieron la mayor parte del contenido de clorofila una semana después del tratamiento, mientras que las líneas transformadas resistentes retuvieron el contenido de clorofila de manera similar a las plantas control no tratadas (datos no mostrados). Las líneas transgénicas con el nivel de expresión del gen CP4 epsps más bajo (22) y más alto (37) mostraron una resistencia similar, lo que sugiere la ausencia de una relación directa entre la resistencia al herbicida y los niveles de expresión a la concentración deglifosato aplicada (datos no mostrados). En este sentido,los ensayos de Southern blot son necesarios para evaluar el número de insertos en las líneas transgénicas y poder correlacionar con el nivel de expresión del/los transgen/es. Un alto número de insertos podría correlacionarse con una mayor producción de proteína, como se informó anteriormente (Altpeter, 2005), y por lo tanto a una mayor resistencia al glifosato. Sin embargo, un estudio reciente en caña de azúcar no mostró correlación entre los niveles de expresión génica y el número de inserciones del transgén (Jackson et al., 2013).
Figura 2: Cuantificación de los niveles de expresión del gen epsps por RT-qPCR y evaluación de la resistencia al glifosato en las líneas transgénicas.
(a) PCR cuantitativa en tiempo real de líneas transgénicas. El promedio de la variación (n veces) en los niveles de expresión del gen epsps en seis líneas transgénicas con respecto a la planta no transformada RA 87-3 fue calculado a partir de muestras por triplicado.
(b-e) Cambios fenotípicos en plantas de la línea transformada (37) a las 0, 1, 2 y 4 semanas, respectivamente, después del tratamiento con herbicida (3 l/ha de glifosato). Las plantas RA 87-3 no transformadas se usaron como control. Para ambos estudios se usaron muestras de plantas cultivadas en invernadero (G).
Evaluación genotípica de líneas transformadas usando marcadores moleculares
Para establecer si la semejanza morfológica entre las líneas RH y la variedad parental tiene una correlación a nivel genético, se usaron nueve marcadores moleculares independientes de tipo TRAP para caracterizar las diez líneas transgénicas evaluadas previamente con los marcadores morfológicos. Además de las líneas RH, se incluyeron tres variedades de caña de azúcar, de las cuales dos presentan un alto grado de similitud (TUC 95-24 y TUC 97-7) y un genotipo más distante (LCP 85-384) a RA 87-3 (Perera et al., 2012). Se obtuvo un total de 339 bandas no ambiguas, de las cuales 212 (62%) fueron polimórficas, con un promedio de 37 bandas por combinación de cebador (datos no mostrados).
En el dendrograma obtenido (Figura 3) no se detectaron diferencias genéticas en cinco (15, 18, 22, 28 y 37) de las seis líneas que muestran un alto parecido fenotípico con el genotipo parental RA 87-3. La línea restante (27) presentó dos bandas diferentes de las 277 que caracterizaron a la variedad (99% de similitud), mientras que las cuatro líneas transformadas que exhibieron un fenotipo distinto a la línea parental RA 87-3 presentaron un mayor grado de polimorfismo (92 - 95% de similitud). Sin embargo, las diez líneas RH caracterizadas genéticamente mostraron una mayor similitud con RA 87-3 (> 92%) en comparación con el 78% encontrado para variedades genéticamente cercanas y 68% para la variedad genéticamente distante de la colección de germoplasma de la EEAOC.
Figura 3: Dendrograma de las líneas transgénicas de RA 87-3 y de cuatro genotipos de caña de azúcar en base al análisis de 339 alelos originados de nueve combinaciones de primer TRAP utilizando el coeficiente de Jaccard y el método de agrupación UPGMA con InfoStat, presentado como distancia (1-S, S: similitud). Las líneas representaron similitudes de 0.98; 0.92; 0.89; 0.86; 0.75 and 0.65.
Nuestros resultados muestran que el uso de marcadores moleculares TRAP constituye un primer enfoque, rápido y reproducible para seleccionar líneas transgénicas promisorias que se asemejen a su genotipo parental. Además, podría constituir un enfoque más adecuado para detectar cambios genéticos con respecto a otras técnicas de marcadores moleculares como RAPD (Taylor et al., 1995) y AFLP (Joyce, 2014), previamente evaluadas en caña de azúcar, con las cuales no se pudieron detectar diferencias genéticas entre clones con variaciones fenotípicas evidentes. Este resultado es algo sorprendente considerando la naturaleza de los marcadores TRAP que están orientados en loci específicos y no están distribuidos en todo el genoma, por lo tanto el elevado polimorfismo encontrado requiere de estudios complementarios para su interpretación.
Dado que los datos de marcadores moleculares se pueden combinar con los de rasgos morfológicos para caracterizar mejor a los genotipos y revelar las relaciones genéticas y la diversidad genética (Perera et al., 2012), se construyó un dendrograma combinando ambos tipos de datos (Figura 4). La caracterización morfológica de las variedades TUC 95-24, TUC 97-7 y LCP 85-384 se obtuvo de estudios previos. La topología del dendrograma fue similar a la obtenida con marcadores moleculares; las líneas transgénicas compartieron más del 89% de similitud con la línea parental, mientras que las variedades genéticamente cercanas compartieron el 75% y la variedad genéticamente distante, el 65% de similitud. Dado que los rasgos morfológicos revelaron mayor polimorfismo, su inclusión permitió diferenciar todas las líneas RH evaluadas. Al combinar los marcadores morfológicos y moleculares, las líneas RH 28 y 37 resultaron ser las más similares a la variedad parental RA 87-3 (más del 98% de similitud) (Figura 4).
Figura 4: Evaluación molecular y morfológica de líneas transgénicas. El dendrograma de los cuatro genotipos de caña de azúcar y de las líneas transgénicas se basó en el análisis de 339 alelos producto de nueve combinaciones de cebadores TRAP y 60 alelos producidos a partir de los nueve caracteres morfológicos obligatorios propuestos por la UPOV, utilizando el coeficiente de Jaccard y el método de agrupación UPGMA con InfoStat, presentado como distancia (1-S, S: similitud). Las líneas representaron similitudes de 0,97; 0.89; 0.75 y 0.65.
El presente trabajo muestra que la combinación de marcadores TRAP y marcadores morfológicos es un enfoque adecuado para evaluar, caracterizar y seleccionar líneas transgénicas con fenotipo y comportamiento similares al genotipo del receptor. Cabe destacar que esta es la primera vez que este tipo de caracterización integral, incluidos los rasgos de la UPOV, se realiza para evaluar una variedad de caña de azúcar transformada genéticamente.
Número de copias, estabilidad genética y expresión del transgén CP4 epsps
Con el fin de estudiar el número de inserciones del transgén CP4 epsps en el genoma de la planta, se realizaron análisis de Southern blot utilizando las enzimas de restricción MfeI y Bpu10I, las cuales poseen un sitio único de corte en el plásmido pEA1. Todos los eventos transformados RH evaluados en dos edades de cultivo, caña planta (P) y soca 1 (R1), contienen múltiples inserciones. Se comprobó la presencia de al menos siete insertos para las cuatro líneas transgénicas destacadas (15, 18, 28 y 37) cuando se utilizaron sondas para identificar el gen CP4 epsps (Figura 5a-b) y el gen nptII (datos no mostrados). Es interesante observar que todas las líneas transformadas (RH) examinadas contenían múltiples insertos transgénicos, algo que se ha informado previamente en caña de azúcar cuando se usa ADN circular para la transformación (Franks and Birch, 1991). Como este estudio solo analizó las líneas transformadas genéticamente que muestran alta resistencia al herbicida en el análisis de Southern blot, podría haber una correlación entre esta y los múltiples insertos transgénicos (Figura 5a-b)
Uno de los aspectos más importantes al evaluar una variedad transgénica para una posible liberación comercial es garantizar la herencia estable del transgén introducido. Esto es especialmente importante en un cultivo plurianual de propagación vegetativa, como la caña de azúcar, en la cual se debe verificar la expresión estable de(l) los transgén(es) en varios ciclos de cultivo (P, R1 y R2). Para esto, las líneas seleccionadas (28 y 37) se propagaron durante cuatro generaciones (una en invernadero (G) y tres en campo (P, R1 y R2). Para confirmar la transcripción estable del gen CP4 epsps a lo largo de las diferentes generaciones clonales se realizaron análisis RT-qPCR. El gen CP4 epsps se transcribió a niveles similares (20-25 veces) en las dos líneas transgénicas durante las tres edades de cultivo evaluadas (P, R1 y R2) (Figura 5c), los cuales fueron similares a los observados en plantas cultivadas en invernadero (Figura 2a). Estos resultados confirmaron la herencia estable del transgén observada en ensayos de Southern blot en edades de caña planta (P) (Figuras 5a-b) y soca 1 (R1) (datos no mostrados).
Figura 5: Caracterización molecular de líneas transgénicas. (a-b) Análisis del número de copias en líneas transgénicas en caña planta (P) por medio de Southern blot utilizando las enzimas Bpu10I y MfeI, respectivamente, con la sonda epsps. La variedad RA 87-3 no transformada (NT) se utilizó como control. (c) Cuantificación de la estabilidad de la expresión del gen epsps en tres generaciones de propagación (P, R1 y R2) mediante RT-qPCR. El promedio de la variación(n veces) de la expresión del gen epsps en dos líneas transgénicas (28, 37) en comparación con la planta no transformada RA 87-3 se calculó a partir de muestras por triplicado. P: planta de caña; R1: soca 1; R2: soca 2
Rendimiento agronómico de las seis líneas resistentes a herbicida
Los resultados obtenidos de los ANOVA realizados para los caracteres agronómicos mostraron diferencias estadísticamente significativas entre la variedad parental y las líneas transformadas 15, 18 y 22 para algunas de estas variables en los dos sitios de prueba. Además, las dos líneas destacadas (28 y 37) por presentar una mayor similitud genética con la variedad parental RA 87-3 (in vitro y propagación convencional) no evidenciaron diferencias estadísticamente significativas con esta en ninguno de los casos y para todas las características agronómicas estudiadas (Tabla 2).
Tabla 2. Caracteres agronómicos de seis líneas transgénicas resistentes a glifosato y de plantas control de la variedad parental RA 87-3multiplicadas diferencialmente (in vitro y propagadas en campo) evaluadas en las dos principales regiones productoras de Argentina y en dos campañas consecutivas de crecimiento: caña planta (P) y soca 1 (R1).
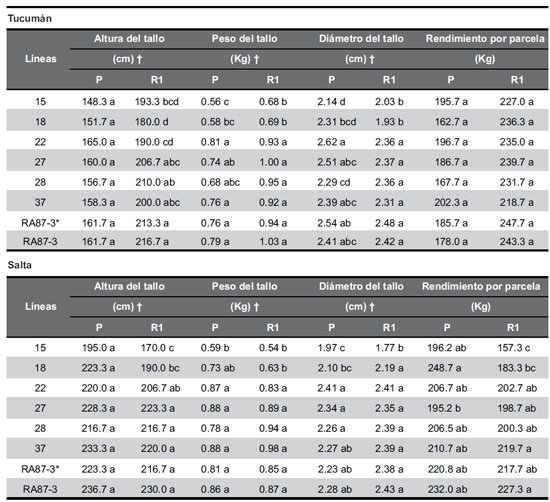
RA 87-3* propagada in vitro.
Datos obtenidos a partir de una muestra de diez tallos (n = 10) por réplica.
Análisis estadístico llevado a cabo por ANOVA y test LSD de Fisher.
Las medias seguidas por la misma letra dentro de una columna para cada región no son significativamente diferentes (p> 0,05).
Composición química de las líneas transgénicas evaluadas
Con respecto a la composición química, las líneas transgénicas no mostraron en general diferencias estadísticamente significativas con la variedad receptora en ninguna de las características estudiadas (proteínas, FDA, FDN y Sacarosa), independientemente de las áreas geográficas y edades de corte evaluadas. Solamente la línea 18 presentó una diferencia en el contenido total de proteína en el primer año de crecimiento (P) en la localidad de Colonia Santa Rosa (Salta) (Tabla 3). Los resultados de las pruebas en campo mostraron solo diferencias menores entre las seis líneas transgénicas y la variedad parental RA 87-3 cuando se analizaron diferentes características agronómicas y de composición
química (Tablas 2 y 3). Las determinaciones de sacarosa, proteína, fibra detergente ácida y fibra detergente neutra se encuentran dentro de los rangos de valores informados por Organization for Economic Co-operation and Development (OECD, 2011).
Tabla 3. Composición química de seis líneas transgénicas resistentes a glifosato y de plantas control de la variedad parental RA 87-3multiplicadas diferencialmente (in vitro y propagadas en campo) evaluadas en las dos principales regiones productoras en Argentina (Tucumán y Salta) y en dos campañas consecutivas de crecimiento: caña planta (P) y soca 1 (R1).
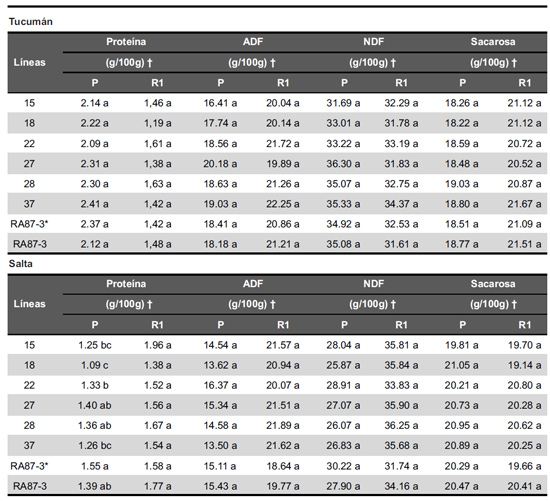
RA 87-3* propagada in vitro.
Datos obtenidos a partir de una muestra de diez tallos (n = 10) por réplica.
Análisis estadístico llevado a cabo por ANOVA y test LSD de Fisher.
Las medias seguidas por la misma letra dentro de una columna para cada región no son significativamente diferentes (p> 0.05).
Se han informado resultados similares en estudios en campo en los cuales se demostró que es posible recuperar completamente el fenotipo parental en eventos de caña de azúcar transgénica con características agronómicas e industriales comparables (Leibbrandt and Snyman, 2003; Gilbert et al., 2005). Estos reportes, junto con los resultados encontrados en este estudio, muestran la utilidad de la ingeniería genética en el mejoramiento de la caña de azúcar.
Los resultados obtenidos demuestran el potencial de la transformación genética como una herramienta efectiva y complementaria a los enfoques clásicos de mejora genética para introducir nuevos caracteres en el complejo genoma de la caña de azúcar. Además, las evaluaciones fenotípicas, morfológicas, genéticas, agronómicas y químicas permitieron la selección de dos líneas RH (28 y 37) casi idénticas a la variedad parental, lo cual fue evidenciado por los análisis de Southern blot que mostraron que estas se originaron a partir de un mismo evento de transformación (Fig. 5a-b). El hecho de que estas dos líneas fueron seleccionadas en forma independiente garantiza la reproducibilidad y confiabilidad de nuestros estudios.
En síntesis, a partir de los resultados obtenidos en los ensayos descriptos anteriormente se reunieron las evidencias científicas que permitieron seleccionar el evento transgénico candidato para llevar a cabo la desregulación comercial, conforme a las normativas vigentes en el país.
AGRADECIMIENTOS
Agradecemos a la Estación Experimental Agroindustrial Obispo Colombres (EEAOC) y al Consejo Nacional de Investigaciones Científicas y Técnicas (CONICET) por el apoyo financiero de los estudios realizados en este proyecto.
BIBLIOGRAFÍA CITADA
1.Aljanabi, S.; L. Forget and A. Dookun. 1999. An improve and rapid protocol for the isolation of polysaccharide and polyphenol free sugarcane DNA. Plant Molecular Biology Reporter 17: 281. [ Links ]
2.Altpeter, F.; N. Baisakh; R. Beachy; R. Bock; T. Capell; P. Christou; H. Daniell; K. Datta; S. Datta and P. J. Dix. 2005. Particle bombardment and the genetic enhancement of crops: myths and realities. Molecular Breeding 15: 305-327. [ Links ]
3.Alwala, S.; A. Suman; J. A. Arro; J. C. Veremis and C. A. Kimbeng. 2006. Target region amplification polymorphism (TRAP) for assessing genetic diversity in sugarcane germoplasm collections. Crop Science 46: 448-455. [ Links ]
4.ANKOM. 2006a. Acid detergent fiber in feeds filter bag technique. [En línea] Disponible en http://www.ssco.com.tw/Ankom/PDF_file/ADF%20Method%20A200pdf. [ Links ]
5.ANKOM. 2006b. Neutral detergent fiber in feeds filter bag technique. [En línea] Disponible en http://www.ssco.com.tw/Ankom/PDF_file/NDF%20Method%20A200.pdf. [ Links ]
6.AOAC. 2009. Single laboratory validation acceptance criteria (chemistry methods). [En línea] Disponible en http://www.aoac.org/dietsupp6/Dietary-Supplementweb- site/SLV_criteria.pdf. [ Links ]
7.Arencibia, A.; E. Carmona; M. Cornide; E. Menéndez and P. Molina. 2000. Transgenic sugarcane (Saccharum spp.). In: Bajaj, Y. P. S. (eds.) Transgenic Crops I. Springer-Berlag Berlin Heidelberg 2000, pp 188-206. [ Links ]
8.Arencibia, A. D.; E. R.Carmona; M. T. Cornide; S. Castiglione; J. O'Relly; A. Chinea; P. Oramas and F. Sala. 1999. Somaclonal variation in insect-resistant transgenic sugarcane (Saccharum hybrid) plants produced by cell electroporation. Transgenic Research 8 (5): 349-360.
9.Basnayake, S. W.; T. C. Morgan; L. Wu and R. G. Birch. 2012. Field performance of transgenic sugarcane expressing isomaltulose synthase. Plant biotechnology journal 10 (2): 217-225. [ Links ]
10.Bower, R.; A. R. Elliott; B. A. Potier and R. G. Birch. 1996. High-efficiency, microprojectile-mediated cotransformation of sugarcane, using visible or selectable markers. Molecular Breeding 2 (3): 239- 249. [ Links ]
11.Di Rienzo, J.; F. Casanoves; M. Balzarini; L. Gonzalez; M. Tablada y C. Robledo. 2009. InfoStat. Grupo InfoStat, FCA, Universidad Nacional de Córdoba, Argentina. [ Links ]
12.Dillon, S. L.; F. M. Shapter; R. J. Henry; G. Cordeiro; L. Izquierdo and L. S. Lee. 2007. Domestication to crop improvement: genetic resources for Sorghum and Saccharum (Andropogoneae). Annals of botany 100 (5): 975-989. [ Links ]
13.Enríquez-Obregón, G. A.; R. I. Vázquez-Padrón; D. L. Prieto-Samsonov; A. Gustavo and G. Selman- Housein. 1998. Herbicide-resistant sugarcane (Saccharum officinarum L.) plants by Agrobacteriummediated transformation. Planta 206 (1): 20-27. [ Links ]
14.Falco, M.; A. T. Neto and E. Ulian. 2000. Transformation and expression of a gene for herbicide resistance in a Brazilian sugarcane. Plant Cell Reports 19 (12): 1188-1194. [ Links ]
15.Finer, J. J.; P. Vain; M. W. Jones and M. D. McMullen. 1992. Development of the particle inflow gun for DNA delivery to plant cells. Plant Cell Reports 11 (7): 323- 328. [ Links ]
16.Franks, T. and R. Birch. 1991. Gene Transfer Into Intact Sugarcane Cells Using Microprojectile Bombardment. Functional Plant Biology 18 (5): 471-480. [En línea] doi:http://dx.doi.org/10.1071 /PP9910471. [ Links ]
17.Gallo-Meagher, M. and J. Irvine. 1996. Herbicide resistant transgenic sugarcane plants containing the bar gene. Crop Science 36 (5): 1367-1374. [ Links ]
18.Gilbert, R.; M. Gallo-Meagher; J. Comstock; J. Miller; M. Jain and A. Abouzid. 2005. Agronomic evaluation of sugarcane lines transformed for resistance to strain E. Crop science 45 (5): 2060- 2067. [ Links ]
19.Gilbert, R.; N. Glynn; J. Comstock and M. Davis. 2009. Agronomic performance and genetic characterization of sugarcane transformed for resistance to sugarcane yellow leaf virus. Field crops research 111 (1): 39-46. [ Links ]
20.Green, J. M. 2009a. Evolution of glyphosate-resistant crop technology. Weed Science 57 (1): 108-117. [ Links ]
21.Green, J. M. 2009b. Review of glyphosate and ALSinhibiting herbicide crop resistance and resistant weed management. [ Links ] Weed Tchnology.
22.Grivet, L.; J. Glaszmann and P. Arruda. 2001. Sequence polymorphism from EST data in sugarcane: a fine analysis of 6-phosphogluconate dehydrogenase genes. Genetics and Molecular Biology 24 (1-4): 161- 167. [ Links ]
23.Gupta, O. 1960. Weed control in sugarcane. PANS (C) 14 (2): 154. [ Links ]
24.Hoy, J. W.; K. P. Bischoff; S. B. Milligan and K. A. Gravois. 2003. Effect of tissue culture explant source on sugarcane yield components. Euphytica 129 (2): 237-240. [ Links ]
25.Ibrahim, A. 1984. Weed competition and control in sugarcane. Weed Research 24 (4): 227-231. [ Links ]
26.ISAAA. 2017. Global Status of Commercialized Biotech/GM Crops in 2017: Biotech Crop Adoption Surges as Economic Benefits Accumulate in 22 Years. ISAAA Brief No. 53. [En línea] Disponible en www.isaaa.org/resources/ publications/Briefs/53/ (consultado: 17 octubre 2018). [ Links ]
27.Jackson, M. A.; D. J. Anderson and R. G. Birch. 2013. Comparison of Agrobacterium and particle bombardment using whole plasmid or minimal cassette for production of high-expressing, low-copy transgenic plants. Transgenic Res 22 (1): 143-151. [En línea] doi:10.1007/s11248-012-9639-6 [ Links ]
28.James, C. 2013. Global Status of Commercialized Biotech/GM Crops: 2013. Brief 46. [En línea] Disponible en www.isaaa.org/resources/ publications/Briefs/46/. (consultado: 8 junio 2018). INTERNATIONAL SERVICE FOR THE ACQUISITION OF AGRI-BIOTECH APPLICATIONS (ISAAA), Manila. [ Links ]
29.Joyce, P.; S. Hermann; A. O'Connell; Q. Dinh; L. Shumbe and P. Lakshmanan. 2014. Field performance of transgenic sugarcane produced using Agrobacterium and biolistics methods. Plant biotechnology journal 12: 411–424. [En línea] doi: 10.1111. [ Links ]
30.Leibbrandt, N. and S. Snyman. 2001. Initial field testing of transgenic glufosinate ammonium-resistant sugarcane. In: Proc S Afr Sug Technol Ass, 2001, pp. 108-111. [ Links ]
31.Leibbrandt, N. B. and S. J. Snyman. 2003. Stability of gene expression and agronomic performance of a transgenic herbicide-resistant sugarcane line in South Africa. Crop Science 43 (2): 671-677. [ Links ]
32.Li, G. and C. F. Quiros. 2001. Sequence-related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction: its application to mapping and gene tagging in Brassica. Theoretical and Applied Genetics 103 (2-3): 455-461. [ Links ]
33.Lichtenthaler, H. K. 1987. [34] Chlorophylls and carotenoids: Pigments of photosynthetic biomembranes. Methods in enzymology 148: 350- 382. [ Links ]
34.Livak, K. J. and T. D. Schmittgen. 2001. Analysis of Relative Gene Expression Data Using Real-Time Quantitative PCR and the 2− __CT Method. Methods 25 (4): 402-408. [ Links ]
35.Mano, A. 2017. Brazil approves world`s first commercial GM sugarcane: developer CTC [En línea] Disponible en www.reuters.com/article/us-brazil-sugargmoidUSKBN18Z2Q6 (consultado: 10 mayo 2018). REUTERS, Sao Paulo. [ Links ]
36.Noguera, A.; N. Paz; E. Díaz; F. Perera; M. Sepúlveda Tusek; M. Filippone y A. Castagnaro. 2010. Proyecto Vitroplantas: La producción de caña semilla de alta calidad comienza en el laboratorio Publicación Especial 40 Proyecto Vitroplantas: producción de caña semilla de alta calidad en la Estación Experimental Agroindustrial Obispo Colombres (EEAOC). [ Links ]
37.OECD. 2011. Consensus Document on Compositional Considerations for New Varieties of SUGARCANE (Saccharum ssp. Hybrids): Key Food and Feed Nutrients, Anti-nutrients and Toxicants [En línea] Disponible en www.oecd.org/science/biot rack/48962816.pdf (consultado: 17 agosto 2015). OECD, Paris. [ Links ]
38.Olea, I.; S. Sabaté y H. Vinciguerra. 2009. Manejo de malezas. En: Romero, E. R.; P. A. Digonzelli y J. Scandaliaris (Eds.): Manual del cañero. EEAOC. 2009. [ Links ]
39.Perera, M.; M. Arias; D. Costilla; A. Luque; M. García; C. D. Romero; J. Racedo; S. Ostengo; M. Filippone and M. Cuenya. 2012. Genetic diversity assessment and genotype identification in sugarcane based on DNA markers and morphological traits. Euphytica 185 (3): 491-510. [ Links ]
40.Rapulana, T. and G. Bouwer. 2013. Toxicity to Eldana saccharina of a recombinant Gluconacetobacter diazotrophicus strain carrying a truncated Bacillus thuringiensis cry1Ac gene. African Journal of Microbiology Research 7 (14): 1207-1214. [ Links ]
41.Sala, F.; A. Arencibia; S. Castiglione; P. Christou; Y. Zheng and Y. Han. 1999. Molecular and field analysis of somaclonal variation in transgenic plants. In: Plant Biotechnology and In Vitro Biology in the 21st Century. Springer, pp. 259-262. [ Links ]
42.Sneath, P. H. and R. R. Sokal. 1973a. Numerical taxonomy. The principles and practice of numerical classification. [ Links ]
43.Sneath, P. H. and R. R. Sokal. 1973b. Numerical Taxonomy. Theory and Application of Genetics 93: 613-617. [ Links ]
44.Taylor, P.; T. Fraser; H-L. Ko and R. Henry. 1995. RAPD analysis of sugarcane during tissue culture. In: Current issues in plant molecular and cellular biology. Springer, pp. 241-246. [ Links ]
45.UPOV. 2005. Draft Test Guidelines for Sugarcane. [En línea] Disponible en http://www.upov.int/edocs/tgdocs/ es/tg186.pdf. [ Links ]
46.Vellicce, G.; A. Noguera; M. Filippone y A. Castagnaro. 2011. Implementación de un sistema de biobalística para la transformación genética de plantas en la Estación Experimental Agroindustrial Obispo Colombres (EEAOC). Avance Agroind. 32 (1): 31-34. [ Links ]
47.Vickers, J.; C. Grof; G. Bonnett; P. Jackson and T. Morgan. 2005. Effects of tissue culture, biolistic transformation, and introduction of PPO and SPS gene constructs on performance of sugarcane clones in the field. Crop and Pasture Science 56 (1): 57-68. [ Links ]



















