Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Phyton (Buenos Aires)
versión On-line ISSN 1851-5657
Phyton (B. Aires) vol.80 no.1 Vicente López ene./jun. 2011
ARTÍCULOS ORIGINALES
Variabilidad genética de aislamientos no-típicos de Azospirillum brasilense por análisis PCR-RFLP del ADN 16S ribosomal
Genetic variability of non-typical Azospirillum brasilense isolates by PCR-RFLP analysis of 16S ribosomal DNA
Pérez Castañeda LM1, MA Cruz Hernández1, A Mendoza Herrera1
1 Centro de Biotecnología Genómica-Instituto Politécnico Nacional, Laboratorio de Biotecnología Vegetal, Boulevard del Maestro s/n Esq. Elías Piña, Col. Narciso Mendoza, C.P. 88730 Cd. Reynosa, Tamaulipas, México. Tel. y Fax: 01(899) 925-1656 y 925-3996.
Address Correspondence to: Dr. Alberto Mendoza Herrera, e-mail: amendozah@ipn.mx
Recibido - Received 10.XI.2010.
Aceptado - Accepted 25.XI.2010.
Resumen. Diversos estudios han demostrado la asociación simbiótica de Azospirillum brasilense con diferentes gramíneas de importancia alimenticia. Así mismo, se ha encontrado una gran variabilidad entre las cepas aisladas, lo cual es consistente con la diversidad genética que Azospirillum ha mostrado en aislamientos de caña de azúcar. Sin embargo, se desconoce si esto sucede en aislados obtenidos de maíz y sorgo. Además, se ha reportado que el mayor número de aislamientos se han obtenido de regiones tropicales donde el agua no es un factor limitante. Considerando la gran diversidad que Azospirillum presenta, en este trabajo se analizó la variabilidad genética de 102 aislados de Azospirillum brasilense, provenientes de maíz y sorgo. Los trabajos, que se efectuaron en zonas áridas del Noreste de México, también evaluaron su relación filogenética con otras cepas de la misma especie provenientes de regiones tropicales. Los aislamientos se realizaron de suelos con regímenes de cultivo distinto; cultivos en secano o bajo riego. Se observó una alta variabilidad genética en los aislamientos, obteniéndose 14 bandas polimórficas en la población estudiada con tamaños de entre 169 y 788 pb. El análisis RFLP del gen 16S ADNr mostró la formación de dos grupos principales de aislamientos, uno comprendió los provenientes de cultivos en secano o temporal y el segundo los de cultivos bajo riego. Al mismo tiempo, no hubo diferencias entre los aislamientos provenientes de maíz o sorgo. El análisis filogenético en base a las secuencias del 16S ADNr de cinco aislados representativos de la diversidad encontrada, mostró la formación de dos clados, uno de ellos conteniendo solo tres aislados. El otro clado con los dos aislados restantes incluyó las cepas tipo. Así mismo, la separación de los aislamientos en los dos grupos se correspondió con el tipo de régimen hídrico del cual provinieron: temporal y riego. Esto sugiere que las diferencias observadas en las agrupaciones podría ser un reflejo de diversos fenómenos de adaptación relacionados con el régimen hídrico bajo el cual crece el cultivo, y no con la especie de gramínea.
Palabras clave: Azospirillum brasilense; Variabilidad genética; RFLP; 16S ADNr.
Abstract. Various studies have shown the symbiotic association of Azospirillum brasilense with different grasses of nutritional importance. A high variability among the isolated strains has been observed. This is consistent with the genetic diversity that Azospirillum has shown in sugarcane isolates. However, if this is also the case in maize and sorghum isolates is unknown. In addition, it has also been reported that most of the isolates come from tropical regions where water is not limiting for maize and sorghum production. We analyzed the genetic variability of Azospirillum brasilense isolates obtained from arid zones of Northeast Mexico in maize and sorghum. Also, we characterized the phylogenetic relationship of five isolates selected from their RFLP profiles with four Azospirillum species, as well as the phylogenetically related bacteria Rhizobium etli. The genetic variability was analyzed by PCR-RFLP of the gene 16S ADNr. The isolates were obtained from crops exposed to either rainfed or irrigated conditions. A high genetic variability was observed on the isolates, obtaining 14 different polymorphic bands in the study population with sizes ranging from 169 to 788 bp. RFLP analysis of the gene 16S ADNr showed the formation of two main clades of isolates: one group contained isolates coming from rainfed conditions, the other clade was composed by isolates from crops exposed to irrigation, and included the wild type strains. Results also showed that there were no differences between isolates either from maize or sorghum. The phylogenetic analysis based on the 16S ADNr sequences of five selected isolates, representative of the found diversity, showed the formation of two main clades, one of them containing only three isolates. The other clade with the two remaining isolates included the wild type strains. The separation of the isolates in both clades corresponded to the moisture regime they came from: either rainfed of irrigated conditions. This suggests that the differences observed in the groupings could be the result of diverse adaptation phenomena related to the water regime the crop grows in, and not to the grass species.
Keywords: Azospirillum brasilense; Genetic variability; RFLP; 16S rDNA.
INTRODUCCIÓN
Azospirillum brasilense es una bacteria gram-negativa ampliamente estudiada (Döbereiner y Day, 1976b) por su alta capacidad para fijar nitrógeno y producir fitohormonas. Existe una amplia gama de variantes en esta especie que van de aeróbicas a microaerofílicas obligadas, mostrando capacidad de vivir bajo diferentes concentraciones de oxígeno (Vande Broek et al., 1996). Azospirillum brasilense se encuentra en el suelo asociada a la raíz de plantas, principalmente de la familia de las Poáceas [ej.: caña de azúcar (Döbereiner y Day, 1976a), mijo (Umali-García et al., 1980), arroz (Saleena et al., 2002), trigo (Jain y Patriquin, 1984), maíz (Gafny et al., 1986), sorgo, avena y forrajes como pastos y alfalfa (Shawky, 1989)]. Estas poblaciones de bacterias juegan un papel destacado en la producción agrícola ya que son promotoras del crecimiento de las plantas (PGPR), efecto mediado por la producción de fitohormonas, principalmente el ácido indol acético (AIA) que afecta positivamente al crecimiento de la planta (Zimmer et al., 1998). Esto es debido al aumento en el número de raíces laterales y pelos radicales (Vande y Vanderleyden, 1995). Se ha reportado que el mayor número de aislamientos de Azospirillum ha sido obtenido de regiones tropicales, aunque también se la encuentra en regiones templadas, frías y desérticas (Döbereiner et al., 1976; Tyler et al., 1979; Haatela et al., 1981; De Coninck et al., 1988). La mayoría de los estudios genéticos realizados se han enfocado en la cepa tipo, denominada Sp7, aislada de caña de azúcar en Brasil (Levanovy y Bashan, 1991).
Aun cuando el género Azospirillum se encuentra ampliamente distribuído, se localiza principalmente en regiones tropicales. El pH del suelo es una característica importante que determina la presencia del genero Azospirillum, encontrándose con mayor abundancia en suelos cercanos a pH=7, aunque también ha sido encontrada esporádicamente en suelos con un pH menor a 5 (Caballero-Mellado, 2002). Las especies más abundantes en suelos neutros son Azospirillum lipoferum y Azospirillum brasilense (Larralde-Corona, 2004). Los Azospirila fuera de estos rangos podrían considerarse como cepas no-típicas, adaptadas a climas extremos, suelos áridos y alcalinos (pH 8,0). Por otro lado, el porcentaje de arcilla, contenido de materia orgánica, capacidad de retención de agua y contenido de nitrógeno en dichas condiciones ambientales afectan la supervivencia de A. brasilense. No obstante, la supervivencia de A. brasilense, en la rizósfera, es independientemente de la aridez del suelo (Bashan y de Bashan, 2010).
Los cultivos principales en el Noreste de México son el sorgo y el maíz, donde los fertilizantes químicos se han usado masivamente y han afectado a las poblaciones nativas de bacterias benéficas disminuyendo su diversidad (Saleena et al., 2002). Actualmente, el uso de biofertilizantes ha favorecido el incremento en el rendimiento de los cultivos agrícolas en los países desarrollados, generando alta productividad y propiciando la conservación de los suelos (Rangarajan et al., 2001). Aun cuando el uso de Azospirillum como bioestimulante del crecimiento de las plantas no se ha generalizado mundialmente, la aplicación de esta bacteria en diferentes cultivos ha sido ampliamente utilizada en México. En el ciclo agrícola primavera- verano (PV) del año 1999, la Secretaría de Agricultura, a través de su Instituto Nacional de Investigaciones Forestales Agrícolas y Pecuarias (INIFAP) y de dos Centros de Investigación nacionales, llevaron a cabo la inoculación de cerca de 450.000 hectáreas de maíz y 150.000 hectáreas de sorgo, cebada y trigo, empleando cepas de A. brasilense seleccionadas por su capacidad para promover el crecimiento de las plantas. En este estudio se encontró un incremento, según la región, de entre el 15 al 25% (Caballero-Mellado, 2002). En el estado de Tamaulipas, México, García-Olivares et al. (2007) también observaron el efecto benefico del uso de cepas de Azospirillum brasilense en cultivos de maíz y sorgo de zonas áridas.
Rodríguez-Salazar et al. (2009), utilizando biofertilizantes o bioestimulantes lograron incrementar significativamente la tolerancia a sequía y la biomasa de plantas de maíz mediante la inoculación con una cepa de A. brasilense, modificada genéticamente en experimentos de invernadero. Además del genero Azospirillum, se han detectado actividades de interés agrobiotecnológico en otras bacterias: Burkholderia unamae, B. xenovorans, B. tropica y B. silvatlantica (Caballero-Mellado et al., 2007), Azotobacter vinelandii (Galindo et al., 2006; Santana et al., 2002), Rhizobium (Singleton, 2004), por mencionar solo algunas.
Una manera de lograr que el uso de biofertilizantes en el noreste de Tamaulipas sea exitoso, seria mediante el análisis de la estructura genética, distribución y variación entre las poblaciones nativas de Azospirillum. Se podría determinar asi cuales podrían ser más eficientes para su utilización y aprovechamiento en los sistemas agrícolas. En otros ecosistemas se han empleado diferentes metodologías que permitieron caracterizar dichas poblaciones, tales como pruebas morfológicas, bioquímicas, fisiológicas, inmunológicas y actualmente las de mayor éxito y precisión son los análisis genéticos. A la fecha se tienen identificadas 15 especies: A. lipoferum y A. brasilense (Tarrand et al., 1978; son las más estudiadas); A. amazonense (Magalhães et al., 1983), A. halopraeferens (Reinhold et a.l, 1987), A. irakense (Khammas et a.l, 1989), A. largimobile (Sly y Stackebrand, 1999), y las recientemente descritas A. doebereinerae (Eckert et al., 2001), A. oryzae (Xie et al., 2005), A. melinis (Peng et al., 2006), A. canadense (Mehnaz et al., 2007), A. zeae (Mehnaz et al., 2007), A. rugosum (Young et al, 2008), Azospirillum picis (Lin et al., 2009), Azospirillum palatum (Zhou et al., 2009) y Azospirillum palatum (Yu Zhou et al., 2009). Con este propósito, se ha recurrido a amplificar los genes 16S ADNr y digerir los amplicones con la enzima AluI, la cual genera fragmentos de tamaños específicos para cada especie, distinguibles en geles de agarosa (Caballero-Mellado, 2002). Además, los tipos y números de organismos en comunidades naturales también pueden ser examinados, secuenciando los genes ribosomales a partir del ADN aislado de células en su ambiente ordinario (Pace, 1996). Los objetivos del presente trabajo fueron (1) analizar la variabilidad genética del gen 16S ADNr en 102 aislamientos de A. brasilense nativos de las zonas áridas del Noreste de México mediante el empleo de PCR-RFLP (Polimorfismos en la Restricción de los Fragmentos de Restricción) y (2) determinar su relación genética con base en la variabilidad de la secuencia del mismo gen en 7 aislamientos representativos: 2 cepas tipo (SP7 y CD1), 4 especies de Azospirillum y una especie relacionada (R. etli), reportadas en el banco de genes (NCBI).
MATERIALES Y MÉTODOS
Material biológico. Se analizaron 102 aislados de A. brasilense, nativos de zonas áridas del Noreste de Tamaulipas, obtenidos a partir de la rizósfera de plantas de maíz y sorgo cultivadas en la región (Tabla 1). Para su aislamiento y purificación se utilizaron los medios específicos Rojo Congo y NFB, respectivamente (Döbereiner et al., 1976). Se incluyeron como testigos las cepas de referencia Sp7 (proveniente de Brasil) y Cd1, derivada de la Sp7 (Itzigsohn y Okon, 1995).
Tabla 1. Procedencia de los aislamientos de A. brasilense utilizados en este estudio.
Table 1. Origin of the A. brasilense isolates.

Extracción de DNA. Los aislamientos crecieron en medio de cultivo TYG líquido suplementado con ampicilina (100 μg/mL), incubando por 36 h a 30 °C a 200 rpm. La extracción de ADN genómico se realizó de acuerdo al método propuesto por Sambrook et al. (1989).
Amplificación por PCR. Para amplificar el gen 16S ADNr se emplearon los oligonucleótidos universales 27f (5'-GAGAGTTTGATCCTGGCTCAG-3') y 1495r (5'-CTACGGCTACCTTGTTACGA-3'), descritos por Grifoni et al. (1995).
Análisis RFLP y análisis bioinformático. El análisis RFLP del gen 16S ADNr se llevó a cabo con la endonucleasa AluI que reconoce la secuencia nucleotídica AGCT, bajo las condiciones descritas por Han y New (1998); los perfiles RFLP obtenidos se analizaron mediante el algoritmo de Neighbor-Joining (Sokal y Sneath, 1963) y el agrupamiento UPGMA (Sneath y Sokal, 1973) del programa Neighbor del paquete estadístico Phyllip (Felsenstein, 1994) para determinar la estructura de la población; la variabilidad genética se estimó mediante el índice de diversidad de Simpson (1949). A partir de dicho análisis se seleccionaron cinco aislamientos (38, 45, 55, 176 y 181) que representaron la población para su clonación, secuenciación enzimática y alineamiento múltiple de las secuencias completas del gen 16S ADNr. Los productos de PCR se ligaron al vector pCR-XL-TOPO (Shuman, 1994); las clonas positivas se secuenciaron en un equipo de secuenciación automática LI-COR (Lincoln, NE, EUA). Las secuencias obtenidas se compararon por alineamiento con el programa BLAST contra las secuencias existentes en el banco de genes internacional (NCBI) para verificar su identidad y se sometieron a alineamiento múltiple con el programa Clustal X ver. 1.81 (Thompson et al., 1997). Para esto último se usó el método de análisis por remuestreo "bootstrap" para 1000 iteracciones con secuencias del mismo gen de otras especies de Azospirillum y Rhizobium etli (Tabla 2), como una cepa filogenéticamente cercana y de referencia, obtenidas del mismo banco de genes, obteniéndose el árbol filogenético correspondiente (Felsenstein, 1994).
Tabla 2. Secuencias nucleotídicas del gen 16S ADNr empleadas para el alineamiento múltiple intra especie, interespecies e intergéneros.
Table 2. Nucleotide sequences of 16S rDNA used for multiple alignment among species and genus.

RESULTADOS
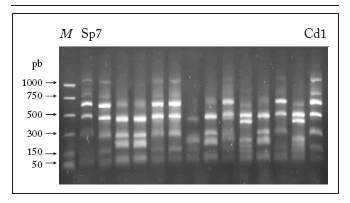
El análisis PCR-RFLP del gen 16S ADNr de los 102 aislamientos y las cepas de referencia Sp7 y Cd1 de A. brasilense detectó amplia variabilidad genética. En la Fig. 1 se puede observar un ejemplo de los perfiles observados con algunos aislados.
La digestión del fragmento amplificado con la enzima de restricción AluI permitió obtener en total 14 bandas amplificadas 100% polimórficas con los siguientes pesos moleculares: 169, 216, 247, 263, 300, 347, 428, 478, 500, 558, 580, 664, 700, 788 pb. Además, se identificaron 3 grupos de patrones de restricción principales; uno de ellos se presentó con mayor frecuencia en toda la población. Los grupos se consideraron representativos para A. brasilense; este perfil coincidió con el perfil que mostró la cepa Sp7 (Fig. 1) que presentó cuatro bandas características (664, 500 300 y 169 pb). Otro tipo de perfil que presentó cinco bandas (500, 428, 300, 247 y 169 pb) fue el preponderante (23 individuos de la población), y el tercer grupo con algunas variaciones que se vieron reflejadas en el Dendrograma. En la Fig. 2 se puede observar el dendrograma obtenido a partir de este análisis, el cual separó al total de los aislamientos en dos grupos principales: el primer grupo (A), que incluyó los aislados provenientes de la rizósfera de maíz y sorgo cultivados bajo condiciones de riego en la región de Díaz Ordaz, Tam., así como también las cepas de referencia Sp7 y Cd1: este grupo mostró menor variabilidad genética entre ellas (IDs=0,7641). El segundo grupo (B) incluyó aislamientos de maíz y sorgo, pero éstos son cultivos bajo condiciones de secano de las regiones de Río Bravo y Valadeces, Tam. Los aislamientos de A. brasilense de esta condición mostraron una mayor variabilidad entre ellos (IDs=0,8581). Sin embargo, existió similitud entre cepas tanto del grupo A como del grupo B (uniformidad de 0,72 y 0,81 respectivamente).
Fig. 1. Patrones de restricción PCR-RFLPs del 16S ADNr de aislamientos de A. brasilense nativos de zonas áridas de Tamaulipas y de las cepas de referencia Sp7 y Cd1 obtenidos con AluI.
Fig. 1. PCR-RFLP profiles of 16S rDNA from native A. brasilense isolated from the arid region of Tamaulipas state and wild type strains SP7 and Cd1 obtained by AluI restriction enzyme.
Fig. 2. Dendrograma de similitud de aislamientos de A. brasilense nativos de zonas áridas de Tamaulipas y cepas de referencia Sp7 y Cd1 con base en patrones de restricción RFLP-PCR del gen 16S ADNr obtenido por el método de Neighbor-Joining. Los aislamientos provienen de cultivos de maíz y sorgo de las localidades (A) Díaz Ordaz, Tam., (riego) y (B) Valadeces, Tam., (temporal o secano).
Fig. 2. Dendrogram of isolates similarity of A. brasilense native from arid zones of Tamaulipas and wild type strains Sp7 and Cd1 according to RFLP-PCR 16S profiles obtained by Neighbor-Joining. Isolates come from Maize and Sorghum from: (A) Díaz Ordaz, Tam., (with irrigation) and (B), Valadeces, Tam., (rainfed).
El análisis filogenético en base a las secuencias del 16S ADNr (Fig. 3) mostró la formación de dos clados. La separación entre ambos clados fue confirmada al 100% por pruebas de remuestreo (bootstrap). Los aislados se separaron entre ambos clados, donde tres de ellos (38, 55, y 45) constituyen exclusivamente uno de ellos y los dos restantes (181 y 176) se agruparon en el clado que incluye las cepas tipo SP7 y CD1. Además, se encontraron estrechamente relacionados con A. lipoferum y A. halopraeferans. Mientras que A. amazonense y A. irakense estuvieron agrupadas con R. etli.
Fig. 3. Árbol filogenético del alineamiento múltiple de secuencias completas del gen 16S ADNr de aislamientos nativos del noreste de México seleccionados (38, 45, 55, 176 y 181) y cepas de referencia Sp7 y Cd1 de A. brasilense, con otras especies del género Azospirillum y R. etli, como cepa de referencia. Los números en cada nodo corresponden al número de repeticiones que se generan en las agrupaciones.
Fig. 3. Phylogenetic tree of multiple alignment of complete sequences of gene 16S rDNA of selected native isolates (38, 45, 55, 176 y 181) from Northeast Mexico, and comparison to wild type strains Sp7 and Cd1 and other Azospirillum strains and R. etli as a related strain. Numbers represent boostraps over 1000 times.
De la misma forma, la separación de los aislamientos en los dos grupos correspondió con el tipo de régimen de donde provienen, de secano y riego respectivamente. Esto sugiere que las diferencias observadas en las agrupaciones podría ser el resultado de diversos fenómenos de adaptación a las condiciones de humedad de la cual provienen los cultivos.
DISCUSIÓN
La variabilidad genética encontrada en el análisis del gen 16S ADNr de los aislamientos nativos de zonas áridas es un indicativo de que dichas bacterias han tenido un período de evolución significativo. Este período les ha permitido adaptarse a zonas como la del noreste de Tamaulipas donde el pH del suelo es alcalino (pH: 8,0), el cual no es el valor óptimo para A. brasilense (Döbereiner, 1992; Caballero-Mellado, 2002). Estos resultados indican que los mismos se deben considerar como no-típicos; así lo demuestran las poblaciones reportadas por Mendoza et al. (2001) y los resultados mencionados por Larralde-Corona (2004). Por otro lado, los perfiles de restricción observados en este estudio confirman la alta variabilidad genética a nivel del 16S ADNr (IDs=0,9123), y por lo tanto la flexibilidad de Azospirillum para adaptarse a diferentes nichos ecológicos o condiciones principalmente edáficas. Esta flexibilidad podría ser el resultado de la estructura de su genoma en multi replicones (Caballero-Mellado, 2002). Dicha variabilidad genética es la que determina su capacidad de adaptación a medios favorables o extremos (Saleena et al., 2002).
La planta huésped (maíz o sorgo) no tuvo impacto en la agrupación de estos aislamientos. Es decir, que independientemente de las condiciones edáficas, esta especie ya está bien adaptada a su huésped principal, llámese maíz o sorgo, sin distinción alguna. Sería importante determinar que tipo de estructura tendría una población mixta de aislamientos nativos de esta misma especie provenientes de diferentes tipos de huéspedes, por ejemplo de Poáceas y no Poáceas. En este trabajo los agrupamientos fueron claramente formados en base al estrés hídrico. Se resalta una mayor variabilidad al interior de las poblaciones de A. brasilense que fueron aisladas en cultivos de secano, es decir que año tras año estuvieron sujetos a estrés hídrico. En la Fig. 2 se muestra que el clado (A) formado por aislados provenientes de cultivos de maíz o sorgo (que año tras año estuvieron bajo un sistema de riego, es decir, sin estrés hídrico) mostró una menor variabilidad entre individuos al interior de dicho grupo. Además, las dos cepas de referencia, SP7 y CD1 se encontraron inmersas en este mismo grupo. En el segundo clado (B), que incluyó los aislados de la rizósfera de maíz o sorgo de la región de secano (Valadeces, Tam.), donde anualmente el estrés hídrico es una constante, se muestra una mayor variabilidad al interior de la población de los aislados.
La variabilidad genética encontrada entre los aislamientos de la colección de A. brasilense, podría deberse en cierto grado a la recombinación que ocurre en especies clonales por mecanismos parasexuales (Maynard-Smith et al., 1991), y entre bacterias relacionadas pertenecientes a la misma especie (Palleroni, 1997), dando esto origen a la diversidad intraespecífica. Con estos resultados podemos asumir que, para las condiciones edáficas consideradas en este estudio, y analizando el 16S ADN ribosomal, sería posible diferenciar las poblaciones de A. brasilense adaptadas al estrés hídrico, que año tras año puede ocurrir en el noreste de Tamaulipas. Por otro lado, el alineamiento múltiple de la secuencia completa del 16S ADNr de los cinco aislamientos seleccionados mostró claramente como tres de ellos (38, 45 y 55) presentaron suficientes diferencias nucleotídicas con respecto a los otros aislamientos de A. brasilense (181 y 176) y las cuatro especies de Azospirillum para formar un grupo independiente. Sin embargo, esto no quiere decir que se trate de otra especie, ya que dichas diferencias no son suficientes como para considerarlo así. Además, su caracterización biológica y bioquímica en curso asi lo compueban (datos no mostrados). Los aislamientos nativos de cada zona pueden desempeñarse mejor que las cepas introducidas en la promoción del crecimiento de cultivos debido a su superior adaptabilidad al ambiente, como lo constatan los resultados de Kapulnik et al. (1983). Estos autores inocularon tres cultivares de trigo con A. brasilense y cepas locales en diferentes áreas en Israel mejorando notablemente su crecimiento y producción.
La detección de poblaciones adaptadas al ecosistema árido del noreste de Tamaulipas y el conocimiento de su variabilidad podría permitir hacer una mejor selección de aislamientos nativos de A. brasilense, con mayor potencial para la elaboración de biofertilizantes que se podrían aplicar a gramíneas de la región y de otras zonas áridas.
AGRADECIMIENTOS
A la Coordinación General de Postgrado e Investigación (SIP-IPN) Proyectos Nos. 20031486 y 20040239. A la Comisión Nacional Forestal (CONAFOR 2002-01-5443 y FOMIX-TAM 74004) y al Programa Institucional de Formación de Investigadores (PIFI-IPN) por el apoyo económico otorgado. Al Dr. Netzahualcoyotl Mayek y la Dra. Patricia Larralde por su crítica revisión.
REFERENCIAS
1. Bashan, Y. y L.E. de-Bashan (2010). How the plant growth-promoting bacterium Azospirillum promotes plant growth. A critical assessment. Advances in Agronomy 108: 77-136. [ Links ]
2. Caballero-Mellado, J. (2002). El género Azospirillum En: Microbios en Línea, Martínez R.E. y Martínez R.J.C. (eds). México, D F. UNAM, http://www.biblioweb.dgsca.unam.mx/libros/microbios/Cap10/). [ Links ]
3. Caballero-Mellado, J., J. Onofre-Lemus, P. Estrada-de los Santos y L. Martínez-Aguilar (2007). The tomato rhizosphere, an environment rich in nitrogen-fixing Burkholderia species with capabilities of interest for agriculture and bioremediation. Applied Environmental Microbiology 73: 5308-5319. [ Links ]
4. De Coninck, K., S. Horemans, S. Randombage y K. Vlassak (1988). Occurrence and survival of Azospirillum spp. in temperate regions. Plant Soil 110: 213-218. [ Links ]
5. Döbereiner, J. (1992). The genera Azospirillum and Herbaspirillum. En: A. Balows, H.G. Trüper, M. Dworkin, W. Harder y K.-H. Schleifer (eds.). The Prokaryotes, a Handbook on the Biology of Bacteria: Ecophysiology, Isolation, Identification, Applications. Vol. III, Springer-Verlag. New York. pp: 2236-2253. [ Links ]
6. Döbereiner, J. y J.M. Day (1976a). Associative symbioses and free-living systems. En: Proceedings of the 1st International Symposium on Nitrogen Fixation. Newton WE, Nyman C. J. (eds.). Washington State University Press. Pullman, WA, USA. pp: 518-538. [ Links ]
7. Döbereiner, J. y J.M. Day (1976b). Associative symbioses in tropical grasses: Characterization of micro-organisms and dinitrogen-fixing sites. En: Proceedings of the 1st International Symposium on Nitrogen Fixation. Newton W.E., Nyman C.J. (eds.). Washington State University Press. Pullman, WA, USA. Vol 2. pp: 518-538. [ Links ]
8. Döbereiner, J., I.E. Marriel y M. Nery (1976). Ecological distribution of Spirillum lipoferum Beijerinck. Canadian Journal of Microbiology 22: 1464-1473. [ Links ]
9. Eckert, B., O.B. Weber, G. Kirchhof, A. Halbritter, M. Stoffels y A. Hartmann (2001). Azospirillum doebereinerae sp. nov., a nitrogen-fixing bacterium associated with the C4-grass Miscanthus. International Journal of Systematic and Evolutionary Microbiology 51: 17-26. [ Links ]
10. Felsenstein, J. (1994). PHYLIP V3.57 (phylogeny inference package). Department of Genetics, University of Washington, Seattle. [ Links ]
11. Gafny, R., Y. Okon, Y. Kapulnik y M. Fischer (1986). Adsorption of Azospirillum brasilense to corn roots. Soil Biology Biochemical 18: 69-75. [ Links ]
12. Galindo, T., J. Polania, J. Sanchez, N. Moreno, J. Vanegas y G. Holguin (2006). Microbial inoculants effects on growth promotion of Mangrove and Citrullus vulgaris San Andres Isla, Colombia. Acta Biológica Colombiana 11: 83-97. [ Links ]
13. Garcia-Olivares, J.G, V.R. Moreno-Medina, I.C. Rodríguez-Luna, A. Mendoza-Herrera y N. Mayek-Pérez (2007). Efecto de cepas de Azospirillum brasilense en el crecimiento y rendimiento de grano de Maíz. Revista Fitotecnia Mexicana 30: 305-310. [ Links ]
14. Grifoni, A., M Bazzicalupo, C. Di Serio, S. Fancelli y R. Fani (1995). Identification of Azospirillum strains by restriction fragment length polymorphism of the 16S ADNr and of the histidine operon. FEMS Microbiology Letters 127: 85-91. [ Links ]
15. Han, S.O. y P.B. New (1998). Variation in nitrogen fixing ability among natural isolates of Azospirillum. Microbial Ecology 36: 193-201. [ Links ]
16. Haahtela, K., T. Wartiovaara, V. Sundman y J. Skujins (1981). Root-associated N2 fixation (acetylene reduction) by Enterobacteriaceae and Azospirillum strains in cold-climate spodsols. Applied Environmental Microbiology 41: 203-206. [ Links ]
17. Itzigsohn, R. y Y. Okon (1995). The development of Azospirillum as a commercial inoculant for improving crop yields. Journal of Biotechnology 13: 415-424. [ Links ]
18. Jain, D.K. y D.G. Patriquin (1984). Root hair deformation, bacterial attachment, and plant growth in wheat-Azospirillum associations. Applied Environmental Microbiology 48: 1208-1213. [ Links ]
19. Khammas, K.M., E. Ageron, P.A.D. Grimont y P. Kaiser (1989). Azospirillum irakense sp. nov., a nitrogen-fixing bacterium associated with rice roots and rhizosphere soil. Research in Microbiology 140: 679-693. [ Links ]
20. Kapulnik, Y., S. Sarig, I. Nur y Y. Okon (1983). Effect of Azospirillum inoculation on yield of field-grown wheat. Canadian Journal of Microbiology 29: 895-899. [ Links ]
21. Larralde-Corona, C.P. (2004). Laboratorio de Biotecnología Industrial CBG-IPN. (Comunicación Personal). [ Links ]
22. Levanovy, H. y Y. Bashan (1991). Active attachment of Azospirillum brasilense to root surface of non-cereal plants and to sand particles. Plant Soil 137: 91-97. [ Links ]
23. Lin, S.Y., C.C. Young, H. Hupfer, C. Siering, A.B Arun, W.M Chen, W.A. Lai, F.T. Shen, P.D. Rekha y A.F. Yassin (2009). Azospirillum picis sp. nov., isolated from discarded tar. International Journal of Systematic and Evolutionary Microbiology 59: 761-765. [ Links ]
24. Magalhães, F.M., J.I. Baldani, S.M. Souto, J.R. Kuykendall y J. Döbereiner (1983). A new acid-tolerant Azospirillum species. Anais da Academia Brasileira de Ciencias 55: 417-430. [ Links ]
25. Maynard-Smith, J., C.G. Dowson y B.G. Spratt (1991). Localized sex in bacteria. Nature 349: 29-31. [ Links ]
26. Mehnaz, S., B. Weselowski y G. Lazarovits (2007). Azospirillum canadense sp. nov., a nitrogen-fixing bacterium isolated from corn rhizosphere. International Journal of Systematic and Evolutionary Microbiology 57: 620-624. [ Links ]
27. Mehnaz, S., B. Weselowski y G. Lazarovits (2007). Azospirillum zeae sp. nov., a diazotrophic bacterium isolated from rhizosphere soil of Zea mays. International Journal of Systematic and Evolutionary Microbiology 57: 2805-2809. [ Links ]
28. Mendoza, A., A. Cruz, B. Lugo y H. Barrera (2001). Symbiotic behavior of Azospirillum brasilense strain in a semi-arid zone. CIFN XX Anniversary 1: 172-174. [ Links ]
29. Pace, N.R. (1996). New perspective on the natural microbial world: Molecular Microbial Ecology. The American Society for Microbiology News 62: 463-470. [ Links ]
30. Palleroni, N.J. (1997). Procaryotic diversity and the importance of culturing. Antonie van Leeuwenhoek 72: 3-19. [ Links ]
31. Peng, G., H. Wang, G. Zhang, W. Hou, Y. Liu, E.T. Wang y Z. Tan (2006). Azospirillum melinis sp. nov., a group of diazotrophs isolated from tropical molasses grass. International Journal of Systematic and Evolutionary Microbiology 56: 1263-1271. [ Links ]
32. Rangarajan, S., P. Loganathan, L.M. Saleena y S. Nair (2001). Diversity of pseudomonads isolated from three different plant rhizospheres. Journal of Applied Microbiology 91: 742-749. [ Links ]
33. Reinhold, B., T. Hurek, I. Fendrik, B. Pot, M. Gillis, K. Kersters, S. Thielemans y J. de Ley (1987). Azospirillum halopraeferens sp. nov., a nitrogen-fixing organism associated with roots of Kallar grass [Leptochloa fusca (L.) Kunth]. International Journal of Systematic and Evolutionary Microbiology 37: 43-51. [ Links ]
34. Rodríguez-Salazar, J., R. Suárez, J. Caballero-Mellado y G. Iturriaga (2009). Trehalose accumulation in Azospirillum brasilense improves drought tolerance and biomass in maize plants. FEMS Microbiology Letters 296: 52-59. [ Links ]
35. Saleena, L.M., S. Rangarajan y S. Nair (2002). Diversity of Azospirillum strains isolated from rice plants grown in saline and nonsaline sites of coastal agricultural ecosystem. Microbial Ecology 44: 271-277. [ Links ]
36. Sambrook, J., E.F. Fritsch y T. Maniatis (1989). Molecular Cloning: a Laboratory Manual. 2nd Ed. Cold Spring Harbor Laboratory Press. Cold Spring Harbour, NY, USA. [ Links ]
37. Santana, M., C. Vasquez, M. Martinez y M. Franco (2002). Evaluación de cepas de Azotobacter spp y de bacterias solubilizadoras de fosfato (BFS), como bifertilizante mixto en cultivos de crisantemo (Chrysoanthemum morifolium var. Regal Suerte). Tesis de Microbiologia Industrial. Pontificia Universidad Javeriana. 24 p. [ Links ]
38. Shawky, B.T. (1989). Studies on the occurrence of asymbiotic nitrogen-fixing Azospirillum species in the soils and rhizosphere of some plants in Egypt. Zentralblatt fur Mikrobiologie 144: 581-594. [ Links ]
39. Shuman, S. (1994). Novel approach to molecular cloning and polynucleotide synthesis using Vaccinia DNA topoisomerase. The Journal of Biological Chemistry 269: 32678-32684. [ Links ]
40. Singleton, P. (2004). Bacterias en Biología, Biotecnología y Medicina, Editorial Acribia. Zaragoza, España. pp 265-273. [ Links ]
41. Sly, L.I. y E. Stackebrandt (1999). Description of Skermanella parooensis gen. nov., sp. nov. to accommodate Conglomeromonas largomobilis subsp. parooensis following the transfer of Conglomeromonas largomobilis subsp. largomobilis to the genus Azospirillum. International Journal of Systematic and Evolutionary Microbiology 49: 541-544. [ Links ]
42. Sneath, P.H.A. y R.R. Sokal (1973). Numerical Taxonomy. The Principles and Practice of Numerical Classification. W. H. Freeman. San Francisco. USA. 573 p. [ Links ]
43. Simpson, E.H. (1949). Measurement of diversity. Nature 163: 688. [ Links ]
44. Sokal, R.R. y P.H.A. Sneath (1963). Numerical Taxonomy. W. H. Freeman. San Francisco. USA. [ Links ]
45. Tarrand, J.J., N.R. Krieg y J. Döbereiner (1978). A taxonomic study of the Spirillum lipoferum group, with descriptions of a new genus, Azospirillum gen. nov. and two species, Azospirillum lipoferum (Beijerinck) comb. nov. and Azospirillum brasilense sp. nov. Canadian Journal of Microbiology 24: 967-980. [ Links ]
46. Thompson, J.D., T.J. Gibson, F. Plewniak, F. Jeanmougin y D.G. Higgins (1997). The Clustal X windows interface: flexible strategies for multiple sequence alignment aided by quality tools. Nucleic Acids Research 24: 4876-4882. [ Links ]
47. Tyler, M.E., J.R. Milam, R.L. Smith, S.C. Scank y D.A. Zuberer (1979). Isolation of Azospirillum from diverse geographic regions. Canadian Journal of Microbiology 25: 693-697. [ Links ]
48. Umali-García, M., D.H. Hubbell, M.H. Gaskins y F.B. Dazzo (1980). Association of Azospirillum with grassroots. Applied and Environmental Microbiology 39: 219-226. [ Links ]
49. Vande, A. y J. Vanderleyden (1995). Genetics of the Azospirillum-Plant Root Association. Critical Reviews in Plant Sciences 14: 445-466. [ Links ]
50. Vande Broek, A., V. Keijers y J. Vanderleyden (1996). Effect of oxygen on the free-living nitrogen fixation activity and expression of the Azospirillum brasilense NifH gene in various plant-associated diazotrophs. Symbiosis 21: 25-40. [ Links ]
51. Xie, Cheng-Hui y Akira Yokota (2005). Azospirillum oryzae sp. nov., a nitrogen-fixing bacterium isolated from the roots of the rice plant Oryza sativa. International Journal of Systematic and Evolutionary Microbiology 55: 1435-1438. [ Links ]
52. Young, C.C., H. Hupfer, C. Siering, M.J. Ho, A.B. Arun, W.A. Lai, P.D. Rekha, F.T. Shen, M.H. Hung, W.M. Chen y A.F. Yassin (2008). Azospirillum rugosum sp. nov., isolated from oil-contaminated soil. International Journal of Systematic and Evolutionary Microbiology 58: 959 - 963. [ Links ]
53. Zhou, Yu, Wei Wei, Xu Wang, Lihong Xu y Ren Lai (2009). Azospirillum palatum sp. nov., isolated from forest soil in Zhejiang province, China. The Journal of General and Applied Microbiology 55: 1-7. [ Links ]
54. Zimmer, W., M. Wesche y L. Timmermans (1998). Identification and isolation of the indol-3-pyruvate decarboxylase gene from Azospirillum brasilense Sp7: sequencing and functional analysis of the gene locus. Current Microbiology 36: 327-331. [ Links ]