Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Phyton (Buenos Aires)
versión On-line ISSN 1851-5657
Phyton (B. Aires) vol.81 no.2 Vicente López jul./dic. 2012
ARTÍCULO ORIGINAL
Incremento de la diversidad genética del banco de germoplasma de Nopal usando marcadores moleculares tipo RAPD
Increase of the genetic diversity of the germplasm bank of Cactus pear using RAPD molecular markers
Escalante-González JG1, EA García-Zambrano2, A Gutiérrez-Diez2, RE Vázquez-Alvarado2, JA Torres-Castillo2, SR Sinagawa-García2, F Zavala-García2, J Martínez-de la Cerda2, C Gallegos-Vázquez3 , JI García-Zambrano4
1 Alumno del programa Ingeniero Agrónomo de la Facultad de Agronomía de la Universidad Autónoma de Nuevo León. Apartado Postal 358, San Nicolás de los Garza N. L. C.P. 66450. México.
2 Maestro Investigador de la Facultad de Agronomía de la Universidad Autónoma de Nuevo León, México.
3 Maestro Investigador de la Universidad Autónoma de Chapingo, Centro Regional Universitario Centro Norte. Km. 5,5 Carretera Zacatecas-Guadalajara, El Orito, Zacatecas, C.P. 98060, México.
4 Maestro del Instituto Tecnológico y de Estudios Superiores de Monterrey. Campus Monterrey. Ave. Eugenio Garza Sada 2501, Col. Tecnológico, Monterrey, N.L., C.P. 64700, México.
Address Correspondence to: Eduardo Alejandro García Zambrano, e-mail: eagarci1@hotmail.com ; eduardo.garciazb@uanl.edu.mx
Recibido / Received 28.VII.2011.
Aceptado / Accepted 18.XII.2011.
Resumen. El objetivo de esta investigación fue incrementar la diversidad genética del Banco de Germoplasma de Nopal de la FAUANL utilizando marcadores moleculares del tipo RAPD para estimar la diversidad genética de 15 accesiones de nopal a partir de marcadores moleculares registrados en el Banco. La extracción de ADN se realizó por el método del ruptor celular usando el Kit FastDNA®. La cuantificación del rendimiento del ADN se realizó a través del Picofour. Se observó que las concentraciones de ADN fluctuaron entre 74,91 ng/µL, y 12,32 ng/µL. Se usaron 12 iniciadores generando de 1 a 5 bandas por iniciador con un peso molecular que fluctuó entre los 200 a 1000 pb. El índice de diversidad (ID) promedio de las 15 accesiones fue de 0,8539. Para el análisis de los datos moleculares se utilizó la matriz binaria de 20 marcadores registrados en el Banco, junto con los marcadores generados de las accesiones colectadas. Se utilizó el análisis de conglomerados jerárquico mediante el método de agrupamiento de pares no ponderados (UPGMA). El dendograma generado mostró claramente que no existe ninguna accesión con distancia genética de cero, por lo cual se concluye que no existe duplicidad entre las accesiones mencionadas.
Palabras clave: Marcadores moleculares; RAPD; Accesiones.
Abstract. The objective of this research was to increase the genetic diversity of the Nopal Germplasm Bank of the FAUANL using RAPD molecular markers to estimate the genetic diversity of 15 accessions of cactus pear from molecular markers recorded in the Bank. DNA extraction was performed by the method of cellular ruptor using FastDNA® Kit. Performance measurement of DNA was carried out through Picofour. It was observed that the DNA concentrations ranged from 74.91 ng/µL to 12.32 ng/uL. Twelve primers were used to generate from 1 to 5 bands per primer with a molecular weight ranging from 200 to 1000 bp. The average diversity index (DI) of the 15 accessions was 0.8539. For the analysis of molecular data, it was used the binary matrix of 20 markers recorded in the bank, along with the markers generated from the collected accesions. We used cluster analysis by the method of unweighted pair-grouping (UP-GMA). The generated dendrogram showed clearly that there was no accession with a genetic distance of zero, and it was determined that there was no duplication among the mentioned accessions.
Keywords: Molecular markers; RAPD; Accessions.
INTRODUCCIÓN
Los recursos fitogenéticos constituyen un patrimonio de la humanidad de valor incalculable. Su pérdida es un proceso irreversible que supone una grave amenaza para la estabilidad de los ecosistemas, el desarrollo agrícola y la seguridad alimentaria del mundo (Martín, 2003). Reconociendo la importancia del nopal como recurso natural mexicano, se salvaguarda en bancos de germoplasma. Estos bancos contienen colecciones de valor comercial o agronómico que se describen desde un punto de vista morfológico y antropocéntrico (Pimienta, 1997). La Facultad de Agronomía de la Universidad Autónoma de Nuevo León cuenta con un Banco de Germoplasma de Nopal formado por 100 accesiones. A las mismas se les estimó su diversidad genética a través de una base de datos moleculares registrada en el Banco, a partir de 12 iniciadores los cuales generaron 20 marcadores moleculares por medio de la técnica de RAPD (García, 2006).
Hoelzel (1994) y Liu (2004) informaron que la alternativa para evaluar correctamente la biodiversidad en las poblaciones de cactáceas es la caracterización genética con huellas de ADN. Esta es la única forma de identificación que puede proteger los recursos genéticos en forma eficiente, porque es infalsificable, y en contraste con los marcadores morfofisiológicos, no es afectada por las condiciones ambientales.
El objetivo de esta investigación fue incrementar la diversidad genética del Banco de Germoplasma de Nopal de la Facultad de Agronomía de la Universidad Autónoma de Nuevo León utilizando marcadores moleculares de tipo RAPD en el material colectado de Opuntia spp. a partir de marcadores moleculares registrados en el Banco.
MATERIALES Y MÉTODOS
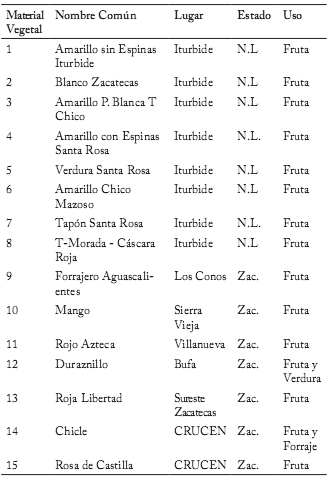
Material vegetal. El material colectado consistió en 15 accesiones de las cuales se colectaron 8 en el municipio de Iturbide, localizado al sureste de Nuevo León, México, en la región denominada Sierra Madre Oriental (24° 43' 60'' N, 99° 53' 60' 0, 1850 msnm). Las otras 7 accesiones provinieron del Banco de Germoplasma del Centro Regional Universitario Centro Norte de la Universidad Autónoma de Chapingo, en el Estado de Zacatecas, dentro de la localidad denominada "Huertas El Orito" (22° 44' 47, 28'' N, 102° 36' 26, 90'' O, Tabla 1). Las muestras fueron llevadas al Laboratorio de Biotecnología de la Facultad de Agronomía de la UANL Campus Ciencias Agropecuarias para ser analizadas.
Tabla 1. Nombre común, lugar y Estado en que se efectuó la colecta, así como el uso principal que se le da a las accesiones de nopal.
Table 1. Common name, location, and State where the collection was conducted and primary use given to the accessions of cactus pear.
Extracción de ADN. Se realizó la extracción de ADN por el método del ruptor celular con el kit FastDNA® (Qbiogene, Inc., C.A.). La cuantificación del ADN se realizó mediante el Picofluor; la calidad del ADN se determinó en un gel de agarosa 1%.
Generación de RAPD en Nopal. Para la generación de RAPD en nopal se siguió como base el protocolo de Williams et al. (1990), realizando algunas modificaciones. Las reacciones de amplificación (Tabla 2) se llevaron a cabo en un volumen de reacción de 25µL, utilizando los 12 iniciadores (Tabla 3) que dieron origen a los 20 marcadores registrados en el Banco, señalados por García et al. (2006). Las muestras fueron colocadas en el termociclador Termo Hybrid PCR Express; el programa térmico utilizado fue 1 ciclo inicial de 93 °C por 1 min, 45 ciclos de 92 °C por 1 min; 34 °C por 1 min; 71 °C por 1 min, y 1 ciclo fnal de 72 °C por 5 min. El periodo de tiempo en que se procesaron las muestras fue de 255 minutos.
Tabla 2. Reactivos para PCR.
Table 2. Reagents for PCR.

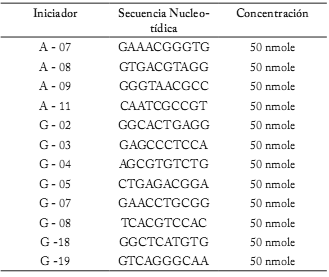
Tabla 3. Iniciadores utilizados para la estimación de diversidad genética.
Table 3. Primers used for estimating genetic diversity.
Se cargaron 20 µL de los productos de amplificación, más 3 µL de buffer de carga BioLine® al 5X utilizando un marcador de 100 pb BioLine® en un gel de agarosa al 2% teñido con 2 µL de Syber Green®; tiempo de corrida del gel fue de una hora a 120V.
Análisis de datos moleculares. Para la clasificación de Opuntia se usó el análisis por conglomerados ("cluster analysis") o "agglomerative hierarchical cluster analysis" (Luna et al., 2007). Se ha demostrado que dicho método es tan robusto para la reconstrucción de relaciones filogenéticas jerárquicas como los métodos cladísticos (James y McCulloch, 1990).
Los fragmentos de ADN amplificados bien definidos (bandas) obtenidos con los iniciadores, se analizaron visualmente para generar una matriz de datos binaria (0 y 1) donde "0" representa la ausencia de la banda y "1" representa la presencia de la banda (Bramardi, 2002). Con la matriz de datos binaria se construyó la matriz de distancias genéticas mediante la comparación de cada par de genotipos utilizando el método de Nei y Li (1979).
Para la formación de los conglomerados por el método jerárquico se usó el método de agrupamiento de pares no ponderados (UPGMA) para maximizar la variación entre los grupos y minimizar dicha variación dentro de ellos (Hair et al., 1992).
RESULTADOS Y DISCUSIÓN
Extracción de ADN. Se observó que las concentraciones de ADN fueron mayores cuando se utilizaron cladodios jóvenes, obteniendo el valor más alto de 74,91 ng/µL en la accesión Verdura Santa Rosa. Sin embargo, el valor más bajo (12,32 ng/µL) se obtuvo en la accesión Tuna Morada-Cáscara Roja, a pesar de utilizar un cladodio joven para la extracción. Esto podría estar relacionado con la rápida oxidación que presentó la muestra.
Generación de datos moleculares. Los resultados obtenidos de los 12 iniciadores (A-07, A-08, A-09, A-11, G-02, G-03, G-04, G-05, G-07, G-08, G-18, G-19) que dieron origen a los 20 marcadores registrados en el Banco fueron satisfactorios. Esto se debió a que se obtuvieron de 1 a 5 bandas por iniciador con un peso molecular que fluctuó entre los 200 a 1000 pb. Los iniciadores que rindieron mejores resultados de amplificación en las 15 accesiones analizadas fueron el A-11 y G-02 con un 100 y 80%, respectivamente. Los que presentaron menos amplificación fueron el A-08 y G-18 con un 13 y 40%, respectivamente. En esta investigación se generaron diez marcadores que no se encontraron registrados en el banco de germoplasma. A estos se deben sumar los veinte marcadores generados por García et al. (2006), formando la base de datos moleculares del Banco de Germoplasma de Nopal de la FAUANL.
El índice de diversidad (ID) promedio de las 15 accesiones de nopal fue de 0,8539, el cual es muy similar al reportado por García et al. (2006). Esta diferencia se debió a que el 46% del material colectado de nopal fue del Banco de Germoplasma del Centro Regional Universitario Centro Norte de la Universidad Autónoma de Chapingo, en el cual existe una alta variabilidad genética; el 54% del material restante se colectó en nopaleras del municipio de Iturbide Nuevo León, México, buscando ejemplares distintos morfológicamente de los que cuenta el banco.
Scheepers et al. (2000) sostienen que las críticas que comúnmente se hacen contra la tecnología RAPD no son generalizables en cuanto a la reproducción de los resultados. Esto es debido a que en sus investigaciones ellos los han repetido utilizando diversos tejidos de las plantas, diferentes tipo de Taq polimerasa y diferentes máquinas PCR, obteniendo siempre los mismos resultados. En esta investigación, se realizaron pruebas con las accesiones Roja Libertad y Chicl usando los iniciadores G-04, G-05, G-19 (Fig. 1). Se hicieron 5 extracciones de ADN en cada muestra, las cuales se procesaron para la generación de RAPD con diferentes tipos de Taq (Taq polimerasa® y Go Taq® Flexi), obteniendo el 100% de reproducibilidad entre las accesiones.
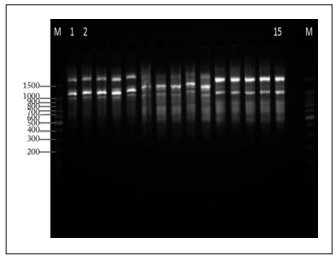
Fig. 1. Productos amplificados por los iniciadores G-04 (carriles 1-5), G-05 (carriles 6-10) y G-19 (carriles 11-15) con la accesión 14 Chicle. El marcador (M) de 100 pb se aprecia en los extremos del gel de agarosa al 2%.
Fig. 1. Amplified products by primers G-04 (lanes 1-5), G-05 (lanes 6-10) and G-19 (lanes 11-15) with the accession 14 Chicle. The marker (M) of 100 bp is apparent at the ends of the agarose gel to 2 %.
Análisis de datos moleculares por el método de UPGMA. Para el análisis de los datos moleculares, se incorporaron los marcadores que se generaron de las 15 accesiones colectadas a la matriz binaria de las 100 accesiones registradas en el banco, considerando solamente los 20 marcadores moleculare reportados por García et al. (2006): A-07(300 pb), A-08 (314 pb), A-08 (400 pb), A-09 (700 pb), A-11 (300 pb), A-11 (500 pb), G-02 (450 pb), G-03 (500 pb), G-04 (300 pb), G-04 (635 pb), G-05 (600 pb), G-07 (550 pb), G-08 (300 pb) G-08 (465 pb), G-08 (500 pb), G-18 (700 pb), G-19 (300 pb), G-19 (500 pb), G-19 (730 pb), G-19 (965 pb).
Del total de las accesiones que tiene el banco, aunado con el material colectado que se desea incorporar, 15 accesiones presentaron una distancia genética de cero, de las cuales 3 son del material colectado en esta investigación (Amarillo P Blanca T Chico, Amarillo Chico Masozo y Tuna Morada-Cáscara Roja, Fig. 2), y las 12 accesiones restantes fueron analizada molecularmente por García et al. (2009). Ellos aplicaron la técnica de AFLP (Amplification Fragment Length Polymorphisms), mencionando que las accesiones reportadas como posibles duplicados por medio de la técnica de RAPD presentaron una distancia genética mayor que cero, por lo cual se descartó la duplicidad.

Fig. 2. Dendrograma generado a partir de datos moleculares de las 100 accesiones que tiene el Banco, y las 15 accesiones colectadas para ser incluidas en el Banco de Germoplasma de la FAUANL.
Fig. 2. Dendrogram generated from molecular data of the 100 accessions of the Germplasm Bank and the 15 accessions collected for inclusion in that Bank of the FAUANL.
El material colectado siguió un comportamiento similar en su agrupamiento a las 100 accesiones reportadas por García et al. (2006), formándose tres grupos en un rango de 0 a 8 unidades, dentro de los cuales se produjo la incorporación del nuevo material dentro del primer grupo en dos subgrupos.
El primer grupo se encontró formado por 53 accesiones teniendo como característica cladodios ovalados y elípticos. Con la inclusión de los nuevos materiales se anexaron dos subgrupos formados por cuatro accesiones (Amarillo sin Espinas Iturbide, Amarillo P Blanca T Chico, Amarillo Chico Masozo y Tuna Morada-Cáscara Roja) y 11 accesiones (Rosa de Castilla, Amarillo con Espinas Santa Rosa, Chicle, Tapón Santa Rosa, Rojo Azteca, Mango, Duraznillo, Forrajero Aguascalientes, Roja Libertad, Verdura Santa Rosa y Blanco Zacatecas), que compartieron la misma característica del grupo. En el subgrupo de las cuatro accesiones se observó que las mismas fueron colectadas en el municipio de Iturbide N.L., y éstas se agruparon en forma consecutiva en el dendrograma: tres de ellas mostraron una distancia genética de cero; es posible que este material sea duplicado, es decir que sea el mismo tipo de nopal.
El segundo grupo se encontró compuesto por 59 accesiones, las cuales se usan para la producción de fruta y verdura. En lo que se refiere al tercer grupo se encontró compuesto por 3 accesiones, las cuales no compartieron ninguna característica en común, por lo cual no se le dio ningún nombre específico.
Para poder determinar si las 3 accesiones que presentaron distancia genética 0 en la Figura 2 son duplicadas, se analizó por separado el material colectado utilizando 30 marcadores moleculares. De estos, 20 marcadores fueron los que usaron para realizar la estimación de la diversidad genética del banco y 10 marcadores se generaron en esta investigación [A-07 (100 pb), A-07 (600 pb), A-09 (200 pb), A-02 (850 pb), G-04 (700 pb), G-04 (810 pb), G-05 (1000 pb), G-05 (800 pb), G-05 (700 pb), G-19 (410 pb)]. Las 15 accesiones colectadas formaron dos grupos: uno con 8 accesiones, procedentes del municipio de Iturbide N. L. ,y el otro con 7 accesiones, las cuales provinieron del Banco de Germoplasma del CRUCEN-UACh en el Estado de Zacatecas (Fig. 3). Las accesiones tuvieron la producción de tuna como característica principal. Ninguna accesión tuvo distancia genética de cero en el dendrograma, por lo cual no existe duplicidad entre las accesiones colectadas.

Fig. 3. Dendrograma generado por las 15 accesiones colectadas utilizando 30 marcadores moleculares.
Fig. 3. Dendrogram generated by the 15 collected accessions using 30 molecular markers.
CONCLUSIONES
La utilización de marcadores genéticos moleculares tipo RAPD es una herramienta eficiente para la estimación de la diversidad genética en nopal, al haberse detectado duplicidad entre las accesiones en el banco de germoplasma.
Se generaron 10 marcadores polimórficos de tipo RAPD, adicionales a los 20 que tiene registrado el banco de germoplasma. Estos sirvieron para estimar de una manera más precisa la diversidad genética entre las accesiones de nopal que se quieren establecer dentro del banco de germoplasma de nopal de la FAUANL.
Los marcadores moleculares tipo RAPD generados de las 15 accesiones colectadas se analizaron junto a la base de datos moleculares que se tienen registrados en el banco. Se concluye que ninguna de las accesiones es duplicada, debido a que la distancia genética existente en las accesiones colectadas nunca fue cero, parámetro por el cual se detectan posibles duplicados en el dendograma.
AGRADECIMIENTOS
Al Programa de Apoyo a la Investigación Científica y Tecnológica (PAICYT) de la Universidad Autónoma de Nuevo León por el financiamiento para la realización de esta investigación.
A la Facultad de Agronomía de la Universidad Autónoma de Nuevo León por el apoyo brindado para realización de esta investigación.
Al Centro Regional Universitario Centro Norte de la Universidad Autónoma de Chapingo por la donación de material vegetal al Banco de Germoplasma de Nopal de la FAUANL.
REFERENCIAS
1. Bramardi, S. (2002). Análisis multivariado. Su aplicación en la caracterización de recursos genéticos. Facultad de Ciencias Agrarias, Universidad Comahue, Estación Exp. INTA, Argentina. 60 p. [ Links ]
2. García, E., A. Gutiérrez, G. Salinas, E. Cárdenas, R. Vázquez, F. Zavala y J. Martínez (2006). Clasificación y estimación de la diversidad genética de Nopal Opuntia spp. en base a descriptores fenotípicos y marcadores genético moleculares. PHYTON Revista Internacional de Botánica Experimental 75: 125-135. [ Links ]
3. García, E., A. Gutiérrez, M. Ojeda, F. Zavala, e I. Cerda (2009). Estimación de la diversidad genética del Nopal, usando marcadores moleculares tipo AFLP. PHYTON Revista Internacional de Botánica Experimental 78: 4-11. [ Links ]
4. Hair, J., R. Anderson, R. Tatham, y W. Black (1992). Multivariate data analysis. MacMillan Publ. Co. Nueva York. 544 p. [ Links ]
5. Hoelzel, A.R. (1994). Molecular Genetic Analysis of Populations. A Practical Approach. IRL Press. Oxford, USA. 315 p. [ Links ]
6. James, F.C., and CH. McCulloch (1990). Multivariate analysis in ecology and systematics: Panacea or Pandora's box Annual Review of Ecology, Evolution and Systematics 21: 129-166. [ Links ]
7. Liu, Z.J. y J.F. Cordes (2004). DNA marker technologies and their applications in aquaculture genetics. Aquaculture 238: 1-37. [ Links ]
8. Luna P., E. Valadez, A. Barrientos y C. Gallegos (2007). Caracterización de Opuntia spp. mediante semilla con Marcadores RAPD e ISSR y su Posible Uso para Diferenciación. 44 p. [ Links ]
9. Martín, I. (2003). Centro de Recursos Fitogenéticos (CRF). Instituto Nacional de Investigación y Tecnología Agraria y Alimentaria (INIA). p 2. [ Links ]
10. Nei, M. y W.H. Li (1979). Mathematical model for studying genetic variation in terms of restriction endonucleases. Proceeding of the National Academy of Sciences of the USA 76: 5269-5273. [ Links ]
11. Pimienta, E. (1997). Suculentas mexicanas / cactáceas. El nopal en México y el mundo. Departamento de Ecología. Centro de Ciencias Biológicas y Agropecuarias. Universidad de Guadalajara. CVS Publicaciones. pp. 87-99. [ Links ]
12. Scheepers, D., M. Eloy y M. Briquet (2000). Identification of larch species (Larix deciduas, Larix kaempferi and Larix X eurolepisi) and estimation of hybrid fraction in seed lots by RAPD fingerprints. Theoretical and Applied Genetics 100: 71-74. [ Links ]
13. Williams, J.G.K., A. Kubelik, K. Livak, J. Rafalski y S. Tingey (1990). DNA polymorphisms amplified by arbitrary primers are useful as genetic markers. Nucleic Acids Research 18: 6531-6535. [ Links ]














