Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Phyton (Buenos Aires)
versión On-line ISSN 1851-5657
Phyton (B. Aires) vol.83 no.2 Vicente López dic. 2014
ARTÍCULOS ORIGINALES
Agrupamiento de genotipos de nopal (Opuntia spp.) de México por medio de la técnica de AFLPs y características del fruto
Cladistics of Mexican nopal genotypes (Opuntia spp.) based on AFLP markers and fruit characters
Espinoza Sánchez EA1, H Silos Espino1, S Flores Benitez1, LL Valera Montero 1 , E Rodríguez Salazar2, C Gallegos Vázquez3, F Guevara Lara4, M González Chavira5, HS Guzmán Maldonado5
1 Laboratorio de Biotecnología Aplicada, Instituto Tecnológico El Llano, Aguascalientes, Km 18 carretera Aguascalientes-San Luis Potosí, México, C.P. 20330.
2 Centro de Bachillerato Tecnológico Agropecuario No. 138, Km 1, carretera Villa Hidalgo-Pinos, Zacatecas, México, C.P. 98980.
3 Depositario Nacional de Opuntia, Centro Regional Universitario Centro Norte de la Universidad Autónoma de Chapingo. Cruz del Sur # 100, Colonia Constelación, El Orito, Zacatecas, México C.P. 98085.
4 Laboratorio de Biotecnología y Funcionalidad de Alimentos, Universidad Autónoma de Aguascalientes, Av. Universidad No. 940, Aguascalientes, Aguascalientes., México, C.P. 20100.
5 Campo Experimental Bajío, Instituto Nacional de Investigaciones Forestales, Agrícolas y Pecuarias, Km 6.5, Carretera Celaya-San Miguel de Allende, Roque, Celaya Guanajuato. México, C.P. 38110.
Address Correspondence to: Héctor Silos Espino, e-mail: silosespino@hotmail.com
Recibido / Received 2.V.2012.
Aceptado / Accepted 15.III.2013.
Resumen. Para contribuir al conocimiento de la diversidad genética del nopal se analizaron 85 genotipos representativos de Opuntia (silvestres y cultivados) por medio de la técnica de AFLPs y características del fruto. El análisis de AFLPs con los iniciadores Mse ICAC/Eco RI-AAG generaron más fragmentos polimórficos (57,5 en promedio) por genotipo; se obtuvo un dendrograma que no mostró una clara relación de acuerdo a la taxonomía conocida. En complemento al perfil genético, y para permitir una referencia morfológica o química directa, agrupamos a los genotipos de acuerdo al contenido de °Brix de sus frutos, debido a que esta es una característica primordial para su identificación visual y aprovechamiento de consumo directo. Se encontró que los genotipos que crecieron bajo condición silvestre mostraron los valores más contrastantes en el contenido de °Brix. Las características de los frutos (morfológicas y °Brix) mostraron un rango amplio para los genotipos silvestres. Por ejemplo, los genotipos Tapón Blanco y Guilanche tuvieron una concentración de entre 21-22 °Brix, y los genotipos correspondientes al tipo xoconostles (frutos ácidos), entre 5,0-8,2 °Brix. Con la información genética y fisicoquímica obtenida se espera referenciar la gran riqueza biológica del nopal conservada en dos bancos de germoplasma de nopal nacional, como una estrategia para la identificación de genotipos Así mismo, esta información podría ser utilizada en programas de mejoramiento genético del género Opuntia.
Palabras clave: Caracterización Molecular; Opuntia; AFLP; Dendrograma; Morfología del fruto.
Abstract. Dendrograms were constructed based on AFLP markers and a table was used to group fruit characters of wild and cultivated genotypes of Mexican nopal (Opuntia spp.). The AFLP dendrogram obtained using 57.5 markers (from primers Mse I-CAC/Eco RI-AAG) did not agree with the known taxonomic classification for this species. Additionally, a complementary table for fruit characters (morphological and °Brix) showed a wide range for wild genotypes, e.g. from 21-22 °Brix for 'Tapón Blanco' and 'Guilanche' genotypes, to 5.0-8.2 °Brix for xoconostles (acidic fruits). Even though there were some divergences with taxonomic classification, it is expected that both genetic and physicochemical information would contribute to a more appropriate identification of Opuntia genotypes that, in turn, may be used in breeding programs of this genus.
Keywords: Molecular Characterization; Opuntia; AFLP; Dendrogram; Fruit morphology.
INTRODUCCIÓN
Las plantas de nopal del género Opuntia se encuentran distribuidas por todo el territorio mexicano (Bravo-Hollis, 1978), en casi todos los tipos de regiones fisiográficas. Crece en forma silvestre y se propaga de forma sexual y asexual por medio de cladodios (clonal). Esto permite una gran cantidad de variantes susceptibles de estudio (Gallegos y Mondragón, 2011). El aspecto morfológico y de forma de crecimiento de los nopales hace que se les asignen nombres comunes de acuerdo a cada región para un solo tipo de nopal, y en tipos de vegetación conocidas como nopaleras (Rzedowski, 1978), donde crecen de manera silvestre. Como resultado, es altamente probable que se generen nuevos genotipos con algún potencial, sin que se le brinde la atención respectiva para su aprovechamiento o conservación (Gallegos-Vázquez et al., 2012). Algunas características específicas del nopal son su color de fruto, contenido de semillas, espinas, gloquidios, y una alta frecuencia de polinización cruzada, lo que hace difícil algunas veces establecer un criterio apropiado para la identificación de genotipos. Se han realizado diversos estudios para conocer la riqueza biológica del nopal. Por ejemplo, en morfología, Scheinvar (2010) propuso 93 especies para el subgénero Opuntia y 10 para el subgénero Nopalea. Mondragón-Jacobo y Bordelon (2002) encontraron que el color de fruto es un carácter apropiado para la agrupación en 32 genotipos de nopal. Reyes-Agüero et al. (2005, 2009) con base en la morfología de la planta, cladodio, flor, fruto y semilla reportaron 21 especies de 243 genotipos en la Altiplanicie Meridional de México, con variantes distribuidos de la siguiente manera: O. albicarpa (trece), O. chaveña (seis), O. durangensis (uno), O. ficus-indica (siete), O. hyptiacantha (siete), O. joconostle (seis), O. lasiacantha (cuatro), O. lindheimeri (uno), O. megacantha (veintidós), O. robusta (dos) y O. streptacantha (ocho) genotipos respectivamente. Por otro lado, también se han realizado algunas investigaciones que incluyen marcadores moleculares para establecer diferencias entre las especies. Usando la técnica de RAPDs, Wang et al. (1998) encontraron diferencias entre O. ficus-indica, O. hyptiacantha, O. cochinifera, O. lindheimeri var. Inermis y O. ellisinana; García-Zambrano et al. (2006) separaron dos grupos de 100 genotipos sin encontrar una clara relación en cuanto a la caracterización taxonómica conocida. Por medio de la técnica de AFLPs y chSSR, Labra et al. (2003) encontraron similitud entre especies distintas, y propusieron que O. ficus-indica debería ser considerada como una planta domesticada a partir de O. megacantha. Luna-Páez et al. (2007) usando una combinación entre RAPDs e ISSR en embriones apomícticos de 22 genotipos silvestres y cultivados encontraron alta relación entre genotipos de O. ficus-indica y O. megacantha con los de O. streptacantha. Estos autores consideraron a los dos primeros genotipos como mejorados, mientras que el último fue un genotipo silvestre. Como ejemplo de genotipos cultivados tenemos a O. megacantha (Morada T-10 y Pico Chulo), O. ficus-indica (genotipos sin espinas, Milpa Alta y Rojo Pelón), O. albicarpa (Cristalina y Burrona). Como ejemplo de genotipos silvestres; O. joconostle (Xoconostle Cuaresmeño), O. streptacantha (Cardona), O. robusta (Tapón); O. hyptiacantha (Charola), O. lasiacantha (Sanjuanera), O. undulata (Bolañera) y O. cochinifera (Cacalote). La técnica de AFLPs provee más información para agrupar genotipos por la amplificación de fragmentos de ADN, lo que podría contribuir a la identificación de genotipos de Opuntia. Por lo tanto, el objetivo de este trabajo fue analizar 85 genotipos representativos para obtener su perfil genético y agrupamiento por el tamaño y contenido de °Brix del fruto, por ser éste último un elemento de rápida relación identificación y con potencial de aprovechamiento para consumo.
MATERIALES Y MÉTODOS
Material vegetal. Se utilizaron 85 genotipos de nopal (Opuntia spp.), un genotipo de Pereskiopsis rotundifolia y otro de Opuntia imbricata, siendo estos dos últimos controles del análisis. Los genotipos fueron proporcionados por los Bancos de Germoplasma "Efraím Hernández Xolocotzi" (CBTA No. 138 Villa Hidalgo, Zac., México) y el Depositario Nacional de Opuntia (Centro Regional Universitario Centro Norte de la Universidad Autónoma Chapingo) ubicado en el Orito, Zacatecas, México (22° 44.7' N, 102° 36.4' O). Los genotipos de nopal se encuentran debidamente registrados e identificados a nivel de especie y la información puede ser consultada en las siguientes citas (Reyes Agüero et al., 2009; Gallegos y Mondragón, 2011).
Extracción de ADN. De cada genotipo, se obtuvieron muestras de 200 mg para la extracción de ADN los cuales fueron macerados hasta polvo fino con nitrógeno líquido. El ADN fue extraído por dos métodos: el descrito por Tel-Zur (1999) y el protocolo CTAB con modificaciones (CTAB-Fenol, CTAB- Fenol-Cloroformo, CTAB-Etanol y CTAB-Octanol).
Análisis de AFLP. La metodología para los análisis de AFLPs fue realizada en el laboratorio de análisis moleculares del INIFAP-Celaya, México, siguiendo a LI-COR Biosciences® (LI-COR, 2010) y Vos et al. (1995). Se utilizaron 6 combinaciones de iniciadores: Mse I-CAA/Eco RI-ACC, Mse I-CAA/Eco RI-AGC, Mse I-CAA/Eco RI-AAC, Mse I-CAA/Eco RI-AAG, Mse I-CAC/Eco RI-AAG y Mse ICAC/Eco RI-ACA. La digestión se realizó con un volumen final de 515 µL de mezcla de reacción que contenía: 100 µL de ddH2O Sigma®, 300 µL de Buffer R/L 10 X, 50 µL de Tru 9 (10 U/µL), 15 µL de ADN muestra (100 ng/ µL), 50 µL de Eco RI (10 U/µL), a 37 °C por 3,5 h y 70 °C por 15 min. La ligación se realizó con una mezcla de reacción de 200 µL de ddH2 O Sigma®, 100 µL de Buffer R/L 10 X, 500 µL de T4 ADN Ligasa (1 U/µL), 100 µL de Adaptador Mse I (50 pmol/µL), 100 µL de Adaptador Eco RI (5 pmol/µL), 120 µL de ATP 10 mM, a 37 °C por 5,5 h; la dilución de muestra fue de 2 µL por 18 µL de agua ultrapura. La preamplificación se realizó con un volumen final de 1 mL de la mezcla de reacción que contenía: 540 µL de ddH2O Sigma®, 125 µL de PCR Buffer 10 X, 60 µL de MgCl2 50 mM, 100µL de dNTPs 2.5 mM c/u, 25 µL de Taq (5 U/µL), 75 µL deIniciador Mse I+C 50 ng/µL, y 75 µL de Iniciador Eco RI+A 50 ng/µL con un ciclo de 94 °C por 2 min, 20 ciclos de 94°C por 30 s, 56 °C por 30 s, 72 °C por 1 min y 7 min a 75 °C; las muestras fueron diluidas utilizando 1 µL con 39 µL de agua ultrapura. La amplificación se realizó con un volumen final de 377,2 µL de mezcla de reacción que contenía: 148µL de ddH2O (Sigma®), 50 µL de PCR Buffer 10 X, 24.2 µL de MgCl2 50 mM, 40 µL de dNTPs 2.5 mM c/u, 15 µL de Taq (5 U/µL), 50 µL de Mse I 50 ng/µL, 25 µL del Iniciador Eco RI 10 pmol/IRDye700 nm, y 25 µL Iniciador Eco RI 10 pmol/IRDye800 nm; la reacción se realizó a 94 °C por 2 min, 94 °C por 30 s, 65 °C por 30 s, 72 °C por 1 min, 12 ciclos del ciclo 94 °C por 30 s, 65 °C por 30 s, 72 °C por 1 min, 23 ciclos de 94 °C por 30 s, 56 °C por 30 s, 72 °C por 1 min, un ciclo de 7 min a 72 °C, 4 °C. El touchdown incluyó 94 °C por 3 min, un ciclo de 94 °C por 30 s, 56 °C por 30 s, 72 °C por un min, 12 ciclos de 94 °C por 30 s, 65 °C por 30 s (0.7°C por ciclo), 23 ciclos de 94 °C por 30 s, 56 °C por 30 s, 72°C por un min y un ciclo de 5 min a 72 °C, 4 °C. Se añadió 1,5 mL de buffer de carga a igual volumen de reacción (80% formamida, 1 mg/mL xileno-cianol FF, 1 mg/mL bromofenol-blue, 10 M EDTA, pH 8,0), desnaturalizado con Blue stop buffer® (LICOR®) por 3 min a 92 °C, cargado en un gel de poliacrilamida al 6,5% en buffer TBE por 2 h a 40 W. La secuenciación fue realizada en un secuenciador ADN Li-Cor 4200® con dos filtros: 700 nm y 800 nm. Las bandas polimórficas fueron analizadas y contabilizadas manualmente y las matrices resultantes fueron utilizadas para la construcción de un dendrograma basado en el coeficiente de Jaccard usando UPGMA con el software NTSYS pc 2.1 (Exeter Software, Setauket, New York, U. S.A).
Mediciones morfológicas de los frutos. Para inferir el perfil genético obtenido con la morfología del nopal se analizaron frutos de los mismos genotipos. Así, se determinó su coloración con el código Munsell (Munsell, 1977), y se realizaron mediciones de longitudes polares y diámetros ecuatoriales utilizando un vernier digital (CALDI-6MP Truper®, Switzerland). Los pesos se obtuvieron con una balanza analítica (OHAUS® analytical plus, Ohaus Inc. Florham Park, Switzerland). El contenido de sólidos solubles expresados como °Brix fue obtenido con un refractómetro portátil (Erma®, Hand Refractometer® Tokio, Japón). Las medidas fueron realizadas en el Laboratorio de Biotecnología Aplicada del Instituto Tecnológico El Llano, Aguascalientes, México.
RESULTADOS Y DISCUSIÓN
Análisis genético. El número de fragmentos generados con los diferentes iniciadores en los genotipos dulces y ácidos fue de entre 37,9 a 57,5 en promedio (Tabla 1), los cuales variaron en tamaño entre 50-700 pb (Fig. 1, Fig. 2). Uno de los genotipos de Opuntia (Camueso Redondo) mostró 336 fragmentos amplificados. Le siguió un xoconostle (Limón) con 315 fragmentos lo que podría indicar que la característica dulce o ácido puede residir en muy poca diferenciación en sus ADN. Los fragmentos generados con el iniciador M-CAC/E-AAG permitieron distinguir a la especie más alejada (P. rotundifolia) de los genotipos de nopal analizados. Por consiguiente, los fragmentos generados con este iniciador se utilizaron para construir un dendrograma y obtener la relación genética respectiva (Fig. 3 A).
Tabla 1. Fragmentos de ADN amplificados por AFLPs de genotipos silvestres y cultivados de Opuntia spp. de México.
Table 1. AFLP amplicons from wild and cultivated Mexican Opuntia spp. genotypes


Iniciadores: A = M-CAA/E-ACC; B = M-CAA E-AGC; C = M-CAA/ E-AAC; D = M-CAA/ E-AAG; E = M-CAC/ E-AAG; F = M-CAC/E-ACA.

Fig. 1. Perfil genético de genotipos de nopal de frutos dulces (Opuntia spp., >10,3 °Brix) generado por AFLPs con los iniciadores M-CAC/E-AAG y dos controles (C1: Pereskiopsis rotundifolia y C2: Opuntia imbricata). El número de la parte superior indica la línea con el genotipo descrito en la Tabla 2. El número al lado izquierdo indica el marcador de peso molecular.
Fig. 1. AFLP fragments, using primers M-CAC/E-AAG, of sweet Opuntia spp. (>10.3 °Brix) and Pereskiopsis rotundifolia and Opuntia imbricata (C1 and C2, respectively). Numbers on top correspond to the ID numbers from Table 1. Molecular weight is described on the left side.

Fig. 2. Perfil genético de genotipos de nopal de frutos ácidos (Opuntia spp., <10,3 °Brix) generado por AFLPs con los iniciadores M-CAC/E-AAG. El número de la parte superior indica la línea con el genotipo descrito en la Tabla 2. El número al lado izquierdo indica el marcador de peso molecular.
Fig. 2. AFLP profile of nopal (Opuntia spp.) genotypes having acidic fruits (<10.3 °Brix) using primers M-CAC/E-AAG. Numbers on top of the figure refer to genotypes described on Table 2. Numbers on the left correspond to molecular weight.
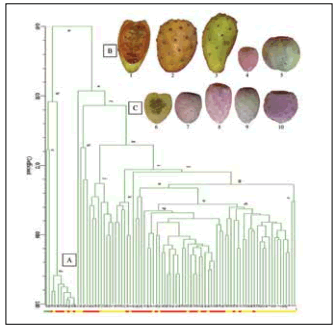
Fig. 3. (A) Dendrograma de 85 genotipos de nopal (Opuntia ssp.) generado por AFLPs con los iniciadores M-CAC/E-AAG. El número inferior indica el genotipo de nopal descrito en la Tabla 1. La barra de color verde indica control, la de color amarillo a genotipos de nopal de fruto ácido y la de color rojo a genotipos de nopal de fruto dulce. (B) Frutos de genotipos de nopal de fruto dulce. 1: Anaranjada, 2: Zarca (2.5 GY 8/10), 3: Blanca de Castilla (2,5 GY 8/8), 4: Duraznillo Rojo (5RP 6/10) y 5: Tapón Blanco (5 GY 7/6). (C) Frutos de genotipos de nopal de fruto ácido conocidos como xoconostles. 6: Chiché (5 RP 5/9), 7: Rosa (5 RP 6/10), 8: Púrpura (5 RP 7/8), 9: Chacalote (5 RP 5/8) y 10: Pijo (5 RP 6/8).
Fig. 3. (A) AFLP dendrogram from 85 Mexican nopal genotypes (Opuntia spp.) using primers M-CAC/E-AAG. Numbers at the bottom of the dendrogram correspond to the ID numbers from Table 1. Clusters: yellow bars indicate acidic genotypes, red bars indicate sweet genotypes, and the green bar is for control. (B) Sweet-fruit genotypes (numbers from pictures). 1: Anaranjada, 2: Zarca (2.5 GY 8/10), 3: Blanca de Castilla (2.5 GY 8/8), 4: Duraznillo Rojo (5RP 6/10) and 5: Tapón Blanco (5 GY 7/6). (C) Acidic-fruit genotypes (xoconostles, numbers from pictures). 6: Chiché (5 RP 5/9), 7: Rosa (5 RP 6/10), 8: Púrpura (5 RP 7/8), 9: Chacalote (5 RP 5/8) and 10: Pijo (5 RP 6/8).
El dendrograma mostró dos grupos muy diferenciados denominados ramas b y e, respectivamente. La rama o grupo b incluyó a la c donde se ubica el grupo control (P. rotundifolia y O. imbricata). El primero considerado ancestro de los nopales modernos y el segundo correspondiente a un genotipo del género Cylindropuntia, pariente cercano del género Platyopuntia. La rama o grupo d incluyó genotipos mayoritariamente silvestres, de fruto pequeño y sabor ácido del tipo xoconostle (Chiché, Blanco Crucen, Cuaresmeño Blanco, Ombligón y Canica). La mayor relación genética se encontró entre los llamados Canica y Meloncillo rosa con un coeficiente de similitud de 0,8.
El grupo e incluyó la mayoría de los genotipos silvestres y cultivados analizados (76), y con diferentes características morfológicas. De manera sobresaliente se separó el genotipo Rojo Pelón y posteriormente se formó la rama f que agrupó a los genotipos restantes. De manera general podemos diferenciar dos grandes grupos. Uno fue el grupo p que agrupó a la mayoría de los genotipos silvestres de fruto pequeño y sabor ácido, con la excepción de un genotipo cultivado (Reyna). El otro grupo fue el q que integró 20 genotipos identificados por su potencial frutícola y hortícola (Blanco Pelón, Pico chulo, Masuda Blanca, Milpa Alta, Verdulero Huarachón y Anaranjada), y 3 genotipos de fruto ácido de tipo xoconostle (Xoconostle Rojo Los Campos, Amarillo Grande y Manso, respectivamente). Al final se formó el grupo r con solo dos genotipos (Roja Cereza y Terciopelo) que se caracterizaron por presentar gran cantidad de espinas en el cladodio, y sus frutos fueron pequeños de color rojo (0,7). En términos generales, el dendrograma sugiere que los genotipos de frutoácido o dulce mantienen una estrecha relación genética; por ejemplo, los genotipos más dulce (Tapón rastrero) y más ácido (Pijo) con una cercanía de 0,71 (Fig. 3 A).
Descripción morfológica del fruto. De acuerdo al contenido de °Brix en el fruto (Tabla 2) fue posible diferenciar cuatro grupos principales debido a que este es un carácter distintivo a simple vista, y de rápida relación al genotipo en estudio (Fig. 3 B). El grupo I incluyó genotipos de fruto ácido (5,0-9,5 °Brix) conocidos comúnmente como xoconostles de origen silvestre y cultivado. En el Grupo II se ubicaron genotipos con valores de entre 10,3-17,9 °Brix. Estos dos grupos consideran los genotipos de importancia frutícola y hortícola. El Grupo III se caracterizó por genotipos de mayor tamaño y con un contenido de entre 14,1-17,5 °Brix. El Grupo IV se distinguió por dos genotipos silvestres, con el mayor contenido de °Brix y menor tamaño de fruto, menor contenido de mucilago y mayor contenido de espinas.
Tabla 2. Genotipos de nopal mexicanos Opuntia spp. agrupados de acuerdo a características del fruto.
Table 2. Fruit features on Mexican Opuntia genotypes.

** Genotipos cultivados (reproducción por cladodio y selección individual).
** Promedio ± SD de un solo genotipo; cuando dos valores se encuentran distantes se muestran rangos de múltiples genotipos (mínimo y máximo ± SD).
* Cultivated genotypes (reproduction by cladode and individual selection).
** Mean ± SD of a single genotype; when two values are far apart, ranges for multiple genotypes are reported (minimun and maximum ± SD).
DISCUSIÓN
Las caracterizaciones genéticas acompañadas de características morfológicas han permitido una mejor identificación de especies de importancia agronómica como se indica a continuación. Con el uso de marcadores moleculares tipo RAPDs, Tapia et al. (2005a) encontraron alta relación en 12 genotipos de piña (Ananas spp.) con algunas características morfológicas de la planta (peso, presencia de espinas, color y longitud de hoja, peso de fruto, resistencia a plagas y enfermedades), Azuara et al. (2008) obtuvieron un dendrograma que mostró una relación entre la agrupación genética y genotipos de ajo con menor número de dientes y mayor rendimiento. Melgoza-Villagómez et al. (2009) concluyeron que la respuesta de maduración del fruto se relacionó con la agrupación obtenida por los fragmentos amplificados al azar analizando 24 genotipos de durazno (Prunus persica). Sin embargo, varios estudios hallaron relación entre los perfiles genéticos y las características morfológicas utilizando marcadores moleculares tipo AFLPs. Por ejemplo, Tapia et al. (2005b) no encontraron relación genética con las características de las flores, las hojas y las frutos en mandarina (Citrus spp.). Resultados similares fueron obtenidos por Gutiérrez-Diez et al. (2009) al analizar el perfil genético con la morfología del fruto (tamaño, radio polar, diámetro ecuatorial y peso) en genotipos de aguacate. Con la información obtenida en este trabajo, consideramos que la estrecha relación observada entre genotipos silvestres y cultivados es debido a la interpolinización cruzada entre las especies (México es considerado centro de origen: Bravo-Hollis, 1978), con niveles de ploidías variantes de 2n, 4n, 6n y 8n (Rodríguez y Nava, 1998). Por lo tanto, la información generada no mostró relación directa de acuerdo a la taxonomía aceptada en Opuntias, basada en la morfología. La información es recurrente en diversos dendrogramas obtenidos por caracterizaciones genéticas (Wang et al., 1998; Labra et al., 2003; Guerrero-Muñoz et al., 2006; García-Zambrano, 2006; Luna-Páez et al., 2007). Los taxónomos consideran que el nopal continúa en una activa diferenciación genética (Granados y Castañeda, 1991). Por lo tanto, se requieren más estudios que detecten diferencias en las especies para crear una posible reclasificación de los mismos (Barrientos et al., 1993; Wang et al., 1998). Finalmente, nuestro estudio contribuyó a determinar el perfil genético y su agrupamiento de genotipos de nopal (Opuntia spp.) representativos de México, presentes en dos bancos nacionales de conservación de germoplasma de nopal.
AGRADECIMIENTOS
Los autores agradecen a la Red de Nopal del Sistema Nacional de Recursos Fitogenéticos para la Alimentación y la Agricultura, del Servicio Nacional de Inspección y Certificación de Semillas dependiente de la Secretaría de Agricultura Ganadería, Desarrollo Rural, Pesca y Alimentación (SINAREFI-SNICS-SAGARPA), al Consejo de Ciencia y Tecnología (CONACYT) por el apoyo económico otorgado al alumno Alexander Espinoza Sánchez a través de una beca para estudios de postgrado y a la División de Estudios de Posgrado e Investigación de la Dirección General de Educación Superior Tecnológica (DGEST-SEP) por todo el apoyo brindado.
REFERENCIAS
1. Azuara, H.L., H. Silos-Espino, C. Perales-Segovia, L J.F. Gómez, A.G.S. Alpuche y V.L.M. Macías (2008). Morphological and genetic characteristics allow the identification of a collection of garlic cultivars in the North-Central region of Mexico. Phyton, International Journal of Experimental Botany 77: 81-91. [ Links ]
2. Barrientos-Pérez, F., A. Muratalla-Lua y F. Barrientos-Priego (1993). New hybrids of Opuntia. En: Biotechnology for arid land plants. Institute The University of Texas at Austin. Austin, TX, U. S. A. Chapter 18. pp. 243-251. [ Links ]
3. Bravo-Hollis, H. (1978). Las Cactáceas de México. Vol 1 UNAM, México, pp. 235-351. [ Links ]
4. García-Zambrano, E., A. Gutiérrez-Díez, G. Salinas-García, E. Cárdenas-Cerda, R. Vázquez-Alvarado, F. Zavala-García y C. J. Martínez (2006). Classification and estimation or genetic diversity of nopal (Opuntia spp.) on the basis of phenotypic descriptors and genetic molecular markers. Phyton, International Journal of Experimental Botany 75: 125-135. [ Links ]
5. Gallegos-Vázquez, C., y C. Mondragón-Jacobo (2011). Cultivares selectos de tuna, de México al Mundo. Servicio Nacional de Inspección y Certificación de Semillas, Secretaría de Agricultura, Ganadería, Desarrollo Rural, Pesca y Alimentación (SNICS-SAGARPA) y Universidad Autónoma Chapingo (UACh). Chapingo, Méx. 159 p. [ Links ]
6. Gallegos-Vázquez, C., L. Scheinvar, C. Nuñez Colín y C. Mondragón Jacobo (2012). Morphological diversity of xoconostles (Opuntia spp.) or acidic cactus pears: a mexican contribution to functional foods. Fruits 67: 109-120. [ Links ]
7. Granados, D. y A. Castañeda (1991). Nopal. Historia, fisiología, genética e importancia frutícola Editorial Trillas, 1a Edición. México, D.F. 227 p. [ Links ]
8. Guerrero-Muñoz, M.P., M.H. Zavaleta, A. Barrientos-Priego, C. Gallegos-Vázquez, C. Núñez-Colín, E. Valadéz-Moctezuma y J. Cuevas-Sánchez (2006). Techniques for studying internal micromorphology from Opuntia hard seeds. Revista Fitotecnia Mexicana 29:37-43. [ Links ]
9. Gutiérrez-Díez, A., J. Martínez, E. García-Zambrano, L. Iracheta-Donjuan, J. Ocampo-Morales y I. Cerda-Hurtado (2009). Study of genetic diversity of native avocado in Nuevo León, México. Revista Fitotecnia Mexicana 32: 9-18. [ Links ]
10. Labra, M., F. Grassi, M. Bardini, S. Imazio, A. Guiggi, S. Citterrop, E. Banf, y S. Sgorbati (2003). Genetic relationships in Opuntia Mill. genus (Cactaceae) detected by molecular marker. Plant Science 165: 1129-1136. [ Links ]
11. LI-COR (2010). IRDye Fluorescent AFLP protocol for large plant genome analysis. Doc# 988-11159 http://biosupport.licor.com; última consulta: 20. IX. 2010. [ Links ]
12. Luna-Páez, A., E. Valadéz-Moctezuma, A. Barrientos-Priego y C. Gallegos-Vázquez (2007). Characterization of Opuntia spp. by means of seed with RAPD and ISSR Markers and its possible use for diferentiation. Journal of the Professional Association for Cactus Development 9: 43-59. [ Links ]
13. Melgoza-Villagómez, C., H. Silos-Espino, A. Nava-Cedillo, L. Valera-Montero, C. Perales-Segovia. M. Martínez, C. Martínez, A. Cruz y O. Paredes López (2009). Identification of peach genotypes (Prunus pérsica L. Batsch) in the North-Central Region, México. International Journal of Botany 5: 160-165. [ Links ]
14. Mondragón, J.C. y B.B. Bordelon (2002). Apomixis presence on crosses from Mexican nopal and their preliminary molecular identification. Revista Fitotecnia Mexicana 25: 247-252. [ Links ]
15. Mondragón-Jacobo, C. (2004). Nopal breeding: Updates at 2003 and perspectives. Horticultura 9: 97-114. [ Links ]
16. Munsell, A.H. (1977). Munsell color charts for plant tissues. Munsell Color Co. Baltimore, MD, E.E.U.U. 40 p. [ Links ]
17. Reyes-Agüero, J., J. Aguirre A. y J. Flores (2005). Morphological variation of Opuntia (Cactaceae) related to domestication in the Mexican Southern Uplands. Interciencia 30: 476-484. [ Links ]
18. Reyes-Agüero, J., J.R. Aguirre Rivera, F. Carlín Castelán y A. González-Durán (2009). Catálogo de principales variantes silvestres y cultivadas de Opuntia en la Altiplanicie Meridional de México. UASLP, SAGARPA y CONACYT San Luis potosí, S.L.P. México. 350 p. [ Links ]
19. Rodríguez, S.E. y A. Nava-Cedillo (1998). Nopal. Riqueza agroecológica de México. Subsecretaría de Educación e Investigación Tecnológicas. S.E.P. México, D.F. 160 p. [ Links ]
20. Rzedowski, J. (1978) Vegetación de México. Limusa, México, D. F. 432 p. [ Links ]
21. Scheinvar, L., G. Olalde-Parra, D. Olvera-Shule y C. Martínez-González (2010). Base de datos Especies Silvestres de Nopales Mexicanos. Laboratorio de Cactología, Jardín Botánico IB-UNAM. [ Links ]
22. Tapia, C.E., H. Guillen y M. Gutiérrez (2005a). Genetic Characterization of pineapple (Ananas spp.) accessions by RAPD and ISSR. Revista Fitotecnia Mexicana 28: 187-194. [ Links ]
23. Tapia, C.E., E.M.A. Gutiérrez, M.L. Warburton, V.A. Santacruz y M.A. Villegas (2005b). Characterization of mandarin (Citrus spp.) using morphological and AFLP markers. Interciencia 30: 687-693. [ Links ]
24. Tel-Zur, N., S. Abbo, D. Myslabodski y Y. Mizrahi (1999). Modified CTAB procedure for DNA isolation from epiphytic cacti of the genera Hylocerus and Selenicereus (Cactaceae). Plant Molecular Biology Reporter17: 249-254. [ Links ]
25. Vos, P., R. Hogers, M. Bleeker, M. Reijans, T. Van De Lee, M. Hornes, A. Frijters, J. Pot, J. Peleman, M. Kuiper y M. Zabeau (1995). A new technique for DNA fingerprinting. Nucleic Acids Research 23: 4407-4414. [ Links ]
26. Wang, X., P. Felker, D., M. Burow y A. Paterson (1998). Comparison of RAPD marker patterns to morphological and physiological data in the classification of Opuntia accessions. Journal of the Professional Association for Cactus Development 3: 3-14. [ Links ]














