Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Phyton (Buenos Aires)
versión On-line ISSN 1851-5657
Phyton (B. Aires) vol.83 no.2 Vicente López dic. 2014
ARTÍCULOS ORIGINALES
Predicción de rendimiento de fruto de híbridos de tomate con huellas genómicas
Prediction of tomato hybrid performance with genomic markers
Hernández-Ibáñez L, J Sahagún-Castellanos, JE Rodríguez-Pérez, MG Peña-Ortega, LM Rodríguez-Martínez
Instituto de Horticultura. Departamento de Fitotecnia. Universidad Autónoma Chapingo. Km. 38,5 Carretera México-Texcoco. Chapingo, Estado de México. C.P. 56230. México.
Address Correspondence to: Jaime Sahagún Castellanos, e-mail: jsahagunc@yahoo.com.mx
Recibido / Received 26.XII.2012.
Aceptado / Accepted 18.IV.2013.
Resumen. En muchos países, el tomate (Solanum lycopersicum L.) es la especie hortícola más importante, pero la semilla de variedades sobresalientes es cara. En el mejoramiento genético mediante hibridación, el número de cruzas simples posibles es demasiado alto con pocas líneas. Por ejemplo, con 60 líneas el número de híbridos posibles es 1770. Esto hace inaccesible (en términos de costos), y aún imposible efectuar una evaluación experimental adecuada. Situaciones como ésta generan la necesidad de disponer de métodos de predicción del rendimiento de fruto de los híbridos. Este estudio se desarrolló para evaluar un método de predicción del rendimiento de fruto de híbridos basado en huellas genómicas, teoría de los modelos mixtos y en la evaluación experimental de una muestra de híbridos. Se estudiaron 97 líneas con 17 iniciadores ISSR, el coeficiente de Jaccard y el método de la varianza mínima de Ward. Estas líneas se clasificaron en cuatro grupos, y se eligieron dos de ellos denominados X e Y para predecir el rendimiento de fruto. Con 5 y 8 líneas de X e Y, respectivamente, se generaron 40 cruzas intergrupales y se evaluó su rendimiento por planta. Se desarrolló una ecuación de predicción y se predijo el rendimiento de fruto según el método del mejor predictor lineal insesgado o BLUP (best linear unbiased prediction). Con m = 50, 100, 200, 400, 600, 1000 muestras aleatorias independientes de tamaños de n = 5, 10, 15, 20, 25, 30 se hicieron 40 híbridos. Con 35 de estos híbridos se predijo el rendimiento de los 40-n = 35, 30, 25, 20, 15, 10, 5 híbridos restantes y se calculó el coeficiente de correlación entre rendimientos predichos y observados. Las correlaciones promedio fluctuaron entre 0,448 y 0,791 (a=0,01). Los resultados obtenidos sugieren que la predicción del rendimiento de híbridos en tomate con BLUP podría ser exitosa.
Palabras clave: Solanum lycopersicum L.; Mejor predictor lineal insesgado; Marcadores moleculares; ISSR.
Abstract. In many countries, tomato (Solanum lycopersicum L.) is the most important horticultural species, but good variety seeds are expensive. In hybrid breeding, the number of possible single crosses is too large with only a few lines. With 60 lines, for example, 1770 single-cross hybrids can be formed. This makes it expensive and even impossible to conduct an adequate experimental evaluation. These cases require the availability of methods for predicting hybrid performance. This study was designed to evaluate a method to predict fruit yield in tomato hybrids based on genomic fingerprints, the theory of mixed models, and the experimental evaluation of a sample of hybrids. Ninety seven inbreds were studied with 17 ISSR primers, the Jaccard coefficient and the minimum variance method of Ward. The inbreds were assigned to four groups. Two of them named X and Y were chosen to predict performance, with 5 and 8 inbreds, respectively; their forty intergroup single-crosses were generated, and their fruit yield determined thereafter. The performance of crosses was predicted with the best linear unbiased prediction method (BLUP). With m = 50, 100, 200, 400, 600, 1000 sets of n = 5, 10, 15, 20, 25, 30, it was possible to obtain 40 hybrids. Thirty five of them were randomly selected and the performance of the remaining 40-n crosses was predicted. The correlation coefficients between predicted and measured yields were calculated. The average correlation coefficients fluctuated between 0.448 and 0.792 (a=0.01). Results suggest that the prediction of hybrid fruit tomato performance might be successful using the method BLUP.
Keywords: Solanum lycopersicum L.; Best linear unbiased prediction, BLUP; Molecular markers; ISSR.
INTRODUCCIÓN
En México, el tomate (Solanum lycopersicum L.) es una especie hortícola muy importante por la superficie sembrada, por el valor de la producción y por las divisas generadas. En su cultivo, el empleo de híbridos es determinante para obtener altos rendimientos de fruto. Sin embargo, el proceso de selección de líneas puras como progenitores de híbridos sobresalientes usualmente requiere: 1) identificación de grupos heteróticos (Melchinger y Gumber, 1998); 2) formación de líneas de los diferentes grupos; 3) realización de cruzamientos intergrupales, 4) evaluación de rendimiento y características agronómicas de los híbridos generados, y 5) selección de los mejores híbridos.
Típicamente, menos del 1% de los productos de cruzamientos examinados de maíz (Zea mays L.) llegan a ser híbridos comerciales (Bernardo, 1996). Esto implica la necesidad de evaluar cientos o miles de cruzamientos simples. Sin embargo, si el número de líneas elite es grande, el número de cruzamientos simples puede ser inaccesible (en términos de costos) e imposible de evaluar experimentalmente en forma adecuada. Afortunadamente, es posible reducir la cantidad de trabajo y costos mediante la predicción de prácticamente todos los híbridos después de la formación y evaluación experimental de una pequeña muestra de ellos. Existe un método para predecir los híbridos que no son parte de la muestra. Esto es posible con información generada experimentalmente a partir de los híbridos que forman la misma y las coancestrías de las líneas estimadas en base a sus huellas genómicas (Henderson, 1985; Bernardo, 1994; Gbur et al., 2012). Este método produce "los mejores predictores lineales insesgados" (BLUP, por su sigla en inglés: Best Linear Unbiased Prediction) mediante una ecuación de predicción, para cuya construcción se pueden utilizar huellas genómicas para estimar los coeficientes de coancestría mencionados (Bernardo, 1993, 1995).
En maíz (Zea mays L.) se ha predicho el rendimiento de grano de cruzas simples con la combinación de marcadores moleculares RFLPs y el método del mejor predictor lineal insesgado (Bernardo, 1994; 1996), y con la combinación de marcadores microsatélites y BLUP (Balestre et al., 2010). Los coeficientes de correlación entre rendimientos observados y predichos fluctuaron entre 0,426 y 0,762, y entre 0,55 y 0,70, respectivamente. En tomate, Mirshamsi et al. (2008) utilizaron marcadores RAPDs para estimar distancias genéticas entre progenitores, y determinar la correlación entre éstas y el rendimiento de fruto de los híbridos producidos por sus cruzas. Sin embargo, los bajos valores de los coeficientes de correlación obtenidos (r<0,40) sugieren que este método es de poca utilidad práctica.
Los objetivos de esta investigación fueron: (1) generar una ecuación de predicción de rendimiento de fruto de híbridos de tomate generados con líneas de grupos diferentes, identificados a partir de huellas genómicas basadas en ISSRs, y (2) verificar la eficiencia de la ecuación de predicción de rendimiento, mediante la evaluación experimental de los híbridos predichos.
MATERIALES Y MÉTODOS
La presente investigación se realizó de febrero de 2010 a agosto de 2011, y consistió de tres fases: (1) Selección de líneas progenitoras de tomate y formación de grupos mediante marcadores genéticos moleculares (ISSR), (2) Evaluación de rendimiento de fruto de cruzas intergrupales, y (3) Predicción de rendimiento de cruzas mediante BLUP y calibración del método.
Selección de líneas progenitoras mediante ISSRs. Se evaluaron 92 líneas homocigóticas avanzadas provenientes de la selección de poblaciones segregantes generadas a partir de cruzamientos entre híbridos comerciales de tomate, y 5 líneas seleccionadas de poblaciones de tomate nativo de México.
Las 97 líneas se sembraron en bandejas de poliestireno de 200 cavidades con turba como sustrato. Entre 40 y 50 días después de la siembra se obtuvieron muestras de tejido de hojas tiernas, verdes y sin daño aparente. Cada muestra se calentó a 65 °C en bloque térmico con 700 µL de buffer de extracción (Tris HCl 100 mM, EDTA 50 mM, NaCl 500 mM, 2-mercaptoetanol 10 mM, SDS 1,3%, pH 8.0), contenidos en un microtubo de 1,5 mL. Las hojas de plántulas se lavaron con etanol al 70% y se secaron con toallas de papel. Se (1) colocaron 0,3 g de hoja en un mortero de porcelana, (2) agregó nitrógeno líquido, y (3) molió hasta obtener un polvo fino que fue transferido al microtubo, y se agitó hasta homogenizar.
Nuevamente se (1) calentó a 65 °C durante 10 minutos en bloque térmico, (2) agregaron 200 µL de acetato de potasio 5M, (3) enfrió en el congelador durante 60 min, y (4) centrifugó por 20 min a 20000 g. El sobrenadante se (1) transfirió a un microtubo con 600 µL de isopropanol frio, que se mantuvo en el congelador por 60 min para precipitar el ADN, (2) centrifugó a 12000 g durante 10 min, y (3) decantó el sobrenadante. A la pastilla se agregaron 500 µL de solución para disolver (Tris HCl 10 mM, EDTA 1 mM, NaCl 10 mM, pH 8.0) y se dejó en refrigeración durante una noche.
Una vez re-suspendido el ADN se (1) agregaron 500 µL de la mezcla cloroformo-alcohol isoamílico (24:1), (2) homogeneizó mediante agitación, y (3) centrifugó a 12000 g durante 4 min. La fracción acuosa se separó en un tubo nuevo, se agregaron 4 µL de ARNasa e incubó a 37 °C por una hora. EL ADN se volvió a precipitar con 75 µL de acetato de sodio 3M y 500 µL de isopropanol frío, se agitó por inversión e introdujo al congelador por 2 h. Se (1) centrifugó a 12000 g durante 5 min, (2) eliminó el sobrenadante, y (3) lavó la pastilla con 500 µL de etanol al 70% (grado reactivo). Nuevamente se centrifugó, eliminó el sobrenadante y secó la pastilla a temperatura ambiente. Finalmente, ésta se disolvió con 50 µL de TE y se almacenó a 4 °C.
Para cuantificar la pureza del ADN se colocaron 594 µL de agua destilada y 6 µL de la solución de ADN en un microtubo de 1,5 mL. La absorbancia de la solución obtenida se determinó a 260 nm (A260) con un espectrofotómetro. La concentración de ADN se determinó con la fórmula: ADN µg/µL = (A260 x 50 µg/mL x 2.0) / 1000. Finalmente, se uniformaron las muestras a una concentración de 100 µg/µL y se prepararon soluciones de trabajo con 10 µg/µL de ADN.
La calidad del ADN se verificó en un gel de agarosa al 0,8% y TAE al 0,5% de 4 mm de grosor. En cada pozo se depositaron 10 µL de la solución de trabajo con ADN. La electroforesis se realizó por dos horas. El gel se tiñó con solución de bromuro de etidio a una concentración de 0,5 µg/mL durante 20 min. El gel se fotografió con fotodocumentador modelo MiniBIS Pro (DNR Bio-Imaging Systems). El ADN de muestras que generaron manchas demasiado tenues u obscuras se concentró o diluyó, respectivamente, y se repitió el gel de calidad. Este proceso se repitió hasta que se generaron manchas uniformes. En la amplificación y separación de fragmentos mediante PCR se emplearon 17 iniciadores ISSR; las temperaturas de alineamiento se muestran en la Tabla 1. La reacción de amplificación se realizó con un volumen de 25 µL; la mezcla de reacción consistió de: 5,2 µL de agua grado Biología Molecular; 10,0 µL de dNTP's [500 µM]; 2,5 µL de bufer [10 x]; 1,5µL de MgCl2 [50 mM]; 3,0 µL de iniciador ISSR [10 ng/µL]; 0,3 µL de enzima Taq DNA polimerasa [5 u/µL], y 2,5 µL de ADN [10 ng/µL].
Tabla 1. Iniciadores ISSR utilizados en la caracterización de la divergencia genética de 97 líneas de tomate: temperatura de alineamiento empleada (Ta); número total de bandas amplificadas (NTBA); número de bandas polimórficas (NBP), y porcentaje de polimorfismo (PP).
Table 1. ISSR primers employed to measure the genetic divergence of 97 tomato lines (Iniciador): alignment temperature (°C); total number of amplified bands (NTBA); number of polymorphic bands (NBP) and polymorphism percentage (PP).

zBases: A= adenina, G= guanina, C= citosina, T= timina, Y= citosina + timina.
La PCR se llevó a cabo en un termociclador modelo FT-C41H2D marca TECHNE. Se utilizó el siguiente programa: (1) desnaturalización inicial a 93 °C por 1 min; (2) cuarenta ciclos, cada uno con: 20 s a 93 °C, 60 s a la temperatura del iniciador probado, y 20 s a 72 °C, y (3) una extensión final a 72 °C por 6 min. La muestra se enfrió a 10 °C. Los productos finales se llevaron a electroforesis en gel de agarosa al 2% con TAE 0,5%. Posteriormente se realizó la tinción con bromuro de etidio y fotodocumentación.
A partir de la matriz de datos binarios obtenida de las huellas genéticas, se construyó el coeficiente de similitud de Jaccard (Srs) para cada par de genotipos (Khattree y Naik, 2000). Con la matriz de disimilitud (d = 1 - Srs) se realizó el agrupamiento mediante el método de varianza mínima de Ward con el programa Infostat®. El resultado fue la formación de cuatro grupos de los que se eligieron dos, considerados genéticamente diferentes y a los que seguidamente se los denominó X e Y.
Evaluación de rendimiento de fruto de cruzas intergrupales. En base a una evaluación previa, se seleccionaron 13 líneas de alto rendimiento de fruto; cinco pertenecientes al grupo X (líneas 58, 77, 76, 92 y 93), y los otros ocho pertenecientes al Y (líneas 10, 14, 25, 28, 42, 43, 44 y 52). Éstas se sembraron en julio del 2010, y se realizaron los 40 (5x8) cruzamientos directos intergrupales.
Posteriormente, éstos se sembraron en bandejas de poliestireno de 200 cavidades usando turba como sustrato con el propósito de determinar el rendimiento de fruto de los cruzamientos. El trasplante a un sistema hidropónico se hizo 35 días después de la siembra en invernáculo. Se usaron bolsas de polietileno negro con capacidad de 18 litros y sustrato de espuma volcánica (tezontle rojo). La solución nutritiva utilizada fue la propuesta por Cadahia (2000); la cantidad aplicada varió de acuerdo con la etapa fenológica y las condiciones climáticas. El sistema de conducción fue de un tallo y la densidad de plantación correspondió a 3,7 plantas/m2.
La unidad experimental consistió en dos macetas, cada una con dos plantas. Se empleó un diseño experimental de bloques completos al azar con tres repeticiones. El rendimiento de fruto por planta se evaluó en los primeros cuatro racimos.
Predicción de rendimiento de cruzas mediante BLUP y evaluación del método. Inicialmente se calcularon los coeficientes de coancestría con base en ISSR de acuerdo con la expresión (Bernardo, 1993):
fxx' = [Sxx' - 1/2(Sxy + Sx' y)]/[1-1/2(Sxy + Sx' y)]
Donde:
fxx coeficiente de coancestría entre las líneas x y x' del grupo X; Sxx' = proporción de variantes ISSR que comparten x y x'; Sxy (o Sx'y) = proporción promedio de variantes ISSR comunes en x (o en x') y en una serie de líneas del grupo heterótico Y.
El modelo para el rendimiento de fruto (y) fue el utilizado por Bernardo (1994):
y=Xß+Z1ax + Z2ay + Z+e
Donde, en su acepción general, ß es un vector que contiene los efectos de los ambientes de evaluación; ax (ay) es el vector que contiene los efectos de aptitud combinatoria general (ACG) de las líneas del grupo X (Y); Z es el vector de los efectos de aptitud combinatoria específica (ACE); e es el vector de residuos, y X, Z1, Z2 y Z son matrices cuyos elementos son 0 ó 1 para denotar ausencia o presencia de efectos.
La covarianza (Cov) entre los híbridos intergrupales x·y y x'·y' se expresó en base a un modelo de k loci, y siguiendo la simplificación informada por Melchinger (1988). Según esta simplificación:
Cov(x·y,x'·y')= fxx'VA(X/Y) + fyy'VA(Y/X) + fxx'f yy'VD(XY)
Donde VA(X/Y) = Varianza aditiva del alelo x a través de los k loci de y involucrados, y VD(XY) = varianza de dominancia a través de los k loci de los pares alélicos formados cada uno por un alelo de X y otro de Y.
Las estimaciones de máxima verosimilitud restringida fueron obtenidas mediante iteración a partir de sendas ecuaciones para VE, la varianza no genética, VA(X/Y), VA(Y/X) y VD(XY) (Henderson, 1985; Bernardo, 1994; Gbur et al., 2012). Además de la predicción, las estimaciones de estas varianzas se utilizaron para estimar heredabilidades.
La predicción de los rendimientos de fruto de los híbridos se basó en la teoría de los modelos mixtos que produce predictores que son lineales, insesgados y de varianza mínima (BLUP, por sus siglas en inglés: Best Linear Unbiased Predictors). Los rendimientos de los híbridos supuestamente desconocidos fueron predichos en base a los rendimientos observados de los híbridos restantes, llamados híbridos predictores. De acuerdo con Bernardo (1994), el rendimiento de estos híbridos (yp) se expresó como:
yp = (Z'Z)-1 Z' (y-Xß)
Por otra parte, el vector formado con los rendimientos predichos de los híbridos "desconocidos" (yd) fue calculado según la expresión:
yd = CV-1 yp
Donde C es la matriz m x n cuyo ij-ésimo elemento es la covarianza genética entre el i-esimo híbrido desconocido y el j-esimo híbrido predictor, y V es la matriz n x n de las covarianzas de los n híbridos predictores. Los elementos de C y los que van fuera de la diagonal de V se calcularon según la expresión de la covarianza entre un híbrido desconocido y un híbrido predictor, en términos de coancestrías y varianzas genéticas.
Para evaluar el método de estimación en términos de tamaño de muestra, se trabajó con conjuntos de n híbridos predictores (n = 5, 10, 15, 20, 25, 30 y 35), seleccionados al azar, de los 40 posibles. Para verificar la estabilidad de las predicciones, para cada número de híbridos predictores (n) se realizaron m (m = 50, 100, 200, 400, 600, 1000) muestreos aleatorios, y se predijo el rendimiento de fruto de los 40-n híbridos restantes en cada uno de los m casos. Posteriormente, para cada valor de n se calcularon las correlaciones entre los 50, 100, 200, 400, 600 y 1000 rendimientos observados y los predichos correspondientes. Para cada grupo de correlaciones, se determinó siéstas eran homogéneas (Steel et al., 1997).
Finalmente, se predijo el rendimiento de los 567 híbridos intergrupales restantes con las 21 líneas del grupo X y las 27 del grupo Y a partir de los 40 híbridos predictores obtenidos. Con el fin de estimar el potencial genotécnico de estas 48 líneas, se calculó para cada una de ellas el promedio de todos los híbridos predichos en que ésta participó como progenitora (21 o 27).
Para hacer los cálculos necesarios para predecir se construyó un programa en el módulo IML del paquete estadístico SAS.
RESULTADOS Y DISCUSIÓN
Selección de líneas progenitoras mediante ISSR. Se usaron 17 iniciadores ISSR para caracterizar la divergencia genética de 92 líneas avanzadas y cinco materiales nativos. Se amplificaron 116 bandas, de las que 107 fueron polimórficas. El número de bandas por iniciador varió entre 3 y 13, con un promedio de 6,82 (Tabla 1). Estos datos son bajos comparados con los de Aguilera et al. (2011), que muestran la amplificación de 9 a 22 bandas por iniciador, con un promedio de 14,4. Sin embargo, estos autores reportaron un nivel reducido de polimorfismo de 34%, con un promedio de 5,3 bandas polimórficas por iniciador. En el presente trabajo, se amplificaron 6,29 bandas polimórficas por ISSR con 92,2% de polimorfismo general (mínimo = 33, máximo = 100%.
Los iniciadores en cuya secuencia de aminoácidos se incluyó la adenina (A) en combinación con guanina (G) y/o citosina (C) (GA, AG, CA, AC y AGC) generaron el mayor número de bandas, y sobre todo bandas polimórficas (Tabla 1). Por esta razón, los iniciadores identificados como ISSR, previamente reportados por Bojinov y Danailov (2009), dieron mejores resultados que los UBC (University of British Columbia).
A partir de los 107 marcadores polimórficos obtenidos, el coeficiente de Jaccard (distancia genética) y el análisis de agrupamiento, se generó el dendrograma de la Figura 1. En dicho dendrograma se definieron cuatro conjuntos de líneas con una altura de corte de 0,08. La distancia genética promedio fue de 0,304. La mayor disimilitud (0,64) ocurrió entre las líneas 39 y 50; la línea 39 también registró el segundo valor más disímil (0,62) respecto a las líneas 47, 56, 62 y 71. En contraste, las líneas con mayor parecido genético fueron la 54 y la 53 (disimilitud de 0,05), lo que sugiere que comparten el mismo origen genético. También se observaron pares de líneas con alta similitud: 14 y 16, 67 y 72, así como los silvestres 4 y 5, con disimilitud de 0,08.

Fig. 1. Dendrograma de disimilaridad genética de 97 líneas de tomate.
Fig. 1. Genetic dissimilarity dendrogram of 97 tomato lines.
El grupo 1, el más divergente, contuvo 23 líneas. El grupo 2 fue el más pequeño con 13 genotipos en los que se incluyen los cinco nativos y la línea 39. Los grupos 3 y 4 constaron de 35 y 26 líneas, respectivamente. En cuanto a rendimiento, los grupos 2, 3 y 4 incluyeron genotipos muy productivos (6, 9 y 5 líneas, respectivamente); el grupo 1 aglutinó genotipos de medio a bajo rendimiento.
No se detectó ninguna asociación de los grupos generados con marcadores moleculares, con alguna característica fenotípica. En cada grupo se encontró diversidad en crecimiento (determinado e indeterminado), y tipo (riñón, cherry, saladete y bola) y color de fruto (amarillo, naranja y rojo).
Los grupos 3 y 4, los de mayor cantidad de líneas, fueron seleccionados para predecir el rendimiento de híbridos de tomate. Arbitrariamente, el grupo 4 se denominó X y de él se usaron cinco líneas (58, 76, 77, 92 y 93). El grupo 3 fue llamado Y, del cual se usaron ocho líneas (10, 14, 25, 28, 42, 43, 44 y 52). Con estas líneas se hicieron los 40 híbridos intergrupales.
Evaluación de rendimiento de fruto de cruzas intergrupales. El rendimiento de fruto por planta de los 40 híbridos intergrupales producidos se presenta en la Tabla 2. El promedio general de producción fue de 2,316 kg/planta, y varió entre 1,329 y 3,300 kg/planta.
Tabla 2. Rendimiento de fruto (kg/planta) de los híbridos intergrupales entre cinco líneas del grupo X y ocho líneas del grupo Y.
Table 2. Fruit yield (kg/plant) of intergroup hybrids between five lines from group X and eight lines from group Y.
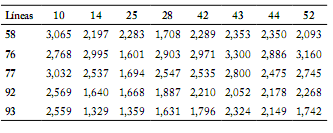
Del grupo X, las líneas de mayor rendimiento fueron la 76 (2,82 kg/planta) y la 77 (2,55 kg/planta), y la de menos rendimiento fue la 93 (1,86 kg/planta). Para el grupo Y, las líneas de mayor rendimiento fueron la 10 y 43 (2,80 y 2,57 kg/planta, respectivamente), y la línea de menor producción fue la 25 (1,72 kg/planta).
Predicción de rendimiento de cruzas mediante BLUP's y calibración del método. Con la información obtenida con los marcadores ISSR se calcularon los coeficientes de coancestría de cada par de líneas del grupo X, y de cada par de líneas del grupo Y. En el grupo X, las coancestrías fueron relativamente bajas (promedio = 0,1429); las mismas variaron desde cero [en los pares de líneas (58,92), (77,92) y (77,93)] hasta 0,3509 [en el par (76,77)]. Con el método utilizado para calcular las coancestrías, se encontraron valores negativos; como por definición una coancestría no puede ser negativa, se asignó el valor cero.
Las coancestrías fueron más altas en el grupo Y (promediaron 0,3098). Los registros fluctuaron entre cero [en los pares de líneas (10,43) y (10,52)] y 0,6315 [en el par (42,43)]. Las líneas 42, 43 y 44, que se obtuvieron de la misma cruza, mostraron coeficientes de coancestría altos y homogéneos en sus diferentes combinaciones, lo que es consistente con su origen.
Las estimaciones de las varianzas genética y no genética, con marcadores ISSR mediante BLUP fueron: 0,1812 para la varianza aditiva de líneas del grupo X; 0,1957 para la varianza aditiva de líneas del grupo Y; 0,1061 para la varianza de dominancia y 0,0128 para la varianza no genética. Con estos valores, las estimaciones de las varianzas genética y fenotípica fueron 0,4830 0,4873, respectivamente. La estimación de la heredabilidad en sentido estricto (h2) del rendimiento de fruto fue de 0,7780, y la heredabilidad en sentido amplio fue de 0,9991. El valor estimado de h2 en esta investigación difiere del publicado por Dordevic et al. (2010, h2 = 0,451). Además de las variaciones aleatorias del error, esta discrepancia se puede atribuir a diferencias entre materiales genéticos y ambientes. Wessel-Beaver y Scott (1992) obtuvieron estimaciones de h2 de 0,65 y 0,81 para el rendimiento de una misma población de tomate cultivada en Puerto Rico y Florida, respectivamente. Sin embargo, la heredabilidad en sentido amplio coincide con los valores informados por Haydar et al. (2007) y Dordevic et al. (2010), quienes obtuvieron estimaciones de 0,9767 y 0,9888, respectivamente. Por otra parte, las heredabilidades obtenidas en este estudio son argumentos que permiten predecir éxito en un programa de mejoramiento basado en selección.
Correlaciones entre rendimientos observados y predichos. Para cada valor de n (n = 5, 10, 15, 20, 25, 30 y 35), se obtuvieron m muestras aleatorias independientes de híbridos predictores (m = 50, 100, 200, 400, 600, 1000). Para cada combinación de n y m, se calcularon las m correlaciones entre los 40-n híbridos predichos y los observados (Tabla 3). Todas las correlaciones fueron significativas (a=0,01) y fluctuaron entre 0,448 y 0,791. Las correlaciones se incrementaron siempre que se incrementó n, aunque los incrementos cada vez fueron menores, y fueron poco claros a partir de n = 25. Además con n = 20, las desviaciones estándar de las m correlaciones en cada caso (m = 50, 100, 200, 400, 600, 1000) fueron las más pequeñas en relación a sus contrapartes para diferentes valores de n, excepto en la combinación n = 25 y m = 200.
Tabla 3. Correlaciones (?OP) promedio (x1000) entre rendimientos de fruto (kg/planta) observados y predichos de 40-n híbridos de tomate y sus desviaciones estándar (S?). Los datos corresponden a m muestras aleatorias independientes de n de los 40 híbridos predictores.
Table 3. Average correlations (?OP; x1000) between observed and predicted fruit yields (kg/plant) of 40-n tomato hybrids, and their standard deviations (S?). Data are from m independent random samples of n out of the 40 predictor hybrids.

Los resultados anteriores sugieren que con 20 híbridos predictores (n = 20) se obtuvo la mayor estabilidad en las predicciones. Aunque la magnitud de las correlaciones siempre aumentó cuando aumentó n, los incrementos fueron de poca consideración. En un escenario de costos, un incremento en n implica una mayor inversión. Por el contrario, no se produjo una tendencia en los valores de los coeficientes de correlación a partir de m = 50.
En los casos n = 5 y n = 10, la prueba de ji cuadrada no detectó homogeneidad de correlaciones para ningún valor de m (a=0,01), pero sí lo hizo con n ≥ 15. Esto implica que a partir de este valor de n, las correlaciones consignadas representan debidamente a las m correlaciones (m = 50, 100, 200, 400, 600, 1000).
Las correlaciones obtenidas son, en general, similares a las reportadas por Bernardo (1994). Este autor utilizó RFLPs en maíz, y las correlaciones más baja y más alta fueron 0,695 y 0,800, respectivamente (a=0,01).
Las altas correlaciones obtenidas sugieren que el método utilizado para predecir el rendimiento de los híbridos es prometedor. De todas maneras, la extrapolación debe hacerse con reservas ya que la correlación entre híbridos observados y predichos aumenta conforme se incrementa el número de híbridos predictores. Sin embargo, esto ocurre hasta cierto nivel ya que después se corre el riesgo de tener correlaciones (?) más pequeñas e incluso negativas. Además, los resultados expuestos son de un conjunto relativamente pequeño de cruzas simples. Bernardo (1996) aplicó esta metodología en una escala mayor (16 combinaciones de 9 grupos heteróticos en maíz), y las correlaciones que obtuvo entre rendimientos predichos y observados fueron ligeramente más bajas (desde 0,426 hasta 0,762).
De los rendimientos predichos para los 567 híbridos no realizados entre 21 líneas del grupo X y 27 del grupo Y, 143 superaron a la media de híbridos observados (2,33 kg/planta). Los híbridos más prometedores fueron: 4x64, 3x75, 3x64, 4x75 y 3x54, con rendimientos predichos de 2,558; 2,550; 2,549; 2,545 y 2,545 kg/planta, respectivamente.
La Tabla 4 muestra el promedio de los rendimientos predichos de los híbridos en que participaron cada una de las 21 líneas X, y cada una de las 27 líneas Y. Sólo tres líneas del grupo X se asociaron a híbridos cuyo rendimiento de fruto superó a la media estimada con BLUP. Estas líneas fueron: 3, 4 y 61 con 2,41; 2,39 y 2,35 kg/planta, respectivamente. En el grupo Y, 7 líneas superaron la media. Los mayores rendimientos correspondieron a los híbridos cuyos progenitores fueron las líneas 64, 11 y 54 (2,41; 2,37 y 2,37 kg/planta, respectivamente). Estos resultados sugieren que existen líneas que tienen potencial genotécnico para participar como progenitores de híbridos sobresalientes, o como base genética para un programa de selección, con miras a obtener mejores líneas.
Tabla 4. Promedio de rendimiento de fruto (kg/planta) predichos de híbridos que tienen una línea en común.
Table 4. Predicted average fruit yields of hybrids which have a common line (kg/plant).
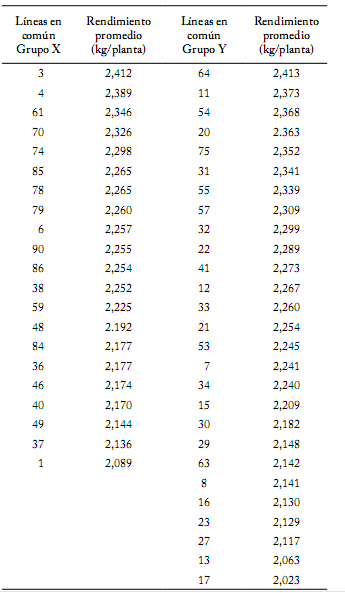
CONCLUSIONES
Los coeficientes de correlación entre rendimientos de fruto observados y predichos con BLUP, de híbridos formados con cruzas intergrupales de dos grupos, fluctuaron entre 0,448 y 0,791 (a=0,01). Estos resultados sugieren que este método puede tener utilidad fitotécnica. Además, la ecuación de predicción permite identificar aquellas líneas que muestran alta aptitud combinatoria general en cruzas intergrupales, lo que les da valor como progenitores en programas de mejoramiento por selección.
REFERENCIAS
1. Aguilera, J.G., L.A. Pessoni, G.B. Rodriguez, A.Y. Elsayed, D.J.H. Da Silva y E.G. De Barros (2011). Genetic variability by ISSR markers in tomato (Solanum lycopersicon Mill.). Revista Brasileira de Ciências Agrárias 6: 243-252. [ Links ]
2. Balestre, M. R.G. Von Pinho y J.C. Souza (2010). Prediction of maize single-cross performance by mixed linear models with microsatellite marker information. Genetics and Molecular Research 9: 1054-1068. [ Links ]
3. Bernardo, R. (1993). Estimation of coefficient of coancestry using molecular markers in maize. Theoretical and Applied Genetics 85: 1055-1062. [ Links ]
4. Bernardo, R. (1994). Prediction of maize single-cross performance using RFLPs and information from related hybrids. Crop Science 34: 20-25. [ Links ]
5. Bernardo, R. (1995). Genetic models for predicting maize singlecross performance in unbalanced yield trial data. Crop Science 35: 141-147. [ Links ]
6. Bernardo, R. (1996). Best linear unbiased prediction of maize singlecross performance. Crop Science 36: 50-56. [ Links ]
7. Bojinov, B.M. y Zh.P. Danailov (2009). Applicability of ISSRs for genotype in a tomato breeding collection. Acta Horticulturae 830: 63-70. [ Links ]
8. Cadahia, C. (2000). Fertirrigación, Cultivos Hortícolas y Ornamentales. 2da. Edición. Ediciones Mundi-prensa. Madrid, España. 475 p. [ Links ]
9. Dordevic, R., B. Zecevic, J. Zdravkovic, T. Zivanovic y G. Todorovic (2010). Inheritance of yield components in tomato. Genetika 42: 575-583. [ Links ]
10. Gbur, E.E., W.W. Stroup, K.S. Mc Carter, S. Durham, L.J. Young, M. Cristman, M. West y M. Kramer (2012). Analysis of Generalized Linear Mixed Model in the Agricultural and Natural Resources Sciences. ASA, SSSA, CSSA, Inc. Madison, Wisconsin, USA. 238 p. [ Links ]
11. Haydar, A., M.A. Mandal, M.B. Ahmed, M.M. Hannan, R. Karim, M.A. Razvy, U.K. Roy y M. Salahin (2007). Studies on genetic variability and interrelationship among the different traits in tomato (Lycopersicum esculentum Mill.). Middle-East Journal of Scientific Research 2: 139-142. [ Links ]
12. Henderson, C.R. (1985). Best linear unbiased prediction of nonadditive genetic merits in noninbred populations. Journal of Animal Science 60: 111-117. [ Links ]
13. Khattree, R. y D.N. Naik (2000). Multivariate Data Reduction and Discrimination with SAS Software. SAS Institute Inc. Cary, N.C., U.S.A. [ Links ]
14. Melchinger, A.E. (1988). Means, variances, and covariances between relatives in hybrids populations with disequilibrium in the parent populations. En: B.S. Wei, E.J. Eeisen et al. (ed.). Proc. 2nd Int. Conf. Quantit. Genet. Raleigh, NC. 31 May - 5 June 1987. Sinnauer Asoc. Sunderland, MA, pp 400-405. [ Links ]
15. Melchinger, A.E. y R.K. Gumber (1998). Overview of heterosis and heterotic groups in agronomic crops. En: Concepts and Breeding of Heterosis in Crop Plants. K.R. Lamkey y J. E. Staub (ed.). CSSA, Madison, WI, pp 29-44. [ Links ]
16. Mirshamsi, A., M. Farsi, F. Shahriari y H. Nemati (2008). Use of random amplified polymorphic DNA markers to estimate heterosis and combining ability in tomato hybrids. Pakistan Journal of Biological Sciences 11: 499-507. [ Links ]
17. Steel, R.G.D. J.H. Torrie y D.A. Dikey (1997). Principles and Procedures of Statistics: A Biometrical Approach. Third Edition. The McGraw-Hill Companies, Inc. New York, USA. 666 p. [ Links ]
18. Wessel-Beaver, L. y J.W. Scott (1992). Genetic variability of fruit set, fruit weight, and yield in a tomato population grown in two high-temperature environments. Journal of the American Society of Horticultural Science 117: 867-870. [ Links ]














