Introducción
La mandioca (Manihotesculenta) es una planta nativa de Sudaméricacuyo tubérculo posee un elevado valor nutricional, siendo una importante fuente de carbohidratos[1]. En la provincia de Misiones, el cultivo de mandioca aporta un 70% de la producción a nivel nacional, siendo su principal destino el consumo fresco[2]. Sin embargo, un 25% de esta producciónse utiliza para la obtención de fécula de mandiocadestinada a la industria alimenticia, textil y papelera[3]. La fécula es obtenida a través de un proceso industrial que genera residuos sólidos y líquidos.Los principales residuos sólidos son la cáscara, una pequeña proporción de harinay un material fibroso denominado bagazo compuesto por un 30-40% de almidón que puede ser utilizado en procesos microbiológicos para la obtención productos de valor[4]. Por otro lado, los residuos líquidos contienen agua, almidón, fibra, minerales y compuestos cianogénicos. La descarga directa de estos efluentes en una corriente de agua produce una caída muy importante en el oxígeno disuelto debido a la presencia de materia orgánica en exceso [5]. Por otra parte, uno de los componentes de estos residuos es un glucósido denominado linamarinacuya hidrólisis da como resultado glucosa, acetona y ácido cianhídrico que es altamente tóxico[6] por lo que la eliminación de los efluentes sin tratamiento pone en riesgo los ecosistemas acuáticos y la salud humana. Normalmente, las fábricas de fécula procesan sus efluentes utilizando lagunas de decantación como un paso que favorece la degradación aeróbica de los compuestos orgánicos. En la actualidad, la incorporación de un proceso adicional de tratamiento anaeróbico permite la producción de biogas a partir de esos efluentes y logra minimizar la toxicidad de ácido cianhídrico [7].
Una alternativa para el tratamiento de efluentes industriales es la biorremediación biológica. Este proceso se puede llevar a cabo mediante dos estrategias: la bioestimulación y la bioaumentación[8]. La primera, consiste en la adición de componentes que mejoren la actividad metabólica de los microorganismos presentes en el sitio contaminado, mientras que en la segunda seincorporan bacterias que sean capaces de eliminar ciertos compuestos tóxicos [8]. En ambos casos se presenta como desafío la prevalencia de los microorganismos cuya actividad fisiológica sea la indicada para el proceso de remediación. Por este motivo, es necesario conocer la diversidad de microorganismos presente en los efluentes para establecer las capacidades metabólicas in situ,ya sea para mejorar el proceso de bioestimulación o para seleccionar aquellos que serán reintroducidos a mayor escala en el ambiente contaminado[8].Varios estudios han demostrado que existen bacterias y hongos que pueden metabolizar el cianuro comoúnica fuente de nitrógeno y carbono para originar productos finales no tóxicos, tanto en ambientes aerobios como anaerobios, incluyendoBacilluspunillus, Pseudomonasfluorescens, Pseudomonaspaucimobii, Pseudomonaspseudoalcaligenes, Leptosphaeriamaculans, Fusarium oxysporum, Burkholderiacepacia, Trichodermasp., Klebsiellaoxytoca,Escherichiacoliy Azotobactervinelandii[9]. Por otro lado, además de compuestos cianogénicos, los residuos en los efluentes contienen un 55-60% de almidón que permanece inseparable de las fibras de celulosa[10]y que podría actuar estimulando la producción de amilasas bacterianas[11, 12].
Dada la importancia de los microorganismos en los procesos de reducción de la carga orgánica en los efluentes de la industria almidonera, se plantea como objetivo de este trabajo el aislamiento y caracterización de bacterias con potencial para la degradación de compuestos cianogénicos, asimilación de cianuro y producción de amilasas, que podrían ser utilizadas en procesos de biorremediación.
Materiales y Métodos
Muestras de efluentes
Las muestras de efluentes provenían de una planta industrial almidonera local. Las mismas fueron colectadas en botellas de plástico de 500mL, mantenidas en frío y procesadas dentro de las 24hs.
Aislamiento de microorganismos
Para el aislamiento de las bacterias se utilizó un medio mínimo con la siguiente composición: 1 g.L-1K2HPO4.2H2O, 0.2 g.L-1 MgSO4.7H2O, 0.01 g.L-1 CaCl2.H2O, 0.01 g.L-1NaCl, 0.2 g.L-1 MnSO4.4H2O, 0.2 g.L-1 CuSO4 y 0.2 g.L-1 ZnSO4.7H2O. Este medio fue suplementado con fécula de mandioca comercial (Medio Mínimo con Fécula de Mandioca, MMFM) o bagazo de mandioca provista por la almidonera (Medio Mínimo con Bagazo de Mandioca, MMBM) a una concentración de 2 g.L-1. Ambos medios fueron esterilizados en autoclave a 121°C por 15 min a 1 atm. de presión. Luego, a cada uno se adicionó 10 mL de muestra y se incubó con agitación 150 rpm a 30°C por 24 h. Los cultivos fueron realizados por triplicado.
Una alícuota de la muestra fue también cultivada en medio Luria-Bertani (LB). Para ello, se utilizó 70 mL de la muestra del efluente y 30 mL de LB líquido, luego de 24 h se tomaron 50 mL de ese cultivo y se adicionaron a 50 mL de LB. Finalmente se adicionaron 30 mL del cultivo anterior a 70 mL de LB. Los cultivos se incubaron por 24 h a 30°C a 150 rpm.
La selección de las bacterias fue realizada por agotamiento de estría en medio sólido. Para el aislamiento, se realizaron diluciones seriadas 1:10 de cada uno de los cultivos en agua destilada estéril y se inocularon 0.1 mL en placas de Agar nutritivo (Britania) conteniendo Carbendazim como antifúngico (0.5 g.L-1) para la obtención de colonias aisladas. Éstas fueron incubadas por 24 h a 28°C y 37°C. Los aislamientos se conservaron en LB con glicerol (10 %, v/v) a -20°C.
Selección de cepas amilolíticas
Las colonias de los microorganismos aislados previamente fueron inoculadas en placas de Petri divididas en cuadrantes conteniendo agar (12 g.L-1) con almidón soluble (10 g.L-1) a pH 7,0 y pH 5,1.Se inocularon por punción entre 22 a 24 aislamientos por placa y se incubaron a 28°C y 37°C durante 72 h. Se realizaron dos repeticiones del ensayo con duplicados de cada placa. Posteriormente, cada placa fue cubierta con solución de iodo 1% (p/v) durante 5 min y se seleccionaron los aislamientos cuyas colonias presentaron un halo de clarificación alrededor de las mismas indicando la hidrólisis del almidón.
Caracterización morfológica y fisiológica de cepas amilolíticas
La caracterización macroscópica de los microorganismos aislados y seleccionados se realizó mediante los parámetros: forma, tamaño, elevación, margen, color, superficie, densidad y consistencia de las colonias desarrolladas en placas de Petri conteniendo Agar nutritivo (Britania). Para la identificación microscópica se llevó a cabo la tinción diferencial de Gram de cada aislado.
Para la prueba bioquímica convencional de la catalasa [13], sobre un portaobjetos se suspendió una pequeña cantidad de cultivo de bacteria con ansa ojal y con ayuda de una pipeta Pasteur se colocó una gota de agua oxigenada (10v), seguidamente, y según la detección de formación de burbujas, se clasificaron como catalasas positivas o negativas.
Identificación molecular
La extracción de ADN se realizó según la técnica descripta por Hoffman y Winston [14]. Se utilizó la técnica de reacción en cadena de la polimerasa (PCR) para amplificar un fragmento de ~1500 nt (nuceótidos), la región del DNA que codifica para el rRNA 16S, utilizando los cebadores sentido y antisentido, 27F y 1492R[15]. Los fragmentos amplificados se observaron mediante electroforesis en gel de agarosa, utilizando 4 µL de producto amplificado y 2 µL de solución Gel Red (Promega). La corrida electroforética se realizó a 110V por 30 minutos en solución de tampónTBE 0.5% (10 mMTris-HCl, 10 mMácido bórico,1 mMEDTA). Los geles se observaron bajo luz UV.
Las secuencias obtenidas fueron alineadas utilizando ClustalW junto a secuencias del mismo género o similares disponibles en la base de datosNational Center forBiotechnologyInformation (NCBI, https://www.ncbi.nlm.nih.gov/). El análisis filogenético fue inferido por el método Neighbor-Joining y las distancias evolutivas se calcularon utilizando el método de máxima verosimilitud compuesta con un soporte estadístico (Bootstrap) de 2000 réplicas para la construcción de árboles filogenéticos mediante el software MEGA X [16].
Crecimiento de cepas amilolíticas
Para determinar la tasa de crecimiento de los aislamientos que mostraron actividad amilolítica, se procedió a cultivarlas en tres medios:(i) medio mínimo con fécula de mandioca 1% p/v, (ii) medio LB y (iii) medio LB con almidón soluble 1% p/v.
Se prepararon preinóculos con una ansada proveniente de una placa en 10mL del medio correspondiente que fueron incubados a 28°C, con 200rpm de agitación durante 24 h. Luego de ese período, se tomó una alícuota de 1mL, se diluyó 1:100 y se midió la absorbancia a 600nm. Se realizó el cálculo correspondiente para determinar el volumen de preinóculo necesario para iniciar un cultivo con una absorbancia de 0.2. Estos cultivos fueron incubados a 28°C con agitación (200rpm). Para realizar la curva de crecimiento se midió la absorbancia cada 1h a partir de 1mL de muestras. Luego de las 3h de iniciada la curva las muestras fueron diluídas 1:100.
Selección de cepas cianogénicas
Los aislados bacterianos seleccionados por su actividad amilolítica fueron inoculados por el método de punción en placas que contenían medio mínimo agarizado suplementado con glucosa al 1% y cianuro en distintas concentraciones (5 mM, 10 mM, 20 mM y 30 mM). Las placas fueron incubadas en estufa a 37°C por 24 h.
Resultados y discusión
A partir de las muestras de efluente se realizaron 93 aislamientos bacterianos por agotamiento de estría en medio sólido. Utilizando el medio MMBM se aislaron28 cepasa 28°C y 25 cepas a 37°C. Con el medio de MMFM se aislaron 9 y 12 cepas a 28°C y 37°C, respectivamente. Finalmente,en medio LB se aislaron 10 cepas a 28°C, mientras que a 37°C, se obtuvieron 11 aislamientos.Cada aislado fue nombrado mediante las siglas BAM (Bacterias Amilolíticas Misiones) y enumeradas (1-93). El número de aislamientos obtenidos en este trabajo puede relacionarse con la elevada carga de material orgánico presente en el efluente, lo que favorece el crecimiento bacteriano. En trabajos previos de análisis de comunidades bacterianas en efluentes de mandioca, se ha reportado que la carga bacteriana es alta, lo que indica una gran diversidad de microorganismos[17, 18].
En la determinación cualitativa de la actividad amilolítica (Tabla 1), se ha observado que cuando las bacterias crecieron a 28ºC y 37°C, tanto a pH 5 como a pH 7, el aislamiento BAM023 siempre se formó un halo de clarificación (Figura 1).
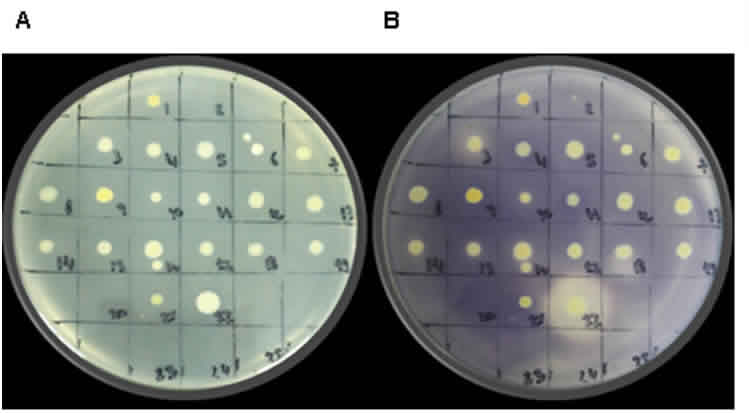
Tabla 1: Determinación cualitativa de actividad amilolítica. Condiciones de pH y temperatura en las que los aislados formaron halos de clarificación por producción de amilasas.

Figura 1: Determinación de actividad amilolítica. La actividad fue revelada mediante tinción con solución de iodo 1% a pH 7 y 37°C. A) Placa inoculada sin tinción. B) Placa con tinción que presenta un pequeño halo de clarificación alrededor de la colonia desarrollada en el cuadrante 3 (BAM005), cuadrante 18 (BAM020) y un halo de mayor tamaño en cuadrante 22 (BAM023).
Si bien la proporción de aislamientos positivos para la actividad amilolítica es relativamente baja (5% del total verificados) hay que tener en cuenta que las condiciones de cultivo,como la temperatura y el pH, así también como la presencia de azúcares simples y fuentes de nitrógeno, pueden afectar la producción y actividad de las enzimas amilolíticas, [19].
Las características morfológicas de las 15 cepas observadas in vivo así como los resultados para la prueba bioquímica de la catalasa se detallan en la Tabla 2.
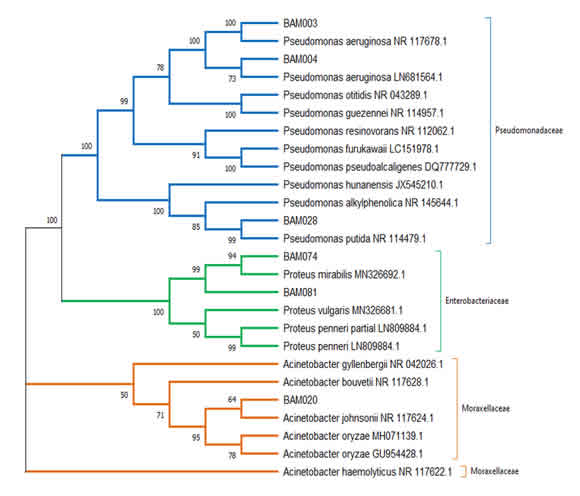
Tabla 2: Características morfológicas de los aislados bacterianos crecidos en placas de Petri con agar nutritivo, clasificación según tinción de GRAM y actividad catalas.
Junto a los 5 aislamientos que mostraron actividad amilolítica, fueron seleccionadas otros 10 aislados: BAM003, BAM004, BAM073, BAM074, BAM075, BAM076, BAM078, BAM081, BAM083, BAM089, que no dieron un resultado concluyente en la prueba cualitativa de degradación de almidón, pero fueron capaces de crecer cubriendo toda la superficie de la placa de Petri, indicando que podrían tener la capacidad de utilizar almidón de mandioca como nutriente.
La identificación molecular de las cepas seleccionadas se realizó mediante la secuenciación del 16SrDNA amplificado por PCR y por comparación de las secuencias obtenidas con la base de datos del NCBI.Selograron identificar a 6 de los 15 aislamientos preseleccionados como pertenecientes a los géneros Pseudomonas, Acinetobacter y Proteus.
Para dar un mayor soporte a la identificación taxonómica obtenida previamente se construyó un árbol filogenético (Figura 2) mediante un análisis de distancias utilizando el método Neighbor-Joining[20] disponible en el programa Mega X [16]. El análisis involucró 26 secuencias nucleotídicas de regiones ADNr 16S correspondiente a los aislados en estudio y algunas de las presentes en la base de datos.
En el árbol construido (Figura 2), las cepas BAM003, BAM004 y BAM028 han sido agrupadas con aquellas pertenecientes al género Pseudomonasde la familia Pseudomonadaceae con un soporte del 100%. La cepa BAM003 mostró una agrupación consistente y con soporte del 100% conPseudomonasaeruginosa (NR_117678.1), mientras que BAM028 se ha agrupado con soporte del 99% con secuencias pertenecientes aPseudomonasputida(NR_114479.1).Las secuencias de las cepas BAM074 y BAM081 se agruparon con aquellas pertenecientes al género Proteus, familia Enterobacteriaceae y con un grupo más pequeño de secuencias deProteusmirabiliscon un soporte mayor al 90%. Por último, BAM020 ha sido agrupada dentro de lasAcinobacterias de la familia Moraxellaceae.
Estos resultados coinciden con trabajos anteriores donde se ha reportado el aislamiento de bacterias comoProteus y Pseudomonasa partir de muestras tomadas en el punto de descarga de aguas residuales de mandioca [21-23]. Adicionalmente, se considera que Pseudomonasposee un gran potencial de biorremediación cuando forman consorcios con otras especies bacterianas [17]. Bacterias del géneroAcinetobacter que incluye especies patogénicas y no patogénicas[24], son abundantes en ambientes naturales[25], también han sido aisladas a partir de muestras de efluentes de fábricas de almidón de mandioca y evaluadas por su eficacia en la degradación de almidón y producción de biohidrógeno[26].

Figura 2: Relaciones filogenéticas entre las secuencias 16S rDNA. El árbol fue construido utilizando el método Neighbor-Joining. El árbol consenso se obtuvo a partir de 2000 réplicas y las distancias fueron computadas utilizando el método Máxima Verosimilitud Compuesta.
A continuación, se realizó una evaluación del crecimiento de las cepas identificadas como amilolíticas en el ensayo cualitativo. Utilizando medio mínimo suplementado con fécula de mandioca, las bacterias no superaron la fase de adaptación de crecimiento; en cambio, lascepas ensayadas mostraron un crecimiento eficiente con medio LB con1% de almidón soluble(Figura 3), la cepa BAM077 mostró mejor desempeño cuando creció en medio suplementado con almidón, mientras que la cepa BAM005, fue la única que mostró un crecimiento más eficiente cuando creció en medio LB sin la presencia de almidón.
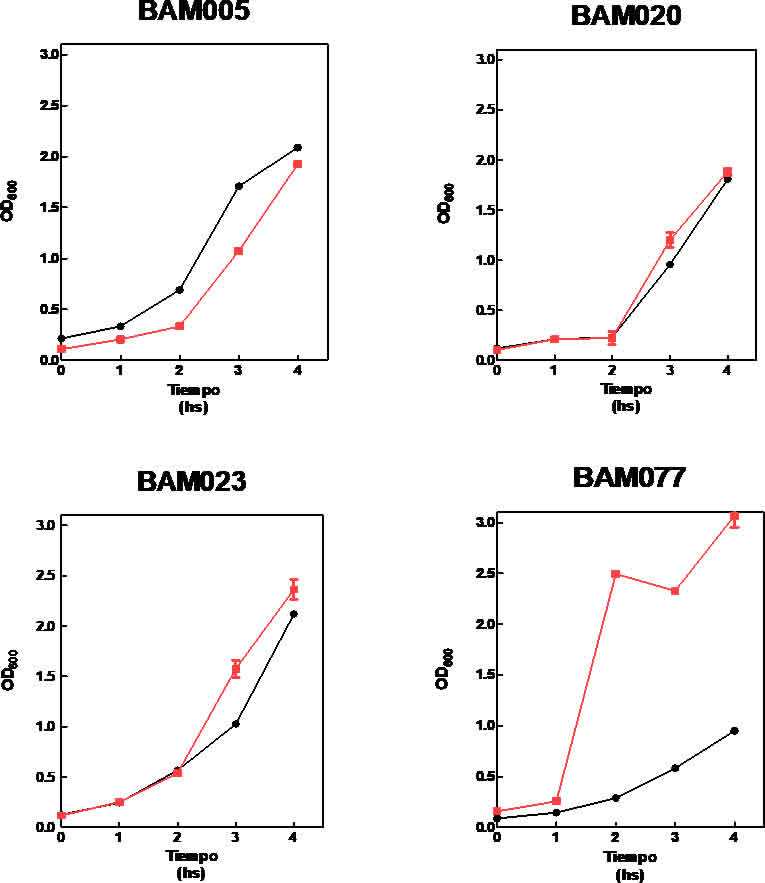
Figura 3: Curvas de crecimiento. Las bacterias seleccionadas crecieron en medio LB (línea negra) o medio LB suplementado con 1% de almidón (línea roja). A intervalos de 1 hr se midió la densidad óptica a 600nm.
Finalmente, las 15 cepas seleccionadas inicialmente fueron incubadas durante 24 h a 37°C, en las placas conconcentraciones crecientes de cianuro. Se visualizaron pequeñas colonias redondas de coloración celeste fluorescente con una tolerancia de hasta 20mM de cianuro en las inoculaciones realizadas en las posiciones 1, 2 y 7 (Figura 4), correspondientes a las cepas BAM003, BAM004 y BAM028. Ciertos microorganismos son capaces de utilizar cianuro y sus derivados como única fuente de carbono y nitrógeno, empleando una variedad de rutas para su descomposición [27, 28]. Sin embargo, en ocasiones, la toxicidad del cianuro restringe su uso como fuente primaria de carbono y, dado que la cantidad de nitrógeno necesaria para el crecimiento es menor que la de carbono, podría ser más fácil utilizarlo como fuente de nitrógeno en presencia de otra fuente de carbono como la glucosa. En nuestro ensayo, las cepas que crecieron en medio con cianuro pertenecen al género Pseudomonas, por lo que se presume que podrían ser capaces de utilizarlo como fuente de nitrógeno para su crecimiento.Se ha demostrado que cepas de Pseudomonasputidaaisladas a partir de aguas residuales de una fábrica de mandioca han presentado tolerancia y capacidad de crecer en concentraciones de cianuro por encima de 5mM, incrementando la tasa de eliminación en presencia de glucosa como fuente de carbono, con la formación de amoníaco y formiato como productos metabólicos finales sugiriendo una vía hidrolítica [29].
En otras investigaciones se ha demostrado la capacidad de Pseudomonasaeruginosa en la biodegradación de cianuro libre bajo condiciones alcalinas [30], por lo que su presencia y aislamiento a partir de muestras de efluentes industriales en el presente trabajo podría sugerir su potencial como biorremediador de estas aguas residuales.
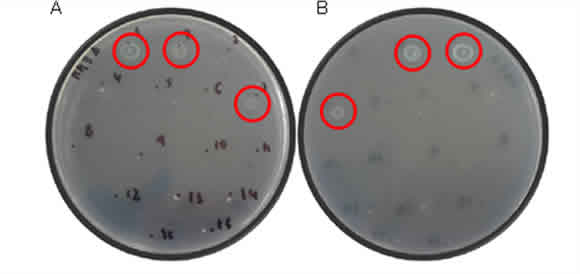
Figura 4: Tolerancia a cianuro. Crecimiento en medio mínimo con agar suplementado con glucosa 1% y cianuro 20 mM de colonias redondas con bordes de coloración celeste fluorescente en posiciones 1, 2 y 7 correspondientes a las cepas BAM003, BAM004 y BAM028, respectivamente. (A) Reverso de la placa. (B) Anverso de la placa.
Conclusiones
La cepa BAM028 identificada como perteneciente al géneroPseudomonas y cercana a la especie Pseudomonasputida, posee tanto la capacidad de degradar almidón como la de crecer en medio que contiene cianuro utilizándolo como fuente de nitrógeno. Dos de las cepas que crecen en forma de césped en las placas suplementadas con almidón, BAM003 y BAM004, cercanas a la especie Pseudomonasaeruginosa, no evidenciaron cualitativamente la capacidad de degradar almidón, pero sí pudieron crecer en presencia de cianuro.
Este estudio contribuye al conocimiento de la variedad de microorganismos que pueden ser aislados de efluentes de almidoneras industriales de la Provincia de Misiones y su potencial para ser utilizadas en la degradación de almidón y compuestos de cianuro como parte de una estrategia de biorremediación.
Conflicto de Interés
Los autores declaran que no existe conflicto de interés














