Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
BAG. Journal of basic and applied genetics
versión On-line ISSN 1852-6233
BAG, J. basic appl. genet. vol.21 no.1 Ciudad Autónoma de Buenos Aires ene./jun. 2010
ARTÍCULOS ORIGINALES
Mezcla génica y linajes uniparentales en Esquel (Pcia. de Chubut). Su comparación con otras muestras poblacionales argentinas.
Avena Sergio A1,2,5, Parolin M Laura1,2,5, Boquet Mariel3, Dejean Cristina B1,2, Postillone M Bárbara2, Alvarez Trentini Yisela1, Di Fabio Rocca Francisco1,2, Mansilla Florencia2, Jones Laura4, Dugoujon Jean Michel6, Carnese Francisco Raúl1,2.
1Sección Antropología Biológica. ICA. Facultad de Filosofía y Letras. Universidad de Buenos Aires (UBA). Puán 480, Ciudad Autónoma de Buenos Aires (CP1406). E-mail: antibiol@fi lo.uba.ar
2Departamento de Ciencias Naturales y Antropología. CEBBAD. Universidad Maimónides. Hidalgo 775 6º piso, Ciudad Autónoma de Buenos Aires (CP1405). E-mail: cebbad@maimonides.edu
3Facultad de Ciencias Naturales. Universidad Nacional de la Patagonia. SJB. Sede Esquel
4Servicio de Hemoterapia. Hospital Zonal de Esquel.
5Consejo Nacional de Investigaciones Científicas y Técnicas (CONICET), Argentina.
6Centre d'Anthropologie. UMR 8555. Toulouse, Francia.
Correspondencia a: Dr. Francisco R. Carnese. Sección Antropología Biológica. Facultad de Filosofía y Letras. Universidad de Buenos Aires. Puán 480. Piso 4, Of. 407. Ciudad Autónoma de Buenos Aires. Argentina. Tel: 54-11-4432-0606, Int. 145. E-mail: antbiol@gmail.com
RESUMEN
En este trabajo se estimó la composición genética de una muestra poblacional de Esquel, situada en la Patagonia Andina Argentina. Se estudiaron 59 donantes de sangre no emparentados que concurrieron al Hospital Zonal. Fueron analizados 5 sistemas eritrocitarios, alotipos GM, haplogrupos mitocondriales europeos, amerindios y africanos, y la mutación M3 del cromosoma Y. Se realizó una encuesta para obtener información genealógica de los donantes. La mezcla génica se estimó mediante el Programa ADMIX. Los marcadores proteicos arrojaron 46.9% de componente indígena y 1.9% de africano. Se observó un 79.6% de linajes mitocondriales amerindios, no registrándose aporte subsahariano. Un 23% de los varones analizados poseen la variante aborigen M3. Esa diferencia en la contribución genética sexo-específi ca revelaría un aporte asimétrico por género en la historia de esta población. Se compararon estos datos con los obtenidos con anterioridad en el Área Metropolitana de Buenos Aires, Bahía Blanca y Comodoro Rivadavia y también con migrantes a Buenos Aires provenientes del norte y oeste argentino. La mezcla génica mostró los mayores valores de aporte amerindio en el Noroeste Argentino seguido por Esquel, donde por otra parte se constató el más bajo aporte subsahariano. Con respecto a los marcadores uniparentales, en Esquel y en el norte del país se observaron altos porcentajes de linajes maternos autóctonos, pero la localidad patagónica se diferenció por presentar el más elevado aporte paterno nativo de todas las muestras analizadas, probablemente por la tardía incorporación de la Patagonia al Estado Nacional, conservando las comunidades autóctonas durante mayor tiempo su autonomía.
Palabras claves:Análisis genético; Sistemas proteicos; ADN mitocondrial; Cromosoma Y; Región Andina Patagónica.
ABSTRACT
In the present study the genetic compositon of Esquel, city located in the Argentinean Andean Patagonia, was estimated in a population sample. A total of 59 non-related blood donors from the Zonal Hospital were included. Five eritrocitary systems, GM allotypes, Amerindian, European and African mitochondrial haplogroups, and the M3 mutation in Y chromosome were analysed. A survey was done to obtain genealogical information about the donors. Genetic admixture was estimated employing the ADMIX program. Protein markers showed the presence of 46.9% for the indigenous component and 1.9% for the african component. Amerindians mitochondrial haplogroups represented a 79.6%, no Subsaharian lineage was present. 23% of males analysed exhibited the aboriginal variant M3. This difference in the sex-specifi c genetic contribution would reveal a gender asimetry in the population history. The data were compared to those obtained previously in the Buenos Aires Metropolitan Area, Bahía Blanca and Comodoro Rivadavia and also with migrants to Buenos Aires coming from the north and the west from the country. Genetic admixture showed higher values for the Amerindian contribution in the Argentinian Northwest followed by Esquel, where the subsaharian component was the lowest found. When considering uniparental markers the higher percentages of autoctonal lineages were observed in Esquel and the north of the country, although the patagonic village presented the highest paternal native component considering all the samples analysed, probably due to the late incorporation of the Patagonian Region to the National State, keeping the autoctonal communities for a longer their authonomy.
Key words: Genetic analysis; Protein markers; Mitochondrial DNA; Y chromosome; Andino-Patagonian Region.
INTRODUCCIÓN
La ciudad de Esquel (ESQ) es cabecera del Departamento de Futaleufú en la Provincia de Chubut, se encuentra 1900 Km al sudoeste de Buenos Aires. Se ubica en la Patagonia Andina, en la transición entre el bosque andino y la estepa. Luego de las campañas militares contra la población autóctona, que concluyen con la ocupación de sus tierras y la incorporación de la Patagonia al territorio argentino, se realizó en 1885 una expedición hacia el oeste chubutense integrada en su mayoría por galeses y dirigida por el Gobernador Fontana. La región parecía muy apropiada para la agroganadería, por la fertilidad del suelo y la existencia de agua, creándose en 1888 la Colonia 16 de octubre. En 1904 arribó al lugar el Ing. Molinari con instrucciones de establecer un pueblo, el cual se fundó en el límite norte de dicha colonia. Finkelstein y Novella (2006) señalan que en la región se dieron dos tipos de poblamiento: a) el planificado, que a su vez tiene tres variantes, la colonia galesa 16 de Octubre, las"Colonias Pastoriles Aborígenes" como Cushamen, Epulef y Nahuelpan (ubicadas generalmente en áreas marginales y rodeadas por latifundios) y las"empresas de colonización", las cuales muchas veces fracasaron y pasaron a engrosar las propiedades de las grandes compañías, como la inglesa Argentine Southern Land, hoy en manos de la firma Benetton; b) el no planificado, mayoritariamente desde Chile, ingresando al actual territorio argentino por Neuquén y Río Negro y desde allí hacia el sur por el límite con la meseta para reingresar a los valles andino-patagónicos no ocupados. De esta forma se inició el poblamiento de El Bolsón, Cholila, Epuyén, El Hoyo, Corcovado y Río Pico (Gavirati et al., 2006). A este tipo de asentamientos también contribuían grupos autóctonos, que ante el despojo sufrido y al no obtener tampoco tierras en las reservas indígenas, ocuparon terrenos fiscales, pero en este caso se trataba generalmente de desplazados desde otras regiones como Chile, Neuquén, Río Negro y La Pampa (Novella, 2005).
Un aspecto interesante es que en el Censo Nacional de 1895 no existen datos oficiales sobre la cantidad de población autóctona, pues la categoría de"indio" no figuraba en las planillas censales. En Chubut, el Gobernador Eugenio Tello afirmaba que los indígenas habían sido censados como"argentinos" y residían en su totalidad en el oeste, donde constituían el 63% de la población (Novella, 2005). Esto nos marca una diferencia importante con Comodoro Rivadavia (CR), situada en la costa, donde la población originaria para esa época era muy escasa. Recordemos que esta ciudad, que estudiamos con anterioridad, está situada en una árida estepa con reducida sustentabilidad ecológica, en un área donde actualmente tiene una gran incidencia la industria petrolera. ESQ, por su parte, se ubica en una región más húmeda que permite la actividad agroganadera, forestal y turística. Esto repercute en el carácter de su inmigración, la cual presenta un flujo más constante y no tan influido por los vaivenes de una única actividad, siendo además de características más locales.
Hacia 1915 Esquel contaba con 1500 habitantes. En ese momento la actividad principal era la ganadería ovina y bovina, con destino a Chile, pero desde 1911 las restricciones aduaneras desalentaron la circulación de pobladores y mercancías entre ambos lados de la cordillera. Esto ocasionó que se intensificaran el cultivo de cereales y la creación de varios molinos en la zona, que empiezan a declinar tres décadas después por la competencia con la producción de la pampa húmeda, facilitada por la llegada del ferrocarril en 1945. El desarrollo de la red ferroviaria y vial favoreció la transformación de la ciudad en un centro comercial y turístico (Troiano, 1993). Además existe una creciente actividad agropecuaria de fruta fina. Cabe destacar también la presencia de numerosas instituciones vinculadas al desarrollo y la extensión de tecnología, en estrecha vinculación con el uso de los recursos naturales. Pueden mencionarse la sede local de la Universidad Nacional de la Patagonia"San Juan Bosco", la Estación Experimental Agroforestal del INTA y el Centro de Investigación y Extensión Forestal Andino Patagónico (Oriola, 2003).
El poblamiento de ESQ fue paulatino dado el carácter diversificado de su estructura productiva (Tabla I). Sin embargo, se observa un aumento en los años 60, claramente por encima del total provincial, cuando se incrementó significativamente la presencia del Estado y su rol como empleador, sobre todo en las oficinas públicas.
Tabla I. Porcentaje de crecimiento intercensal en Esquel y Provincia de Chubut.

Estos comentarios acerca de los principales acontecimientos histórico-demográficos ocurridos en ESQ, y que han tenido seguramente repercusión sobre la estructura genética de la población, resultan pertinentes y necesarios para contextualizar la información biológica obtenida en este estudio.
Dentro de ese marco, el propósito de este trabajo es analizar la composición genética de una muestra poblacional de la ciudad andino-patagónica de Esquel (ESQ), y comparar los datos obtenidos con otras 6 muestras del país, 3 de ellas fueron estudiadas con anterioridad por nuestro equipo de investigación: el Área Metropolitana de Buenos Aires (AMBA), Bahía Blanca (BB) y Comodoro Rivadavia (CR), mientras que las tres restantes provienen de inmigrantes al AMBA desde el Noroeste (NOA), Nordeste (NEA) y Cuyo.
MATERIALES y MÉTODOS
Se analizó una muestra de 59 dadores de sangre no emparentados que concurrieron al Hospital Zonal de Esquel durante el año 2008, a quienes se les informó sobre los alcances del presente trabajo y prestaron su consentimiento para la realización del mismo. A los donantes se les realizó una encuesta con la finalidad de obtener información sobre su origen y el de sus ancestros, para lo cual se recabaron datos sobre lugar de nacimiento, residencia actual e información genealógica de las dos generaciones precedentes. Las muestras se tipificaron para los sistemas ABO, Rh (C, c, D, E, e) y Gm. Para las determinaciones eritrocitarias se empleó la técnica de microtipificación en tarjetas DIAMED (Avena et al., 2006). El sistema Gm se determinó utilizando el método basado en la inhibición de la aglutinación (Field y Dugoujon, 1989). Para las determinaciones moleculares se realizó la extracción de ADN con el método del fenolcloroformo, (Sambrook et al., 1989). En el sistema Duffy se amplificó un segmento de ADN de 221 pares de bases del exón 1 (cromosoma 1 q21-q23) del gen DARC (Duffy antigen chemokine receptor) que fue digerido con Sty I. El alelo Fy*null tiene un sitio de corte en la posición 94. Los fragmentos fueron visualizados en un gel de poliacrilamida al 12% (Tournamille et al., 1995; Pogo and Chaudhury, 2000). En el sistema Diego se amplificó un segmento de 149 pares de base del exón 19 (cromosoma 17 q12-q21). El alelo Di*A se reconoce por no presentar sitio de corte para la enzima MspI en la posición 70 (Baleotti et al., 2003), realizándose la lectura en un gel de agarosa al 2%.
Para la identificación de los linajes mitocondriales se procedió de la siguente manera: a) Amerindios: se empleó la técnica descripta por Stone y Stoneking (1993). Los haplogrupos A, C y D fueron digeridos con sus enzimas de restricción específicas (Hae III, Hinc II y Alu I, respectivamente). La deleción de 9 pares de bases que caracteriza a B fue observado en un gel de poliacrilamida al 12%. b) Subsaharianos: Se realizó la determinación del sitio de corte para HpaI en la pn 3592, presente en los subhaplogrupos L1 y L2. Estos tienen una prevalencia de entre el 75 al 95% en África Subsahariana (Chen et al. 2000) y se encuentra ausente en las poblaciones autóctonas de los otros continentes. c) Europeos: Los haplogrupos X y U fueron caracterizados siguiendo el protocolo de Torroni et al. (1996), mientras que para la tipificación de J se siguió el de Martínez Marignac et al. (1999). Para H, K, T, I, V y W se modificó la técnica de Torroni et al. (1996), alterando la longitud de los fragmentos obtenidos para una mejor lectura. A excepción del caso ya señalado de B, todo el resto de los haplogrupos fueron visualizados en geles de agarosa al 2%. Dos muestras que no habían podido ser determinadas fueron secuenciadas entre las posiciones 15989 y 16410 de la región HVI en el laboratorio del Instituto de Biotecnología CICVyA - CNIA - INTA. La alineación de las secuencias se realizó utilizando el programa Sequencher 4.10.1. La corroboración para eliminar indeterminaciones se realizó utilizando el programa Sequence Scanner v 1.0 (http://www. appliedbiosystems.com/absite/us/en/home. html). En ambos casos se trataba de haplogrupos europeos, J en un caso y U en el otro.
El marcador M3 del cromosoma Y fue analizado siguiendo el protocolo de Underhill et al. (1996). Se realizó una PCR alelo específica, desarrollando dos reacciones de amplifi cación, difiriendo el cebador reverso alelo específico entre sí sólo por una base, en la posición 181: G para la variante C y A para la T, esta última define el linaje americano Q1a3a. Esta transición alcanza una frecuencia del 75 al 90% en aborígenes sudamericanos y se encuentra ausente en europeos y africanos (Bianchi et al., 1997). Aunque ampliamente mayoritario entre los linajes autóctonos, este polimorfismo puede estar ausente en el caso de aquellos nativos que pertenezcan al parahaplogrupo Q*, el cual tiene en el Cono Sur una incidencia menor al 6% a excepción de los Ayoreo y Lengua del Gran Chaco (Bailliet et al. 2009)
El cálculo de las frecuencias génicas se estimó empleando los métodos de máxima verosimilitud de Reed y Schull (1968) para los grupos sanguíneos (programa de computación MAXLIK) y de Edwards (1984) para el sistema Gm. Para los marcadores uniparentales se empleó el conteo directo. La proporción de mezcla génica se estimó a partir de los marcadores proteicos mediante el método de identidad génica de Chakraborty (1985) implementado en el programa ADMIX.95 (disponible en http:// www.genetica.fmed.edu.uy/software.htm). La construcción de la parental europea fue realizada promediando las frecuencias alélicas de españoles e italianos, que constituían más del 90% de los inmigrantes transoceánicos. La parental africana se obtuvo a partir de los datos disponibles de las regiones desde donde se produjo el tráfico de esclavos hacia Sudamérica que corresponden principalmente a los territorios actuales de Senegal, Nigeria, Angola y Mozambique. Para sudamerindios se utilizaron los datos existentes sobre comunidades del Cono Sur (la explicación detallada de la conformación de las parentales ha sido expuesta en Avena et al., 2006).
El análisis molecular de la varianza (AMOVA) y las diferencias genéticas entre pares poblacionales (pairwise Fst) fueron determinadas mediante la ejecución del programa ARLEQUIN 3.1 (Excoffier et al., 2005). La construcción de los correspondientes dendrogramas de afinidades biológicas se llevó a cabo por el algoritmo UPGMA (Unweighted Pair Group Method with Arithmetic Averaging) (Sneath y Sokal, 1973) utilizando el paquete estadístico Phylip v.3.65 (Felsenstein, 2004) y el programa Treeview 32 (Page, 1996).
Para la comparación de la información genética y con el objetivo de abordar también otras importantes regiones del país, hemos incluido datos correspondientes a migrantes al AMBA provenientes del Noroeste Argentino (NOA, N=100, Provincias de Jujuy, Salta, Santiago del Estero, Tucumán, Catamarca y La Rioja), Nordeste del país (NEA, N=98, Provincias de Formosa, Chaco, Corrientes y Misiones) y Cuyo (N=32, Provincias de Mendoza, San Juan y San Luis).
RESULTADOS
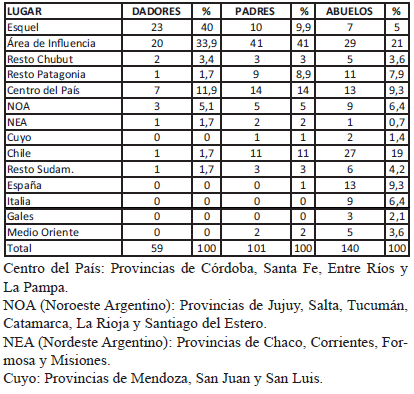
En la Tabla II se presenta la información genealógica obtenida a partir de las encuestas realizadas a los donantes de sangre, la cual guarda una estrecha relación con los fenómenos histórico- demográficos ya descriptos. Estos datos permitieron constatar que el 40% de los dadores, 10% de los padres y solamente 5% de los abuelos han nacido en ESQ, lo que se condice con lo reciente de su poblamiento. Del 32 % de los abuelos inmigrantes oriundos de la Patagonia, las dos terceras partes son originarios del noroeste del Chubut. Entre los argentinos, se destacan los nacidos en el centro del país (9%) y el NOA (6%), mientras que son escasos los provenientes del NEA y Cuyo. En lo que respecta a extranjeros, el 23% provenían de países limítrofes, fundamentalmente de Chile, seguidos por los que nacieron en Europa (18%), en su mayoría en España e Italia, pero también de Gales (2%). Considerando la generación de los dadores, sólo un 40% de nuestra muestra nacieron en Esquel. Pero si le sumamos el 34% nacido en la zona cercana vemos que casi 3/4 partes de nuestra muestra es de la ciudad o su área de influencia. Si observamos las tres generaciones vemos que todas las categorías de inmigrantes, a excepción de los que provienen de las zonas cercanas, disminuyen hacia el presente. Un detalle muy significativo se observó al comparar las abuelas maternas y los abuelos paternos, pues 16 linajes maternos eran locales, contra 6 paternos. Se realizó un test de X2, ubicándose estas diferencias sobre el límite de significancia (p=0,053).
TABLA II. Lugar de nacimiento de los dadores, sus padres y sus abuelos.
En la Tabla III se presentan los datos de las frecuencias génicas obtenidas para los marcadores proteicos y de las parentales utilizadas en este estudio. La participación autóctona se evidencia principalmente a partir de los valores superiores detectados, respecto a la parental europea, para ABO*0 (86%) y Gm*1 y 2 (53%). A su vez el 2.7% de Di*A es un claro indicador del aporte aborigen dado que su prevalencia es nula en las otras poblaciones parentales. Si bien se registró un 6,6% de Rh*cDe, los bajos valores de Fy*null (0,8%) y Gm*5 (0,8%) señalarían una escasa presencia africana. A partir de los datos obtenidos se estimó la mezcla génica, registrándose un 51.2% (s.e. 4,5), 46,9% (s.e. 4,6) y 1.9% (s.e 1,3) de contribuciones europea, indígena y subsahariana, respectivamente
Tabla III. Frecuencias alélicas de los sistemas proteicos.

El estudio del ADN mitocondrial mostró que el 79,6% de los haplogrupos eran de origen amerindio, distribuidos en un 1.7% de A, 22% de B, 23,7% de C y 32.2% de D. Asimismo, no se observó la presencia de linajes africanos (L1 y L2). El 20,4% restante fue asignado a linajes europeos, detectándose un 8,5% de H, un 5,1% de U, un 3,4% de T y un 3,4% de J.
En lo que respecta al marcador M3 del cromosoma Y, se estudiaron 30 varones, de los cuales 7 presentaron esta variante específica amerindia, 5 de ellos tienen un origen local, un caso proviene de Chile y el restante lo hace desde Santiago del Estero. Estas 7 personas también fueron asignadas al haplogrupo amerindio Q1a3a mediante la determinación de STRs (Parolin et al., 2010). La mezcla génica obtenida a partir de marcadores autosomales reveló un aporte autóctono intermedio (47%) respecto a los marcadores uniparentales, los cuales por su parte revelaron valores disímiles, un 23% de linajes amerindios en la línea paterna y un 80% en la materna. Este hecho revela que ha habido un aporte diferencial por género en la historia de esa población, en el sentido de una importante contribución de las mujeres nativas en los procesos de mestizaje.
DISCUSION
a) Del análisis genealógico
Al comparar los datos obtenidos en la muestra poblacional de Esquel con los resultados previos del AMBA, BB y CR, cuyas muestras fueron recolectadas siguiendo el mismo protocolo de investigación que en este trabajo, constatamos que el porcentaje de los abuelos de los dadores de la muestra de ESQ (26%) nacidos en la ciudad y su zona de influencia, supera a los registrados en el AMBA (20%), BB (7%) y especialmente CR (2%). El AMBA y BB mostraron similares porcentajes de europeos entre sí (35% y 36% respectivamente), superiores a los registrados en ESQ (24%) y CR (22%). ESQ presentó un marcado descenso de inmigrantes transoceánicos desde la categoría de abuelos hasta la generación actual, lo cual había sido observado, también, en las muestras de AMBA, BB y CR (Avena et al., 2006; 2007 y 2009) y se encuentra en concordancia con lo registrado por los censos nacionales de población desde 1947 al presente.
Al analizar en nuestras muestras el origen de los inmigrantes de países limítrofes, se observó que provenían de Chile en un muy alto porcentaje: 83% en ESQ, 87% en CR (Avena et al., 2009) y 90% en BB (Avena et al, 2007), esta última situada en la transición entre la región patagónica y la pampeana. Nuestros datos fueron totalmente coincidentes con lo encontrado en la información censal, pues esos valores son del 83%, 86% y 91% en ESQ, CR y BB, respectivamente (Dirección Provincial de Estadística del Chubut, 2010 y Censo Nacional de Población y Vivienda, 2001). En la muestra del AMBA, en cambio, habíamos observado más diversidad en el origen de los abuelos de los dadores nacidos en otros países sudamericanos, y el contingente más numeroso estaba constituido por paraguayos (54%) y uruguayos (20%) (Avena et al., 2006).
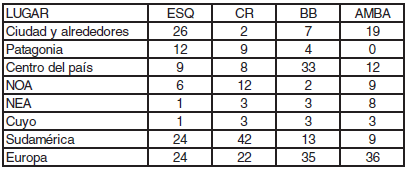
Si consideramos a los migrantes internos, también existe otra importante diferencia. Mientras que en el AMBA y BB el aporte principal se origina en el centro del país, y en CR destaca la inmigración producida desde el Noroeste, en Esquel se observó un aporte más local, desde la propia Patagonia (Tabla IV). En este caso también la información genealógica de nuestra muestra se corresponde con los datos de la Dirección Provincial de Estadística del Chubut (2010).
Tabla IV. Origen de los abuelos de los dadores de las muestras de ESQ, CR, BB y AMBA.
b) Del análisis genético
A partir de los datos obtenidos para los marcadores autosómicos, se observó que el valor de aporte aborigen en ESQ (47%) fue superior a los registrados por nosotros en otras poblaciones de la Argentina, especialmente las de la región pampeana (AMBA y BB), con la única excepción de la muestra del NOA (58%). La contribución africana tuvo un rango de variación de entre el 2% en ESQ al 5% en NOA (Figura 1). Recordemos que en esta última región se introdujeron una gran cantidad de esclavos para reemplazar a la mano de obra nativa que fue diezmada por la explotación española. En 1778 su porcentaje, incluyendo a las variantes mestizadas de zambos y mulatos, superaba el 50% en el Tucumán colonial (Lorandi, 1992). Con respecto a la Patagonia, según fuentes históricas la presencia de africanos era importante en las guarniciones de frontera. Allí se producían deserciones hacia las comunidades autóctonas dada las difíciles condiciones de vida en los fortines (Andrews, 1989). Este fenómeno tiene un correlato en poblaciones aborígenes actuales de esta región, donde se ha detectado mezcla génica con europeos y subsaharianos (Carnese et al., 1993, 2003; Goicoechea et al., 2001).

Figura 1. Contribución genética de los componentes europeo, amerindio y africano observada en ESQ y en seis poblaciones estudiadas previamente.
A su vez, ESQ presentó un mayor componente nativo y un menor subsahariano respecto a lo registrado en la ciudad bonaerense de La Plata, 26% y 6%, respectivamente (López Camelo et al., 1996 y Martínez Marignac et al., 2004). Asimismo, comparte los elevados valores de contribución autóctona registrados en el noroeste argentino (40%) por Alfaro et al. (2005) en muestras de Jujuy, Salta, Tucumán, Santiago del Estero, Catamarca y La Rioja, aunque en ellas se detectó un mayor aporte africano (10%). Se aprecia que, a un nivel general, los valores de componente europeo son más altos en el centro de la Argentina, y decrecen hacia el sur y el norte. Seldin et al. (2007), a partir del estudio de 78 SNPs, calcularon en 19% el componente aborigen y en 2% el subsahariano para todo el país. Sin embargo, el reducido tamaño de la muestra (94 individuos) no resulta adecuado para dar cuenta de las diversidades regionales, especialmente porque las cinco ciudades muestreadas en ese trabajo pertenecen a la pampa húmeda. En un reciente trabajo, Corach et al. (2010) analizaron 24 SNPs en 246 personas provenientes de 8 provincias argentinas, del sur, centro y nordeste del país, encontrando también un 19% de aporte autóctono y aproximadamente un 4% de componente subsahariano. A su vez, nuestro grupo de investigación también realizó la determinación de 106 SNPs en las mismas muestras presentadas en este trabajo, obteniendo prácticamente los mismos resultados de mezcla génica poblacional (Avena, Fejerman et al., 2010).
En lo que respecta al ADN mitocondrial, en ESQ se registró un elevado componente amerindio (80%), similar al NOA (80%) y NEA (79%). A nivel regional, se aprecia que los porcentajes de linajes maternos nativos son mayores en el norte y sur del país, respecto a la región pampeana y Cuyo. Por el contrario, la línea paterna autóctona sólo alcanzó un 23% en ESQ, y ese valor fue inferior incluso en todas las otras muestras, con un rango entre el 8%, en NOA y Cuyo, al 3% en el AMBA, (Figura 2).
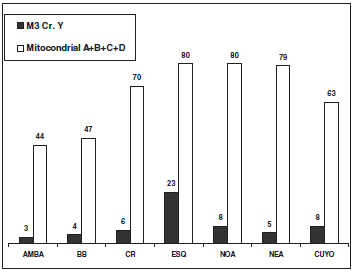
Figura 2. Porcentajes de linajes amerindios paternos (M3) y maternos (mitocondrial) en las muestras analizadas.
La mayor presencia del linaje indígena por vía materna, respecto a la paterna, resulta concordante con un modelo donde se dé principalmente el cruzamiento de mujer nativa con varón de otro origen, lo cual ha sido ampliamente observado en nuestro país, como lo evidencian los trabajos de Dipierri et al. (1998) en dos localidades a distinta altitud de la Quebrada de Humahuaca, Martínez Marignac et al. (1999, 2004) en La Plata, de Carnese (2006) en el AMBA, de García y Demarchi (2006) sobre dos poblaciones criollas cordobesas, de Ramallo et al. (2007) en un aislado rural de Catamarca, de Marino et al. (2007) y Corach et al. (2010) en muestras de diez y ocho provincias argentinas, respectivamente. Este proceso también fue detectado en otros países de Sudamérica, como Brasil (Bortolini et al., 1999; Santos et al., 1999, Marrero et al., 2007), Uruguay (Sans et al., 2002), Chile (Rocco et al., 2002) y Colombia (Bedoya et al., 2006). Recientemente, Wang et al. (2008) han analizado los patrones geográficos de miscegenación en"mestizos" de Argentina, Brasil, Chile, Colombia, Costa Rica, Guatemala y México, corroborando que el cruzamiento diferencial por género fue la regla en Iberoamérica. Este proceso comenzó seguramente en el momento mismo de la conquista, con el arribo de hombres europeos en su mayoría sin familia, continuándose posteriormente en la etapa colonial y, en menor medida, durante fin del siglo XIX y la primera parte del XX, con las oleadas inmigratorias desde el viejo continente con un alto índice de masculinidad (Lorandi, 1992; Avena et al., 2006). En ese sentido, la mayor prevalencia de M3 en ESQ podría explicarse porque la Patagonia fue uno de los últimos territorios en ser incorporado al Estado Nacional, por lo tanto las comunidades autóctonas pudieron conservar durante mayor tiempo su autonomía y, seguramente, mantener uniones biológicas preferentemente endogámicas. Se realizó un test exacto de Fisher, observándose diferencias significativas entre ESQ y el resto de las muestras, con la única excepción de Cuyo (p=0,135).
Mediante el análisis de pares de poblaciones fueron observadas diferencias significativas (Fst P-values < 0.05) con el NOA para el sistema Gm, con BB y NEA respecto a los haplogrupos mitocondriales y con el AMBA para los sistemas ABO, Gm y ADNmt. Respecto a las parentales Amerindia, Europea y Africana, ESQ mostró diferencias significativas para todos los sistemas estudiados, a excepción de Diego para las 3 parentales y de Duffy con europeos. A partir del análisis molecular de la varianza (AMOVA), se observó en general un bajo índice de diferenciación genética (FST) entre las poblaciones, con un aumento de la divergencia para los sistemas Gm y ADNmt. A este respecto puede observarse que la diversidad genética intrapoblacional explica la mayor parte de la variabilidad observada (Tabla V).
Tabla V. Matriz de signifi cancia Fst P-values obtenida a partir de las comparaciones entre pares poblacionales.

A partir de las matrices de distancias genéticas, se procedió a construir los dendrogramas de afi nidades biológicas utilizando el método UPGMA. En el correspondiente a los marcadores proteicos, se observó por un lado un subconjunto de similitud entre AMBA y BB, que se agrupó luego con los europeos y por el otro, un subconjunto integrado por las poblaciones andinas (NOA y ESQ), que luego se agrupó con CR, NEA y Cuyo (Figura 3). Recordemos que en estas cinco poblaciones se habían registrado mayores valores de aporte amerindio. En el dendrograma correspondiente al ADNmt por su parte, la parental amerindia se agrupó con las muestras del norte, oeste y sur argentino. Esto guarda relación con el hecho de que en esas muestras se observaron mayores valores de haplogrupos autóctonos respecto a la región pampeana (AMBA y BB), (Figura 4). Es interesante remarcar que la similitud genética detectada en ambos dendrogramas entre ESQ y NOA, podría explicarse porque estas dos poblaciones presentaron los mayores valores de componente autóctono, tanto a nivel autosómico como del ADN mitocondrial.

Figura 3. Dendrograma UPGMA empleando los marcadores proteicos.

Figura 4. Dendrograma UPGMA empleando los haplogrupos mitocondriales.
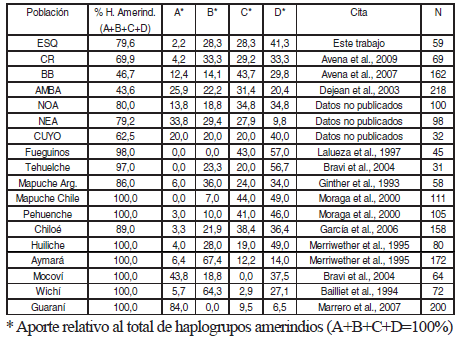
Además de esta diferencia, al analizar la representación de los cuatro haplogrupos autóctonos se observó otra importante particularidad. En ESQ se registró el menor valor de A, lo que se corresponde con lo encontrado en poblaciones autóctonas del suroeste argentino y Chile (Mapuches, Tehuelches, Huiliches, Pehuenches) que han contribuido probablemente a la conformación de esta población. Por otra parte, las muestras donde se registró el mayor valor de A fueron el NEA y el AMBA. Recordemos que hacia esta ciudad se sucedieron migraciones de personas provenientes, principalmente, del nordeste argentino y Paraguay (Avena et al, 2006), cuyos pueblos originarios presentan una alta frecuencia de este haplogrupo, como por ejemplo entre guaraníes y mocovíes (Tabla VI).
Tabla VI. Distribución geográfi ca de los haplogrupos amerindios
Por último, es importante destacar que de los 20 donantes que no recordaban el lugar de nacimiento de sus abuelas, 19 poseían un haplogrupo amerindio, lo que nos permite suponer que las mismas habrían tenido un origen local. Por otra parte, se observó un gran aporte de linajes maternos amerindios locales, pues de las 16 abuelas nacidas en Chubut, 13 presentaban haplogrupos mitocondriales autóctonos. Esto ilustra la importancia de contar con la información genealógica de los dadores, porque nos ha permitido abordar las diferencias regionales existentes y rastrear el origen de las líneas uniparentales.
En conclusión, la muestra de ESQ presentó en los niveles proteico y mitocondrial mayor componente autóctono respecto al AMBA, BB, CR, NEA y CUYO. Además otra interesante particularidad es que en la ciudad surandina se registró el menor valor de aporte subsahariano y el mayor aporte paterno amerindio respecto a todas las muestras. Por último, en la muestra del NOA se registraron los mayores valores de aporte autóctono y africano. Estas diferencias nos advierten de la existencia de especifidades que son especialmente importantes en países de gran extensión y diversidad histórica como Argentina, por lo que no puede abordarse el análisis de la constitución genética de la población del país sin dar cuenta de las particularidades regionales.
AGRADECIMIENTOS
A los dadores de sangre que han participado y dado su consentimiento para la realización de este estudio.
Este trabajo recibió el apoyo fi nanciero del Consejo Nacional de Investigaciones Científicas y Técnicas (CONICET) y de la Secretaría de Ciencia y Técnica de la Universidad de Buenos Aires (UBACyT).
BIBLIOGRAFÍA CITADA
1. Andrews G (1989). Los afroargentinos de Buenos Aires. Ediciones de la Flor. Buenos Aires. [ Links ]
2. Alfaro EL, Dipierri JE, Gutiérrez NI, Vullo CM (2005). Genetic structure and admixture in urban populations of the Argentine North- West. Ann Hum Biol. 32(6):724-737. [ Links ]
3. Avena SA, Goicoechea AS, Dugoujon JM, Rey J, Dejean CB, Carnese FR (2006). Mezcla génica en la Región Metropolitana de Buenos Aires. Medicina (B Aires). 66:113-118. [ Links ]
4. Avena SA, Goicoechea AS, Bartomioli M, Fernández V, Cabrera A, Dugoujon JM, Dejean CB, Fabrykant G, Carnese FR (2007). Mestizaje en el sur de la región pampeana (Argentina). Su estimación mediante el análisis de marcadores proteicos y moleculares uniparentales. Rev Arg Antr Biol. 9(2):59-76. [ Links ]
5. Avena SA, Parolin ML, Dejean CB, Ríos Part MC, Fabrikant G, Goicoechea AS, Dugoujon JM, Carnese FR (2009). Mezcla génica y linajes uniparentales en Comodoro Rivadavia (Prov. de Chubut, Argentina). Rev Arg Antr Biol. 11(1):25-42. [ Links ]
6. Avena SA, Fejerman L, Via M, Dejean CB, Beckman KB, Pérez-Stable E, Gonzalez Burchard E, Ziv E, Parolin ML, Acreche N, Boquet M, Rios Part MC, Fernández V, Rey J, Carnese FR (2010). Ancestría, mezcla génica y estructuración poblacional en muestras cosmopolitas de argentina. Simposio de Genética de Poblaciones Neoamericanas. XI Congreso de la Asociación Latinoamericana de Antropología Biológica. Bogotá [ Links ].
7. Bailliet G, Rothhammer F, Carnese FR, Bravi CM y Bianchi NO (1994) Founder mitochondrial haplotypes in Amerindian populations. Am. J. Hum. Genet. 55(1):27-33. [ Links ]
8. Bailliet G, Ramallo V, Muzzio M, García A, Santos MR, Alfaro EL, Dipierri JE, Salceda S, Carnese FR, Bravi CM, Bianchi NO, Demarchi DA (2009). Restricted geographic distribution for Y-Q* paragroup in South America. American Journal of Physical Anthropology 140(3):578-582. [ Links ]
9. Baleotti W, Rios JM, Reid ME, Fabron A, Pellegrino J, Saad ST, Castilho L. (2003). A novel DI*A allele without the band 3-Memphis mutation in Amazonian Indians. Vox Sang. 84:326-330 [ Links ]
10. Bedoya G, Montoya P, Garcia J, Soto I, Bourgeois S, Carvajal L, Labuda D, Alvarex V, Ospina J, Hedrick PW, Ruiz-Linares A (2006). Admixture dynamics in Hispanics: a shift in the nuclear genetic ancestry of a South American population isolate. Proc Natl Acad Sci. 103(19):7234-7239. [ Links ]
11. Bianchi NO, Bailliet G, Bravi CM, Carnese RF, Rothhammer F, Martinez-Marignac VL, Pena SD (1997). Origin of Amerindian Y-chromosomes as inferred by the analysis of six polymorphic markers. Am J Phys Anthropol. 102(1):79-89. [ Links ]
12. Bortolini MC, Da Silva WA Junior W, De Guerra DC, Remonatto G, Mirandola R, Hutz MH, Weimer TA, Silva MC, Zago MA, Salzano FM (1999). African-derived South American populations: A history of symmetrical and asymmetrical matings according to sex revealed by bi- and uni-parental genetic markers. Am J Hum Biol. 11(4):551-563. [ Links ]
13. Bravi CM (2004). Análisis de Linajes Maternos en Poblaciones Indígenas Americanas. Tesis Doctoral. Facultad de Ciencias Naturales y Museo. Universidad Nacional de La Plata. [ Links ]
14. Carnese FR, Goicoechea AS; Rey JA, Niborski RC, Kohan AI y Arrayago A (1993). Grupos sanguíneos en una población Mapuche de Blancura Centro, Provincia de Río Negro, Argentina. Bol Soc Esp Antrop Biol. 14:31- 39. [ Links ]
15. Carnese FR, Avena SA, Goicoechea AS, Dejean CB, Sevin A, Dugoujon JM (2003) Sistemas Gm y Km en poblaciones aborígenes de la Patagonia Argentina. En: Antropología y Biodiversidad. (Aluja MP, Malgosa A y Nogués RM Eds.). Ed. Bellaterra. Barcelona, España. 2:67-73. [ Links ]
16. Carnese FR (2006). Grupos étnicos y marcadores genéticos uniparentales en muestras poblacionales neoamericanas. Etnicidad, autodefi nición y genética. 52º Congreso Brasilero de Genética. Foz do Iguazú, Brasil. [ Links ]
17. Censo Nacional de Población, Hogares y Vivienda (2001). Instituto Nacional de Estadísticas y Censos. Dirección en internet: www.indec.gov.ar/webcenso/index.asp [ Links ]
18. Chakraborty R (1985). Gene identity in racial hybrids and estimation of admixture rates. En Ahuja JR, Neel JV (Eds.): Genetic differentiation in human and other animal populations. Delhi, Indian Anthropological Association, pp.171-180. [ Links ]
19. Chen YS, Olckers A, Schurr TG, Kogelnik AM, Huoponen K y Wallace DC (2000). mtDNA variation in the South African Kung and Khwe-and their genetic relationships to other African populations. Am J Hum Genet. 66:1362-1383. [ Links ]
20. Corach D, Lao O, Bobillo C, van Der Gaag K, Zuniga S, Vermeulen M, van Duijn K, Goedbloed M, Vallone PM, Parson W, de Knijff P, Kayser M (2010). Inferring continental ancestry of argentineans from Autosomal, Ychromosomal and mitochondrial DNA. Ann Hum Genet. 74(1):65-76. [ Links ]
21. Dirección Provincial de Estadística del Chubut (2010). Dirección en internet: http://www.estadistica.chubut.gov.ar/publicaciones/informes-tematicos/migraciones.pdf [ Links ]
21. Dejean CB, Goicoechea AS, Avena SA, Salaberry MT, Slepoy AS, Carnese FR (2003). Linajes mitocondriales amerindios en una muestra poblacional de la Región Metropolitana de Buenos Aires. Sextas Jornadas Nacionales de Antropología Biológica. Universidad Nacional de Catamarca. [ Links ]
22. Dipierri JE, Alfaro E, Martínez-Marignac VL, Bailliet G, Bravi CM, Cejas S, Bianchi NO (1998). Paternal directional mating in two Amerindian subpopulations located at different altitudes in northwestern Argentina. Hum Biol. 70(6):1001-1010. [ Links ]
23. Edwards AWF (1984). Likelihood. Paperback edition. Cambridge University Press. Cambridge [ Links ]
24. Excoffier L, Laval LG, Schneider S (2005). Arlequin ver. 3.1: an integrated software package for population genetics data analysis. Evol Bioinf Online. 1:47-50. [ Links ]
25. Felsenstein J (2004). PHYLIP (Phylogeny Inference Package) version 3.6. Washington. Department of Genome Sciences and Department of Biology University of Washington [ Links ]
26. Field LL, Dugoujon JM (1989). Immunoglobulin allotyping (Gm and Km) of GAW5 Families. Gen Epidem. 6:31-34. [ Links ]
27. Finkelstein Débora y Maria Marta Novella (2006) Actividades económicas y proceso de construcción social en las áreas andinas de Rio Negro y Chubut. En: Hecho en Patagonia. La historia en perspectiva regional. (Bandieri Susana, Graciela Blanco y Gladys Varela, Eds.). Ed. Educo - Universidad Nacional del Comahue. Neuquén. pp. 191-209. [ Links ]
28. García A, Demarchi DA (2006). Linajes parentales amerindios en poblaciones del norte de Córdoba. Rev Arg Antr Biol. 8(1):57-71. [ Links ]
29. García F, Moraga M, Vera S, Henríquez H, Llop E, Aspillaga E, Rothhammer F (2006). mtDNA microevolution in Southern Chile's archipelagos. Am J Phys Anthropol. 129(3):473-481. [ Links ]
30. Gavirati Marcelo, Nelcis Jones y Fernando Coronato (2006) Los galeses en la Patagonia: una singular experiencia de colonización. En: Hecho en Patagonia. La historia en perspectiva regional. (Bandieri Susana, Graciela Blanco y Gladys Varela, Eds.). Ed. Educo - Universidad Nacional del Comahue. Neuquén. pp. 279-299. [ Links ]
31. Ginther C, Corach D, Penacino GA, Rey JA, Carnese FR, Hutz MH, Anderson A, Just J, Salzano FM, King MC (1993). Genetic variation among the Mapuche Indians from the Patagonian region of Argentina: mitochondrial DNA sequence variation and allele frequencies of several nuclear genes. EXS. 67:211-219. [ Links ]
32. Goicoechea AS, Carnese FR, Dejean CB, Avena SA, Weimer TA, Franco MH, Callegari- Jacques MS, Estalote A, Simoes ML, Palatnik M, Salomoni P, Salzano FM (2001) Genetic Relationships Between Amerindian Populations of Argentina. Am J Phys Anthropol. 115:133-43. [ Links ]
33. Lalueza C, Pérez-Pérez A, Prats E, Cornudella L, Turbón D (1997). Lack of founding Amerindian mitochondrial DNA lineages in extinct aborigines from Tierra del Fuego-Patagonia. Hum Mol Genet. 6(1):41-46 [ Links ]
34. Lopez Camelo JS, Cabello PH, Dutra (1996). A simple model for the estimation of congenital malformation frequency in admixed populations . Braz J Genet. 19(4):659-663. [ Links ]
35. Lorandi AM (1992). El mestizaje interétnico en el noroeste argentino. Senri Ethnological Studies. 33:133-166. [ Links ]
36. Marino M, Sala A, Corach D (2007). Genetic attributes of the YHRD minimal haplotype in 10 provinces of Argentina. Forensic Sci Int Genet. 1(2):129-133. [ Links ]
37. Marrero AR, Silva-Junior WA, Bravi CM, Hutz MH, Petzl-Erler ML, Ruiz-Linares A, Salzano FM, Bortolini MC (2007). Demographic and evolutionary trajectories of the Guarani and Kaingang natives of Brazil. Am J Phys Anthropol. 132(2):301-310. [ Links ]
38. Martinez Marignac VL, Bertoni B, Parra EJ, Bianchi NO (2004). Characterization of admixture in an urban sample from Buenos Aires, Argentina, using uniparentally and biparentally inherited genetic markers. Hum Biol. 76(4):543-557. [ Links ]
39. Martínez Marignac VL, Bravi CM, Lahitte HB, Bianchi NO (1999). Estudio del ADN mitocondrial de una muestra de la ciudad de La Plata. Rev Arg Antr Biol. 2:281-300. [ Links ]
40. Merriwether DA, Rothhammer F, Ferrell RE (1995). Distribution of the four founding lineage haplotypes in Native Americans suggests a single wave of migration for the New World. Am J Phys Anthropol. 98(4):411-430. [ Links ]
41. Moraga ML, Rocco P, Miquel JF, Nervi F, Llop E, Chakraborty R, Rothhammer F, Carvallo P (2000). Mitochondrial DNA polymorphisms in Chilean aboriginal populations: implications for the peopling of the southern cone of the continent. Am J Phys Anthropol. 113(1):19-29. [ Links ]
42. Novella MM (2005). Composición poblacional del oeste chubutense en 1895. La estadística censal y la"creación" de la Nación en la frontera. Pueblos y Fronteras. 6(6):22-31. [ Links ]
43. Oriola J (2003). Esquel del telégrafo al pavimento. 1º edición. Honorable Senado de la Nación. Esquel. [ Links ]
44. Page RD (1996). Treeview: An application to display phylogenetic trees on personal computers. Comp Appl Biosci. 12:357-358. [ Links ]
45. Parolin ML, Avena SA, Dejean CB y Carnese FR (2010). Análisis de linajes paternos en dos localidades de la Patagonia Argentina (Prov. de Chubut). XI Congreso de la Asociación Latinoamericana de Antropología Biológica. Bogotá [ Links ].
46. Pogo AO, Chaudhuri A (2000). The Duffy protein: a malarial and chemokine receptor. Semin Hematol. 37 (2): 122-129. [ Links ]
47. Ramallo V, Salceda S, Zubrzycki B, Bravi C, Bailliet (2007). Genealogías y linajes moleculares en Azampay. Rev Arg Antr Biol. 9(1):32. [ Links ]
48. Reed T, Schull W (1968). A general maximun likelihood estimation program. Am J Hum Genet. 20:579-580. [ Links ]
49. Rocco P, Morales C, Moraga M, Miquel JF, Nervi F, Llop E, Carvallo P, Rothhammer F (2002). Composición genética de la población chilena: Distribución de poliformismos de DNA mitocondrial en grupos originarios y en la población mixta de Santiago. Rev Med Chil. 130(2):125-131. [ Links ]
50. Sambrook J, Fritisch EF, Maniatis T (1989). Molecular Cloning a laboratory Manual. 2nd Edition. Cold Spring Harbor Laboratory Press, Cold Spring harbor, New York. [ Links ]
51. Santos SEB, Rodrigues JD, Ribeiro-dos-Santos A, Zago MA (1999). Differential Contribution of Indigenous Men and Womem to the Formation of the Urban Population in the Amazon Region as Revealed by mtDNA and Y-DNA. Am J Phys Anthropol. 109:175-180. [ Links ]
52. Sans M, Weimer TA, Franco MH, Salzano FM, Bentancor N, Alvarez I, Bianchi NO, Chakraborty R (2002). Unequal contributions of male and female gene pools from parental populations in the African descendants of the city of Melo, Uruguay. Am J Phys Anthropol. 118(1):33-44. [ Links ]
53. Seldin MF, Tian C, Shigeta R, Scherbarth HR, Silva G, Belmont JW, Kittles R, Gamron S, Allevi A, Palatnik SA, Alvarellos A, Paira S, Caprarulo C, Guillerón C, Catoggio LJ, Prigione C, Berbotto GA, García MA, Perandones CE, Pons-Estel BA, Alarcon-Riquelme ME (2007). Argentine population genetic structure: large variance in Amerindian contribution. Am J Phys Anthropol. 132(3):455- 462. [ Links ]
54. Sneath PH, Sokal RR (1973). Numerical taxonomy. The principles and practice of numerical classifi cation. (Freeman WH & Co), San Francisco, California. p. 230-234. [ Links ]
55. Stone AC, Stoneking M (1993). Ancient DNA from a pre-Columbian Amerindian population. Am J Phys Anthropol. 92(4):463-471 [ Links ]
56. Tournamille C, Colin Y, Cartron JP, Le Van Kim C (1995). Disruption of a GATA motif in the Duffy gene promoter abolishes erythroid gene expression in Duffy-negative individuals. Nat Genet. 10(2):224-228. [ Links ]
57. Torroni A, Huoponen K, Francalacci P, Petrozzi M, Morelli L, Scozzari R, Obinu D, Savontaus ML, Wallace D (1996). Classifi cation of European mtDNAs from an Analysis of Three European populations. Genetics. 144(4):1835-1850. [ Links ]
58. Troiano M (1993). Y así nació Esquel. 1ª edición. Imprenta FB. Esquel. [ Links ]
59. Underhill PA, Jin L, Zemans R, Oefner PJ, Cavalli- Sforza LL (1996). A pre-Columbian Y chromosome-specifi c transition and its implications for human evolutionary history. Proc Natl Acad Sci. 93:196-200. [ Links ]
60. Wang S, Ray N, Rojas W, Parra MV, Bedoya G, Gallo C, Poletti G, Mazzotti G, Hill K, Hurtado AM, Camrena B, Nicolini H, Klitz W, Barrantes R, Molina JA, Freimer NB, Bortolini MC, Salzano FM, Petzl-Erler ML, Tsuneto LT, Dipierri JE, Alfaro EL, Bailliet G, Bianchi NO, Llop E, Rothhammer F, Excoffi er L, Ruiz-Linares A (2008). Geographic Patterns of Genome Admixture in Latin American Mestizos. PLoS Genet 4(3):10.1371/journal.pgen.1000037 [ Links ]














