Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
BAG. Journal of basic and applied genetics
versión On-line ISSN 1852-6233
BAG, J. basic appl. genet. vol.21 no.1 Ciudad Autónoma de Buenos Aires ene./jun. 2010
ARTÍCULOS ORIGINALES
Análisis del efecto de polimorfismos de un solo nucleótido del Gen Fasn en novillos
Marrube G.1#, Soria LA1, Rozen FMB1, Corva PM2, Schor A3, Melucci LM2, Villarreal EL2, Mezzadra CA2, Miquel MC1
1Cátedra de Genética. Facultad de Ciencias Veterinarias, UBA. Av. Chorroarín 280 CABA (1427).
2Unidad Integrada EEA-INTA Balcarce/Facultad de Ciencias Agrarias, UNMDP. Ruta Nacional 226, Km. 73,5 CC 276 (7620) Balcarce, Pcia Buenos Aires.
3Bovinos de Carnes, Facultad de Agronomía, UBA. Av. San Martín 4453 CABA (1417)
# e-mail: gmarrube@fvet.uba.ar
RESUMEN
La enzima Acido Graso Sintasa (FASN) cataliza la síntesis de los ácidos grasos en la célula y presenta un rol fundamental en el metabolismo de los lípidos. Con el propósito de analizar el efecto de dos polimorfismos de un solo nucleótido (SNPs) en el gen FASN (G>C en el exón 1 y A>G en el exón 34) se analizaron los siguientes caracteres productivos: Peso Vivo Final, Ganancia Diaria de Peso Vivo, Tasa Mensual de Engrasamiento, Grasa Riñonada, Porcentaje de Grasa Riñonada, Rendimiento de la Canal, Porcentaje de Grasa Intramuscular, en novillos de razas Angus (n = 20) y Brangus (n = 60). Se desarrollaron técnicas de mismatch PCR para determinar los genotipos de ambos SNPs. El análisis estadístico se realizó mediante un modelo de efectos fijos que incluyó genotipo del SNP y grupo genético. Para el SNP del exón 1 las diferencias fueron significativas entre genotipos (p<0,05) para Grasa Riñonada y Porcentaje de Grasa Riñonada en tanto que entre grupos genéticos las diferencias fueron para todos los caracteres (p<0,05). De acuerdo a los resultados obtenidos, el alelo C de dicho SNP es el que se encuentra asociado a una menor cantidad de Grasa Riñonada y Porcentaje de Grasa Riñonada. Debido a la ausencia de animales con genotipo homocigota AA y a la baja frecuencia de heterocigotas, no se pudo realizar el análisis de asociación para el polimorfismo del exón 34.
Palabras clave: Bovinos para carne; Calidad de la carne; Gen FASN; SNPs
ABSTRACT
Fatty acid synthase (FASN) is an enzyme that catalyzes the synthesis of fatty acids in the cell and has a key role in lipid metabolism. For the purpose of analyzing the effect of two Single Nucleotide Polymorphisms (SNPs) in the FASN gene (G>C in exon 1 and A>G in exon 34) the following productive traits were analyzed: Final Live Weight, Live Weight Daily Gain, Fat Thickness Monthly Gain, Kidney Fat, Kidney Fat Percentage, Carcass Yield, Intramuscular Fat Percentage, in Aberdeen Angus (n = 20) and Brangus (n = 60) steers. Mismatch PCR techniques were developed to determine both SNPs genotypes. Phenotypic data were analysed with a model that included the fi xed effects of SNP genotypes and genetic group. For the SNP in exon 1 signifi cant differences between genotypes (p<0,05) were found in Kidney Fat and Kidney Fat Percentage, while among genetic groups differences were found in every trait (p<0,05). According to the results obtained, the C allele from this SNP is associated with less Kidney Fat and lower Kidney Fat Percentage. Due to the absence of homozygous AA animals and the low frequency of heterozygous animals, statistical analyses for polymorphism in exón 34 could not be performed.
Key words: Beef cattle; Meat quality; FASN gene; SNPs
INTRODUCCIÓN
La enzima Ácido Graso Sintasa (FASN) es una proteína multifuncional que cataliza la síntesis de los ácidos grasos en la célula. Debido a su rol en el metabolismo de los lípidos, el gen FASN ha sido estudiado como candidato para explicar la variabilidad de diferentes caracteres productivos (Sourdioux et al., 1999; Roy et al., 2006; Morris et al., 2007; Zhang et al., 2008; Abe et al., 2009). Un polimorfismo en dicho gen ha sido asociado con el grado de engrasamiento en pavos (Sourdioux et al., 1999). El gen FASN en bovinos se localiza en el cromosoma 19 (19q22) (Roy et al., 2001). Se han identificado en la secuencia genómica correspondiente al número de acceso de GeneBank AF285607, dos SNPs: uno en el exón 1 no traducido (841G>C) que modifica un sitio TFBS (Transcription Factor Binding Site) para miembros de la familia de factores de transcripción Sp y que ha sido asociado con contenido de grasa en leche bovina y otro en el exón 34 (16009A>G), que determina una sustitución no conservativa de treonina por alanina (Thr1953Ala) en dicha proteína (Roy et al., 2006; Ordovás et al., 2009). Recientemente se han informado otros polimorfismos en diferentes regiones del gen bovino con efecto sobre la composición de ácidos grasos en distintos depósitos de tejido adiposo corporal y también en la leche (Morris et al., 2007; Zhang et al., 2008; Abe et al., 2009). El objetivo de este trabajo fue desarrollar métodos para determinar genotipos de los SNPs localizados en el exón 1 y en el exón 34 del gen FASN y relacionar el efecto de dichos polimorfismos sobre características del engorde, la faena y la calidad de la carne en novillos de las razas Angus y Brangus.
MATERIALES Y MÉTODOS
Animales
Se utilizaron 20 novillos Angus y 60 novillos Brangus. Los Angus pertenecían a dos rodeos diferentes de la Estación Experimental INTA Balcarce. Diez novillos (grupo 1) pertenecían a un rodeo de vacas INTA y toros Angus comerciales y los restantes (grupo 2) al rodeo original de dicha estación, el cual no ha sido sometido a selección por crecimiento. Los novillos Brangus (grupo 3, 4 y 5) fueron aportados por tres cabañas pertenecientes a la Asociación Argentina de Brangus. Los 80 novillos fueron engordados en pasturas polifíticas durante el período comprendido entre abril de 2004 y mayo de 2005 en la Reserva 7 de la Unidad Integrada Balcarce (Balcarce, Provincia de Buenos Aires).
Información Fenotípica
Se registraron los siguientes datos de engorde y calidad de carne: Peso de Faena (PF, kg), Ganancia Diaria de Peso Vivo (GDPV, kg/d), Tasa Mensual de Engrasamiento (TE, mm/mes), Grasa Riñonada (GR, kg), Porcentaje de Grasa Riñonada (PGR, %), Rendimiento de la Canal (R, %) y Porcentaje de Grasa Intramuscular (PGI, %). El Peso Vivo se registró a la llegada de los terneros a la Estación Experimental INTA Balcarce y luego cada 28 días hasta la faena. Mensualmente se midió por ultrasonido el Espesor de Grasa Dorsal a la altura del intervalo entre las costillas 12ª y 13ª. La Ganancia Diaria de Peso se calculó como la regresión del Peso Vivo con respecto al número de días en el ensayo, en tanto que la Tasa Mensual de Engrasamiento se calculó de manera similar a partir del Espesor de Grasa Dorsal. Cada grupo fue sacrificado cuando al menos el 50 % de sus integrantes alcanzó un Espesor de Grasa Dorsal de 6 mm entre las 12º y 13º costillas. El Peso de la Canal y de la Grasa Riñonada se midieron en el frigorífico y el Rendimiento de la Canal se calculó como la relación: Peso de Canal Caliente*100)/Peso de Faena. El peso de la Grasa Riñonada se expresó en valor absoluto y en porcentaje con respecto al peso de la canal caliente. Las carcasas fueron conservadas durante 24 horas a 10-12 ° C. Al día siguiente se extrajo un bloque de bifes correspondiente a las costillas 11ª, 12ª y 13ª de cada media canal izquierda fría. Éste se deshuesó y se sometió a tratamientos de maduración. Luego de la maduración, los cortes se mantuvieron a -20° C hasta que se descongelaron para su estudio. El Porcentaje de Grasa Intramuscular (g/100g de tejido fresco) se realizó en el Laboratorio de Carnes de la Facultad de Agronomía de la Universidad de Buenos Aires, según el protocolo 920.39 de AOAC (Association of Official Analytical Chemists, 1992) en los cortes con 1 día de maduración.
Genotipificación
El aislamiento del ADN se realizó a partir de 500 μl de sangre con fenol-cloroformo y precipitación con etanol. Se analizaron las sustituciones G>C en el exón 1 y A>G en el exón 34 del gen FASN. Debido a que ambas mutaciones no presentan sitio de reconocimiento para endonucleasas de restricción, se utilizó el método mismatch PCR (Haliassos, et al., 1989) con el propósito de crear una secuencia de corte. Se diseñaron cebadores degenerados mediante el programa dCAPS Finder 2.0 (http://helix.wustl.edu/dcaps/dcaps. html) a partir de la secuencia de ADN publicada (N° de acceso al GenBank: AF285607). En el exón 1 la sustitución G>C es detectada por un cebador directo degenerado que crea un sitio de reconocimiento para la endonucleasa BanII. El cebador directo para el exón 34 presenta una sustitución para crear un sitio de corte para la enzima BsiHKAI (Tabla I).
Tabla I: Secuencias de los cebadores y condiciones de PCR para los SNPs analizados en el gen FASN
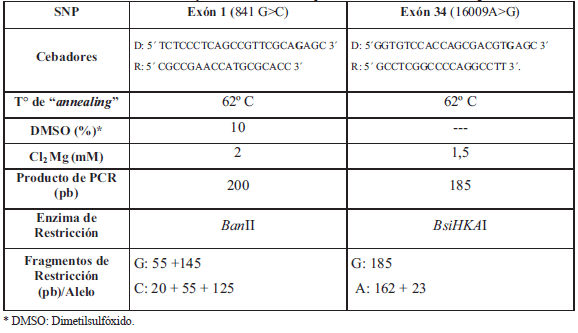
La reacción de PCR para ambos exones se desarrolló en un volumen final de 25 μl para cada muestra, y los amplicones se visualizaron en geles de agarosa al 2 % teñidos con Bromuro de Etidio. Los productos de digestión, fueron visualizados en geles de agarosa al 3,5 % en TBE 0,5X y fotografiados bajo luz UV.
Análisis estadístico
Los datos fenotípicos se analizaron independientemente por ANOVA según el siguiente Modelo de Efectos Fijos:
Yijn = μ + CORi + Gj + eijn
Donde:
Yijn: variable dependiente
μ: media de la población
CORi: efecto grupo genético (clasificación de raza/origen)
Gj: efecto del genotipo (SNP) del animal
eijn: vector aleatorio de los errores
RESULTADOS
Mediante los métodos de mismatch PCR diseñados en este trabajo se pudieron determinar los genotipos de ambos SNPs. Las frecuencias genotípicas calculadas para el exón 34 fueron AG: 0,08 y GG: 0,92 siendo estos valores semejantes a los informados en ganado bovino de raza Holstein (Roy et al., 2006). Debido a la ausencia de novillos con genotipo homocigota AA y a la baja frecuencia de individuos heterocigotas, no se realizó el análisis de asociación con los caracteres productivos de interés.
En la Tabla II, se presentan las estimaciones de media fenotípica, desvío estándar, coeficiente de variación (CV%) de los atributos y coeficiente de determinación del modelo (R2) al analizar el efecto de los genotipos del exón 1. Para dicho exón y teniendo en cuenta los genotipos analizados en la Tabla III se muestran sus frecuencias y las respectivas medias y desvíos estándares de los atributos. Del mismo modo, en la Tabla IV se muestran las medias y desvíos estándares para los caracteres analizados considerando el grupo según raza y origen.
Tabla II: Media Fenotípica, Desvío Estándar, Coeficiente de Variación y Coeficiente de Determinación del modelo para los atributos analizados.
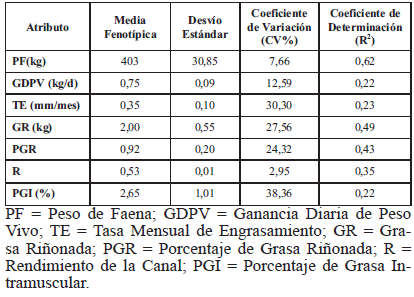
Tabla III: Frecuencia Genotípica, Media y Desvío Estándar para cada atributo, según genotipo del SNP del exón 1 (841 G>C) del gen FASN.

Tabla IV: Media y Desvío Estándar para cada atributo analizado según grupo genético.
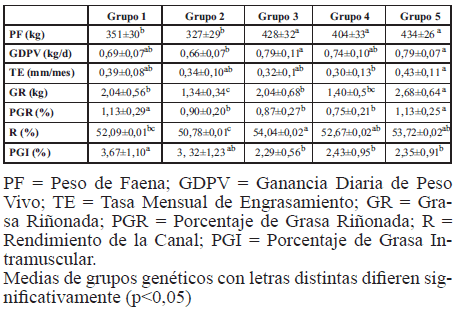
Se detectaron diferencias significativas entre genotipos (Tabla III) en GR y PGR (p<0,05). Si bien la frecuencia de animales con genotipo CC es muy baja (0,08) se hallaron homogéneamente distribuidos entre los distintos grupos genéticos, sólo el grupo 4 no presentó ningún animal con genotipo CC. Entre grupos genéticos se detectaron diferencias significativas (p<0,05) en todos los caracteres analizados (Tabla IV).
DISCUSIÓN
Las comparaciones entre medias de genotipos del SNP del exón 1 para las características analizadas, sugieren que el alelo C está claramente asociado a una menor cantidad y proporción de grasa riñonada.
El efecto significativo de grupo genético entre los Angus (grupo 1 y 2) podría ser atribuido a las diferencias de biotipo, debido a que uno de ellos (grupo 1) tiene padres comerciales, existiendo diferencias en tamaño corporal. En el caso de los Brangus, uno de los orígenes (grupo 5) tiene mayor porcentaje Angus (>62,5%) que los dos restantes (grupo 3 y 4) (Tabla IV). Sin embargo, los resultados también podrían estar influenciados por el bajo número de animales en cada grupo genético.
Dado que las estimaciones de la correlación genética (rg) entre grasa abdominal y grasa subcutánea son bajas (rg = -0,08 a 0,05) (Bergen et al., 2006), dichos valores indicarían que los depósitos de grasa corporal podrían estar bajo el control de diferentes genes o de diferentes patrones de regulación de un mismo gen. Esto permitiría explicar por qué en este trabajo se obtuvieron diferencias significativas asociadas al SNP del exón 1 sobre GR y PGR y no en otros depósitos grasos en los novillos (Tabla III).
La importancia como marcadores genéticos, de algunos SNPs se debe a que tienen consecuencias funcionales por localizarse en regiones regulatorias (rSNP), constituyendo las bases de los diferentes perfiles de expresión génica. La sustitución G>C en el exón 1 altera una secuencia GC localizada en la posición +60/+72 desde el sitio de inicio de la transcripción, la cual sería un potencial TFBS para la familia de factores de transcripción Sp.
Estudios realizados en diferentes líneas celulares y extractos nucleares de glándula mamaria en lactación demuestran que ambos alelos son funcionalmente distintos. Existiría una diferente afinidad de los miembros de la familia Sp a la secuencia +60/+72, de modo que la unión de Sp1 al alelo C reprime la actividad del promotor de FASN, en tanto que su unión con Sp3 lo activaría. Por lo tanto, la relación entre Sp1/Sp3 estaría modulando la actividad del promotor del gen FASN. En la glándula mamaria del bovino durante la lactancia, la sustitución G>C aumenta la afinidad por proteínas nucleares, pero no se ha hallado represión de la actividad del promotor debida a Sp1, sólo se ha encontrado al alelo C asociado con un mayor contenido de grasa en leche, mediado por Sp3 (Ordovás et al., 2008).
De acuerdo a los resultados actuales, se podría afirmar que existe una expresión del gen FASN tejido-específica que no sólo depende de la presencia del alelo G o C del SNP 841, sino también de los miembros de la familia de factores de transcripción Sp que se encuentren unidos a la secuencia en la posición +60/+72. Esta podría ser una de las razones de la discrepancia hallada entre el efecto del alelo C sobre contenido de grasa en leche (Ordovás et al., 2008; Roy et al., 2006) y el resultado del presente trabajo, donde se observó al alelo C asociado a una menor cantidad de GR y PGR en novillos.
CONCLUSIONES
Los resultados obtenidos sugieren que el alelo C del SNP del exón 1 del gen FASN (841G>C) estaría asociado con menor contenido de Grasa Riñonada en novillos engordados en pasturas. Este marcador por lo tanto podría ser integrado a estrategias de selección asistida (SAM). No es el caso del otro marcador evaluado (exón 34) ya que en las razas utilizadas la falta de variación no permitió realizar el análisis de asociación correspondiente.
AGRADECIMIENTOS
El presente trabajo ha sido financiado por la ANPCYT (Agencia Nacional de Promoción Científica y Tecnología). Los animales fueron provistos por INTA Balcarce y criadores miembros de la Asociación Argentina de Brangus.
REFERENCIAS
1. Association of Official Analytical Chemists (1992). Offi cial Methods of Analysis of the Association of Official Analytical Chemistry. 15th Ed. Association of Official Analytical Chemists, Washington 139-140. [ Links ]
2. Abe, T.; Saburi, J.; Hasebe, H.; Nakagawa, A.; Misumi, S.; Nade, T.; Nakajima, H.; Shoji, N.; Kobayashi M.; Kobayashi, E. (2009). Novel mutation of the FASN gene and their effect on fatty acid composition in Japanese Black Beef. Biochem Genet.; 47: 397-411. [ Links ]
3. Bergen, R.; Miller, S.P.; Wilton, J.W.; Mandell, I.B. (2006). Genetic correlations between live yearling bull and steer carcass traits adjusted to different slaughter end points. 2. Carcass fat partitioning. J Anim Sci.; 84: 558-566. [ Links ]
4. Haliassos, A.; Chomel, J.C.; Tesson, L.; Baudis, M.; Kruh, J; Kapalan, J.C.; Kitzis, A. (1989). Modification of enzymaticaly amplified DNA for the detection of point mutations. Nucleic Acids Res.; 17: 3606. [ Links ]
5. Morris, C.A.; Cullen, N.G.; Glass, B.C.; Hyndman, D.L.; Manley, T.R.; Hickey, S.M.; McEwan, J.C.; Pitchford, W.S.; Bottema, C.D.K.; Lee, M.A.H. (2007). Fatty acid synthase effects on bovine adipose fat and milk fat. Mamm Genome; 18: 64-74. [ Links ]
6. Ordovás, L.; Roy, R.; Pampín, S.; Zaragoza, P.; Osta, R.; Rodriguez -Rey, C.; Rodellar, C. (2008). The g.763G>C SNP of the bovine FASN gene affects its promoter activity via Sp-mediated regulation: implications for the bovine lactating mammary gland. Physiol Genomics; 34: 144-148. Erratum in: Physiol Genomics. 2009 36:127. [ Links ]
7. Roy, R.; Gautier , M.; Hayes, H.; Laurent, P.; Osta, R.; Zaragoza, P.; Eggen; A.; Rodella; C. (2001). Assignment of the fatty acid synthase (FASN) gene to bovine chromosome 19 (19q22) by in situ hybridization and confirmation by somatic cell hybrid mapping. Cytogenet Cell Gen.; 93: 141-142. [ Links ]
8. Roy, R.; Ordovás, L.; Zaragoza, P.; Romero, A.; Moreno, C.; Altarriba, J.; Rodellar, C. (2006). Association of polymorphisms in the bovine FASN gene with milk-fat content. Anim. Genet.; 37: 215-218. [ Links ]
9. Sourdioux, M.; Brevelet, C.; Delabrosse, Y.; Douaire, M. (1999). Association of Fatty Acid Synthase gene and Malic Enzyme gene polymorphisms with fatness in turkey. Poultry Sci.; 78: 1651-1657. [ Links ]
10. Zhang, S.; Knight, T.J.; Reecy; J. M.; Beitz D. C. (2008). DNA polymorphisms in bovine fatty acid synthase are associated with beef fatty acid composition. Anim. Genet.; 39: 62-70. [ Links ]














