Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO  uBio
uBio
Share
BAG. Journal of basic and applied genetics
On-line version ISSN 1852-6233
BAG, J. basic appl. genet. vol.21 no.2 Ciudad Autónoma de Buenos Aires July./Dec. 2010
ARTÍCULOS ORIGINALES
La genética molecular de bovinos y equinos criollos en los albores del siglo XXI
Giovambattista G. 1, Rogberg Muñoz A. 1,2 , Ripoli M.V. 1, Villegas Castagnasso E. E. 1, Díaz S. 1, Posik D. M. 1,3, Lirón J.P. 1, Carino M.H. 1,3, Goszczynski D. E. 1,2, Fernández M.E. 1,4, P. Peral García 1
¹ Instituto de Genética Veterinaria (IGEVET), Facultad de Ciencias Veterinarias, Universidad Nacional de La Plata. CCT La Plata-CONICET. Calle 60 y 118 S/N, P.O. Box 296, B1900AVW La Plata, Argentina;
² Consejo Nacional de Investigaciones Científicas y Tecnológicas (CONICET), Buenos Aires, Argentina;
³ Comisión de Investigaciones Científicas de la Provincia de Buenos Aires (CICBA), La Plata, Argentina;
4 Agencia Nacional de Promoción Científica y Tecnológica (ANPCYT).
ggiovam@fcv.unlp.edu.ar
RESUMEN
Los bovinos y equinos criollos americanos son descendientes directos de los animales introducidos al Nuevo Mundo por los europeos durante los siglos XV y XVI. En los comienzos de la década pasada, surgieron tecnologías basadas en el estudio del ADN que permitieron profundizar el análisis genético, por su parte, las especies criollas americanas no quedaron fuera de dichos estudios. En los últimos años, estas metodologías evolucionaron en forma vertiginosa, y en poco tiempo, el aumento de información creció significativamente de modo tal que las investigaciones pasaron de unos pocos loci a la secuenciación y el análisis de los genomas completos de ambas especies. En el presente trabajo se describe como ha evolucionado el estado del arte de la genética molecular de bovinos y equinos criollos en los últimos 15 años. Si bien las razas criollas aun no han entrado en la era de la genómica y la proteómica, su estudio es valioso dado que actualmente se encuentran en progresiva reducción poblacional. Su importancia radica en que aporta información relacionada con la resistencia a enfermedades infecciosas, la adaptabilidad ante condiciones desfavorables, la genética forense, la producción animal y la filigeografía, entre otras. Además, las razas criollas son un reservorio natural e irremplazable de variabilidad genética, y el conocimiento pormenorizado de ellas es clave para la planificación de estrategias de conservación sustentables a lo largo del tiempo.
Palabras clave: Bovinos criollos; Equinos criollos; Bos taurus; Equus caballus; Filogeografía.
ABSTRACT
American Creole bovines and equines are direct descendant from the animals introduced by European conquerors during XV an XVI centuries. At the beginning of the last decade, new technologies based in DNA emerged for genetic analysis, and Creole breeds were not the exception for these kind of studies. During the last years, these methodologies evolved vertiginously. In a short time, the amount of available information increased, and the analysis of few loci turned into analysis of complete genomes. This work describes the evolution of Creole, cattle and equines, molecular genetic studies during the last fifteen years. Even though, Creole breeds have not entered yet into genomic and proteomic era, its study is relevant because they are suffering a progressive population reduction. In this sense, information about infectious diseases resistance, adaptability, forensic genetics, animal production and phylogeography, among others, can be obtained from them. Furthermore, Creole breeds are a unique natural reservoir of genetic variability, and strategies for its conservation should be implemented.
Key words: American cattle; American equines; Bos taurus; Equus caballus; Phylogeography.
LA GENÉTICA MOLECULAR DE ANIMALES DOMÉSTICOS EN EL PERÍODO 1995-2000
En 1994, la Sociedad Argentina de Genética (SAG) festejaba sus bodas de plata. Para esa ocasión la revista de la SAG (denominada por entonces Mendeliana) publicaba un número especial, en el que se incluía un artículo que reseñaba los estudios sobre marcadores bioquímicos y moleculares en animales domésticos en Argentina (Poli, 1994). Del título y de la lectura de dicha revisión surgen al menos dos aspectos: i) la reducida masa crítica de grupos/investigadores que trabajaban en marcadores genéticos en animales domésticos en el país, medido a través del número de publicaciones citadas; y ii) que los marcadores utilizados por excelencia en esos años eran los polimorfismos bioquímicos y los grupos sanguíneos. En los países latinoamericanos científicamente más avanzados, el estado del arte era similar. Esta situación se ha modificado significativamente durante los últimos quince años.
Esos años fueron tiempos de grandes cambios. A fines de la década de los ochenta y principio de los noventa, se publicaron los primeros trabajos basados en el análisis del ADN, en los que se empleaban principalmente las técnicas de Polimorfismos de la Longitud de los Fragmentos de Restricción (Restriction Fragment Length Polymorphism, RFLP) y Southern blot. Sin embargo, recién con la aparición de la técnica de Reacción en Cadena de la Polimerasa (Polimerasa Chain Reaction, PCR) es que comienzan a popularizarse los trabajos basados en ADN. Muchos conceptos que en esos días eran meramente teóricos actualmente se han vuelto realidad. Así por ejemplo, la secuenciación del genoma, el mapeo de Loci (Quantitative Trait Loci, QTLs) y Nucleótidos de Caracteres Cuantitativos (Quantitative Trait Nucleotides, QTNs), la detección de mutaciones causantes de caracteres cualitativos, y la selección asistida por marcadores genéticos (Marker Assisted Selection, MAS), entre otros.
En 1992, se estableció el proyecto Europeo de Mapeo del Genoma Bovino (BovMap) y en 1993, se publicó el primer mapa de ligamiento bovino (Fries et al., 1993). A esta primera publicación le siguieron otros mapas de ligamiento basados principalmente en el uso de microsatélites (Short Tandem Repeat, STR). En 1997, ya se contaba con un mapa genético de segunda generación del bovino, que incluía más de un millar de marcadores genéticos (Kappes et al., 1997). Los mapas genéticos o de ligamiento, construidos mediantes estudios de segregación, fueron complementados con mapas físicos o citogenéticos construidos mediante técnicas tales como hibridación in situ, hibridación de células somáticas, paneles de células híbridas (Rexroad et al., 2000) y métodos de mapeo comparativo (Fronicke y Wienberg, 2001).
Posteriormente, se publicaron los primeros mapas de consenso y comprehensivos para los cromosomas sexuales y autosómicos (Ej., Casas et al., 2001), los que se construyeron mediante la integración de la información obtenida por los diferentes grupos de investigación de los países participantes. Esto permitió contar con mapas de varios miles de marcadores y una densidad o distancia promedio inferior a 2,5 cM (Kappes et al., 1997). El siguiente avance importante consistió en la publicación en el año 2004 del primer borrador del genoma bovino (versión BTau_1.0) con una cobertura de 3x (http://www.hgsc.bcm.tmc.edu/project-species-m-Bovine.hgsc?pageLocation=Bovine), del cual se cuenta actualmente con la versión BTau_4.2 con una resolución de 7.1x (Bovine Genome Sequencing and Analysis Consortium et al., 2009; Liu et al., 2009).
Por su parte, en 1995, a partir del establecimiento del Taller Internacional Mapeo Genético Equino (Horse Genome Project, http://www.uky.edu/Ag/Horsemap/) se construyeron los primeros mapas genéticos en caballos, pero no fue hasta fines del 2007 que se realizó el primer ensamble de secuencias del genoma. En los últimos 15 años, y en el marco del Proyecto Genoma Equino, se han producido varias generaciones de recursos analíticos y diagnósticos que permiten la búsqueda de polimorfismos a lo largo de todo el genoma equino (Guerin et al., 1999; Penedo et al., 2005). Un paso decisivo hacia el mapeo comparativo ocurrió con el "pintado" de cromosomas humanos en el cariotipo equino (Raudsepp et al. 1996), que fueron aumentando la resolución del mapeo mediante localización de genes con mapas de células hibridas somáticas y radiación hibrida y citogenéticos (Milenkovic et al. 2002; Chowdhary et al. 2003). Los primeros intentos involucraban alrededor de 300 marcadores microsatélites, que permitieron el mapeo de varias enfermedades equinas de origen genético (Valberg et al., 2001; Tryon et al., 2007). Sin embargo, este pequeño número de marcadores limitaba los estudios genéticos en caballos al análisis de rasgos simples en familias con un alto número de muestras. Actualmente, se ha publicado la secuencia completa del genoma del equino, con una resolución de 6.8x.
Estos desarrollos en bovinos y equinos han permitido detectar en la última década numerosos QTLs y QTNs para caracteres de importancia económica tales como crecimiento, calidad de carne, carcasa, cantidad y calidad de leche, resistencia/susceptibilidad a enfermedades infecciosas, perfomance, fertilidad, etc. (Peral García et al., 2007; Casas, 2010; Medrano y Rincón et al., 2010). Una recopilación exhaustiva de los QTLs que se han identificado en el ganado bovino puede obtenerse en las páginas de Internet: http://bovineqtl.tamu.edu/, http://www.animalgenome.org/QTLdb/faq.html. En equinos, un beneficio inmediato fue el descubrimiento de la base genética de numerosos rasgos genéticos simples, que incluyen color de pelaje (Overo, Tobiano, Blanco dominante, genes de dilución) y varias enfermedades hereditarias, tales como Hyperkalemic periodic paralysis (HYPP), Severe Combined Immunodeficiency Disease (SCID), Overo Lethal White Foal Syndrome (OLWFS), para los cuales se desarrollaron test moleculares disponibles comercialmente.
Paralelamente al estudio del genoma, se están llevando a cabo proyectos a gran escala de detección y tipificación de Polimorfismos de Nucleótidos Simples (Single Nucleotide Polymorphisms, SNPs) con el fin de desarrollar mapas de haplotipos a nivel genómico e implementar programas de selección a este nivel (Genomic Selection). La importancia que ha adquirido esta área de investigación se pone de manifiesto a través del gran número de proyectos y países dedicados específicamente al estudio de los SNPs. En este sentido, se han organizado consorcios para el estudio e identificación de SNPs y haplotipos en bovinos (Bovine HapMap Consortium et al., 2009) y equinos (Brooks et al., 2009; Wade et al., 2009) (Broad institute - www.broad.mit.edu/mammals/horse/snp). La disponibilidad masiva de SNPs permitió la construcción de microarrays de SNPs de 25 y 50K en bovinos y equinos para un rápido escaneo a nivel genómico (Illumina, San Diego, CA; Affymetrix, Santa Clara, CA). Los estudios de asociación del genoma completo, basados en SNPs han demostrado ser excepcionalmente exitosos cuando se estudian rasgos de herencias mendelianas simples y muy útiles para la detección de QTLs en especies domesticadas. Estos arrays de genotipificación han tenido su correlato en el campo de la expresión génica, y han permitido describir los perfiles de expresión génica a nivel genómico en bovinos y equinos para numerosos tejidos, estados de desarrollo, estados sanitarios, etc.
Otra de las áreas de interés de la genética de animales domésticos es la identificación genética, que representa un campo de investigación dinámico que ha evolucionado continuamente en los últimos quince años (Villegas Castagnasso et al., 2010; Posik et al., 2010). Las décadas de los 70 y 80 fueron los años de aplicación de los polimorfismos de los grupos sanguíneos, de las proteínas del suero, de los glóbulos rojos y de los serotipos del Complejo Principal de Histocompatibilidad (MHC). En los últimos quince años, los marcadores utilizados por excelencia en la identificación genética y en la determinación de parentesco en bovinos y equinos, fueron los microsatélites (Short Tandem Repeats, STR) y en menor medida los AFLP (Amplified Fragment Length Polymorphism) (Villegas Castagnasso et al., 2010). En 1995, el registro oficial en bovinos y equinos aún se llevaba a cabo a través de grupos sanguíneos y polimorfismos bioquímicos. Recién en el año 2002, durante el desarrollo de los Workshops sobre identificación genética en bovinos y equinos (Gottingen, 2002, Alemania) organizados por la Sociedad International de Genética Animal (International Society for Animal Genetics, ISAG), se llegó a un consenso para reemplazar las tipificaciones tradicionales (grupos sanguíneos y polimorfismos bioquímicos) por sistemas basados en polimorfismos de ADN (microsatélites). Sin embargo, debido a los recientes avances en equipos automáticos de alta perfomance y en bioinformática, la era de los microsatélites pareciera estar llegando a su fin. En los próximos años, se espera que el campo de la identificación genética sea dominado por los marcadores basados en SNPs (Heaton et al., 2002). Dentro de la identificación genética animal, ha surgido recientemente una rama denominada Genética Forense Animal (Posik et al., 2007), que ha comenzado a consolidarse a partir de los Workshop organizado por la ISAG en el año 2006 (Porto Seguro, Brasil).
Si la aparición de la PCR a principios de los 90 revolucionó la mayoría de los campos de la biología, actualmente estamos ante cambios de una magnitud hasta hace poco inimaginable. Esto ha sido motorizado por el desarrollo de nuevas tecnologías de análisis de genómica estructural y funcional, desarrollos bioinformáticos, mayor acceso a la información a través de internet, etc.
EL ESTADO DEL ARTE DE LA GENÉTICA EN CRIOLLOS
A principios de los años 90, pocas razas criollas de animales domésticos habían sido caracterizadas mediantes marcadores genéticos. Entre los primeros estudios pueden mencionarse los trabajos de caracterización genética en bovinos y equinos criollos argentinos, basados en polimorfismos bioquímicos y grupos sanguíneos (Quinteros et al., 1973, 1980; Puig, 1984; Poli, 1986; Poli y Antonini, 1991; Peral García et al., 1996, 1997). Es recién a mediados de esta década, que siguiendo los avances tecnológicos, se publican los primeros trabajos de caracterización basados en marcadores de ADN.
A pesar del desarrollo explosivo que ha experimentado la genética molecular de animales domésticos, los trabajos realizados en razas criollas se han centrado principalmente en estudios filogeográficos. El objetivo de los mismos ha sido la evaluación de la diversidad genética para su conservación (Ej., Magee et al., 2002; Miretti et al., 2002; Mirol et al., 2003; Carvajal-Carmona et al., 2003; Lirón et al., 2006a, Giovambattista et al., 2010; Ginja et al., 2010), mediante el uso de marcadores genéticos del ADN mitocondrial, del cromosoma Y, microsatélites y polimorfismos de genes candidatos para caracteres específicos. Debido a que las razas criollas presentarían una mayor resistencia a enfermedades infecciosas y una gran adaptabilidad a condiciones desfavorables, varios trabajos han estudiado la variabilidad genética de genes del MHC (Giovambattista et al., 1999, 2001; Peral García et al., 1999; Díaz et al., 2001, 2005, 2008; Miretti et al., 2001; Ripoli et al., 2002, 2003; Villegas-Castagnasso et al., 2003; Martínez et al., 2004; Hernández Herrera et al., 2009) o genes relacionados con la respuesta inmune (Vázquez-Flores et al. 2006). La relevancia de esta clase de estudios radica en que: i) la diversidad de las especies domésticas es considerada como un importante componente de la biodiversidad global (Hall & Ruane 1993; Hall & Bradley 1995; Ajmone-Marsan et al., 2010; Boettcher et al., 2010; Groeneveld et al., 2010); ii) el pool génico de las diferentes razas criollas puede haber conservado genes o combinaciones de genes que se han perdido en las razas altamente seleccionadas, y que podrían ser de utilidad ante futuros requerimientos; iii) las poblaciones criollas son consideradas como un valioso patrimonio cultural, histórico y genético de los diferentes países americanos; iv) la mayoría de las razas criollas han sufrido en el último siglo una drástica reducción en el tamaño poblacional y/o un detrimento en su grado de pureza, razón por la cual muchas de ellas están en riesgo de extinción (Delgado Bermejo et al., 2010).
Si bien muchas de las razas criollas han sido caracterizadas genéticamente, son escasos los trabajos de asociación entre marcadores genéticos y caracteres de producción animal o rendimiento físico (Ej. Ripoli et al., 2003). Por otra parte, el estudio de los criollos aún no ha entrado en la era de los Omics (proteómica, genómica, etc.), provocando un atraso relativo del conocimiento de estas razas respecto al de razas altamente seleccionadas o más difundidas. Las causas varían según la región y la especie, pero entre ellas podemos mencionar: i) el reducido tamaño poblacional, representado por pequeños grupos pertenecientes a productores de zonas marginales; ii) la escasez de datos productivos sistematizados y ausencia de sistemas de evaluación genética a nivel racial; iii) la falta de fondos suficientes de los sistemas científicos y tecnológicos de los países latinoamericanos, para realizar estudios a nivel genómico; iv) o que paradójicamente estas razas muchas veces no son incluidas en la lista de áreas prioritarias. En este sentido, muchos de los trabajos de caracterización han sido realizados total o parcialmente en el exterior, especialmente en la Comunidad Europea, con la colaboración de grupos locales. Un claro ejemplo son los estudios apoyados por la Red Iberoamericana sobre la Conservación de la Biodiversidad de Animales Domésticos Locales para el Desarrollo Rural Sostenible (RED CONBIAND, CYTED, http://www.uco.es/conbiand/Bienvenida.html).
A continuación, se detallan los principales resultados obtenidos en bovinos y equinos criollos latinoamericanos, aunque también existen estudios en otras especies criollas latinoamericanas. En la Tabla I se detallan el número de trabajos publicados en las dos especies utilizando los diferentes tipos de marcadores genéticos. Esta revisión no pretende recopilar la totalidad de los trabajos publicados, sino marcar la tendencia sobre el perfil de los estudios realizados durante los últimos 15 años, basándose en las principales bases bibliográficas, tales como el PubMed (http://www.ncbi.nlm.nih.gov/PubMed), Google Académico (http://scholar.google.com.ar/), Centro Argentino de Información Científica y Tenológica (CAICYT, www.caicyt.gov.ar/), actas de las reuniones de la RED CONBIAND, entre otros.
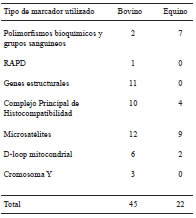
Tabla I. Información relevada de las principales bases bibliográficas, expresada en número de trabajos.
EL ESTADO DEL ARTE DEL CONOCIMIENTO DE LOS BOVINOS CRIOLLOS
La historia oficial define a los bovinos criollos como los descendientes de los bovinos ibéricos introducidos por los conquistadores españoles y portugueses durante los primeros 50 años de colonización, por lo que el grupo fundador no habría superado los 300 - 1000 animales. Sin embargo, como se verá más adelante, los resultados obtenidos evidencian una diversidad y complejidad más compatible con un escenario de múltiples introducciones desde diferentes fuentes de origen. Como consecuencia de su origen y de su historia reciente, las diferentes clases de marcadores genéticos (mitocondriales, holándricos y autosómicos) muestran sólo una visión parcial, siendo necesaria una interpretación conjunta de la información aportada por estos tres tipos de marcadores para obtener una imagen fidedigna de su pasado y presente.
En los últimos años, cinco trabajos describieron la región control del ADNmt en razas criollas americanas de Argentina, Bolivia, Brasil, Colombia y tres islas del Caribe (Magee et al., 2002; Miretti et al., 2002; Mirol et al., 2003; Carvajal-Carmona et al., 2003; Lirón et al., 2006), reportando la existencia haplotipos mitocondriales pertenecientes a haplogrupos africanos y europeos, sin encontrar haplotipos de los haplogrupos cebuinos I1 e I2. El análisis conjunto de todas estas secuencias reportadas mostró que el haplogroupo europeo T3, mayoritario en Europa Occidental, fue también el más común en las razas americanas (64%), seguido por el haplogrupo subsahariano T1 (32%) y el haplogrupo T2 (4%), frecuentemente observado en el Cercano Oriente y raramente en Europa. Con el fin de clarificar y reinterpretar el patrón filogeográfíco en continente Americano, Lirón et al. (2006, 2008) agruparon las secuencias del ADNmt disponibles teniendo en cuenta un criterio geopolítico de la época colonial: los Virreinatos españoles de Nueva Granada (Colombia, Ecuador) y Río de la Plata (Argentina, Bolivia, Uruguay), Nueva España (México, EEUU), Cuba, la colonia portuguesa del Brasil, y las Antillas Menores, colonizadas por otras Coronas europeas (Francia, Reino Unido). La distribución de los haplogroupos resultó ser apreciablemente variable tanto entre como dentro de las diferentes regiones geográficas. Estos resultados fueron confirmados recientemente por Ginja et al. (2010).
El linaje africano T1 puede dividirse en dos clusters: el sub-haplogroupo monofilético T1a y el sub-haplogroupo parafilético T1* (Troy et al., 2001, Miretti et al., 2002, Lirón et al., 2006). Interesantemente, los haplogrupos T1 presentan una distribución geográfica diferente: mientras el sub-haplogrupo T1* se encontró en todas las ex-colonias españolas estudiadas hasta el presente, el haplogrupo T1a estuvo inicialmente restringido a Brasil y las colonias no-españolas del Caribe. Recientemente, este sub-haplogrupo fue detectado en poblaciones de México y Paraguay (Ginja et al., 2010). Varias cuestiones emergen sobre el origen y la distribución de los matrilinajes africanos en los bovinos criollos americanos. En primer lugar respecto del tiempo introducción: ingreso temprano -previo al siglo XIX- vs. Introducción reciente. Si se tiene en cuenta la amplia distribución geográfica del hapogrupo T1 (desde la Patagonia hasta el sur de Estados Unidos) y la considerable frecuencia, que en algunas poblaciones llegan al 50%, el escenario más plausible sería el de una introducción temprana de estos linajes. En segundo lugar respecto del origen geográfico de los linajes africanos, como se describió previamente, es ampliamente asumido que los bovinos llevados por los colonizadores españoles y portugueses provenían de la península Ibérica. Por lo tanto, la presencia de linajes africanos en las razas criollas podría estar reflejando la naturaleza híbrida de las razas ibéricas (Cymbron et al., 1999; Miretti et al., 2004; Ginja 2010). Sin embargo, es difícil reconciliar el actual patrón de genes africanos presente en la península Ibérica con el observado en las razas criollas americanas. Así, por ejemplo, mientras que en las razas portuguesas sólo se detectó el haplogrupo T1* (Cymbron et al., 1999, Ginja et al., 2010), en las razas criollas del Brasil se encontró el linaje T1a en muy alta frecuencia (Miretti et al., 2002).
En España, el subhaplogrupo T1a ha sido reportado en unos pocos animales de las razas Retinta, la más común del sur de España, y de Lidia (Miretti et al., 2005; Cortes et al. 2008). Esto contrasta con lo observado en la mayoría de las poblaciones de bovinos criollos de ex-colonias españolas, donde sólo ha sido detectado el linaje T1*. Alternativamente a la introducción de los haplotipos africanos desde Iberia, se podría considerar la introgresión al menos en parte desde algún lugar del continente Africano, avalado por los datos históricos sobre la ruta del tráfico de esclavos (Rouse 1973; Maillard et al., 1993; Felius, 1995). Esta hipótesis podría explicar las discordancias observadas en los patrones mitocondriales entre América y la Península Ibérica. Aunque el subhaplogrupo T1a no ha sido detectado en África, su aparente ausencia se debería a la limitada distribución geográfica de las muestras analizadas hasta el presente en dicho continente (Bradley et al., 1996; Troy et al., 2001; Beja-Pereira et al., 2006). Sin embargo, cabe destacar que en varias razas del norte de África y en la raza portuguesa Alentejana se han detectado haplotipos africanos que presentaban dos o tres de los polimorfismos característicos del subhaplogrupo T1a. Esto podría estar sugiriendo el origen geográfico del mencionado subhaplogrupo (Beja-Pereira et al., 2006; Ginja et al., 2010). Por otra parte, los datos históricos y genéticos avalan que los esclavos africanos provenían originalmente del oeste, centro-oeste y sudeste de África (Klein, 1999; Thomas, 1998). Mientras que el ADNmt de las razas del oeste africano ha sido estudiado, no existen informes disponibles sobre las restantes potenciales regiones de origen del haplogrupo T1a. Además, T1a es un clado altamente divergente que presenta una forma de estrella compuesto por unos pocos haplotipos, la mayoría de los cuales sólo han sido detectados en un único individuo y se desvían del haplotipo nodal por un solo cambio mutacional. Esta reducida variabilidad sugiere un origen geográfico restringido para el linaje T1a presente en América, y una rápida y reciente diversificación de este haplogroupo.
En resumen, la presencia de los linajes ADNmt africanos y europeos en los bovinos Criollos es indiscutible, pero el análisis realizado por Lirón et al. (2006) sugeriría la existencia de dos fuentes independientes para el componente africano en América. Mientras el linaje T1* podría haber arribado a través de las razas Ibéricas, el linaje mitocondrial T1a provendría de algún lugar desconocido, aún no muestreado de África y podría haber sido introducido junto al tráfico de esclavos desde el continente africano.
Si consideramos que las poblaciones de bovinos criollos derivarían de un reducido grupo fundador y que en los últimos 100 años han sufrido una drástica reducción y subdivisión poblacional, sería esperable que los bovinos criollos presenten bajos niveles relativos de diversidad genética. Sin embargo, existe una considerable diversidad fenotípica, y los resultados obtenidos por varios autores (ej. Zamorano et al., 1998; Postiglioni et al., 2002; Lirón et al., 2002, 2006; Steigleder et al., 2004; Armstrong et al., 2006) mostraron niveles relativamente altos de variación genética, estimados a través de la diversidad alélica y la heterocigosidad, y significativos niveles de subdivisión poblacional (Giovambattista et al., 2001). Aunque las razas criollas americanas han sido relativamente menos estudiadas que las razas bovinas de otras regiones, los estudios sobre estas razas nativas no se han limitado al análisis de los marcadores antes mencionados (microsatélites, ADNmt y cromosoma Y). Entre los loci que se han empleado para caracterizar algunas de estas razas pueden mencionarse los grupos sanguíneos, las proteínas lácteas, RAPD, hormonas y enzimas y genes del complejo principal de histocompatibilidad (Giovambattista et al., 1996, Golijow et al., 1996; Zamorano et al., 1998; Rincón et al., 2000; 2001; Bedoya et al., 2001; Postiglioni et al., 2002; Lirón et al., 2002, 2006; Fernando Moreno et al., 2003; Martínez et al., 2003; Ripoli et al., 2003, 2004, 2006; Steigleder et al., 2004; Armstrong et al., 2006; Quiroz-Valiente et al., 2006; Vázquez-Flores et al. 2006; Egito et al., 2007; Villalobos Cortés et al., 2009). Entre las principales conclusiones observadas en estos trabajos se pueden mencionar: valores relativamente altos de diversidad genética, altos niveles de subdivisión poblacional, frecuencias intermedias entre las taurinas y cebuinas y la presencia de variantes características de las razas cebuinas. Dichas conclusiones concuerdan con lo observado con los marcadores genéticos antes discutidos. Estos resultados podrían ser explicados por los bajos niveles de selección artificial sufrida por estas poblaciones. Por otra parte, estos resultados podrían ser consecuencia de la contribución de genes europeos e índicos, provenientes de múltiples orígenes geográficos, en el grupo fundador y la reciente introgresión de genes cebuinos. Varios trabajos han estudiado las relaciones genéticas de los bovinos criollos con otras razas de bovinos domésticos taurinos y cebuinos.
La profunda diferencia entre los genomas de estos dos grupos se ve reflejada en la presencia de ciertos alelos con altas frecuencias en los cebuinos y ausentes o presentes a bajas frecuencias en los taurinos. Interesantemente, los alelos diagnósticos de cebú también se detectaron, aunque a bajas frecuencias, en casi todas las razas criollas estudiadas. Por otra parte, las relaciones estudiadas mediante árboles filogenéticos y componentes principales muestran la divergencia (aunque con bajos niveles de boostrap) entre las razas europeas e ibéricas-americanas, y ubicando a varias razas criollas entre los grupos cebuinos y taurinos (Lirón et al., 2006; Ripoli et al. 2010). Por otro lado, se han estudiado los patrones de mezcla génica presentes en las poblaciones de criollos, mediante marcadores genéticos uni y bi-parentales. Desde la ventana de los matrilinajes (ADNmt), todos los animales estudiados presentaron únicamente haplotipos taurinos (Magee et al. 2002; Carvajal-Carmona et al. 2003; Miretti et al. 2002, 2004; Mirol et al. 2003, Lirón et al. 2006, 2008, Ginja et al. 2010).
Por el contrario, el análisis del cromosoma Y (morfología cromosómica, microsatélites y SNPs) de las diferentes poblaciones criollas americanas mostró una considerable incidencia del cromosoma Y cebuino (Giovambattista et al. 2000, De Luca et al., 2002; Ginja et al. 2010). Por otra parte, al igual que lo observado en ADNmt, los haplotipos taurinos se dividían en haplogrupos europeos y africanos (Gija, et al. 2010). Como era de esperar, la composición genética a nivel de marcadores autosómicos mostró un escenario intermedio, que estaría representando más exactamente el grado de introgresión de genes índicos en las poblaciones bovinas criollas. Patrones similares de flujo génico han sido previamente reportados en razas de África y de Asia (MacHugh et al. 1997, Freeman et al. 2004, Kikkawa et al 2003), que representarían el modelo general de mezcla génica (introgresión mediada por machos). En este sentido, se observó un claro patrón de introgresión de genes índicos en Sudamérica (Giovambattista et al., 2000), con las frecuencias de los alelos cebuinos decreciendo de este a oeste y de norte a sur, compatible con los datos históricos y ambientales.
EL ESTADO DEL ARTE DEL CONOCIMIENTO DE LOS EQUINOS CRIOLLOS
Los caballos Criollos Americanos constituyen las poblaciones remanentes de los caballos introducidos al Nuevo Mundo por los conquistadores europeos, por lo que generalmente se ha asumido que la fuente original de esos caballos ha sido la península Ibérica. Es por esta razón que sería de esperar que los caballos criollos estuvieran relacionados genéticamente con las razas andaluzas, españolas de origen celta, berberiscas y árabes. En coincidencia con esta hipótesis, la raza nativa americana de origen español Mustang presenta una alta incidencia del haplogrupo D1 (Jansen et al., 2004). Con el fin de establecer la relaciones entre el caballo Criollo Argentino y el Paso Peruano con las razas de caballo españolas, Mirol et al. (2002) caracterizaron el D-loop mitocondrial en 104 animales correspondientes a nueve razas nativas de Sudamérica y España mediante SSCP y secuenciación. Los resultados obtenidos mostraron que el haplotipo más común en los caballos criollos estudiados coincidió con el más abundante en los caballos andaluces. Por otra parte, a pesar que se observaban niveles significativos de subdivisión entre las razas, los análisis filogenéticos no evidenciaron grupos monofiléticos, no existiendo en la topología del árbol una clara relación entre las razas Criollas y las otras razas analizadas. Aunque, estos resultados podrían ser interpretados como una mezcla de ancestros en la formación de los caballos criollos, es probable que estén indicando la retención de linajes maternos muy antiguos en las razas estudiadas.
Los estudios de caracterización genética de razas de equinos criollos basados en marcadores genéticos autosómicos, tales como polimorfismos bioquímicos, genes del MHC equino y microsatélites, mostraron un elevado nivel de diversidad genética y cierto nivel de subestructuración poblacional. Además, las razas criollas evidenciaron una mayor semejanza genética entre ellas, y en menor medida con razas ibéricas, que con razas de otras regiones geográficas (Peral-García et al., 1996; Peral García et al., 1999; Cothran et al., 1998; Kelly et al., 1998, 2002; Díaz et al., 2001, 2005, 2008; Paredes et al., 2002; Villegas-Castagnasso et al., 2003; Vinocur et al., 2003; Mujica et al., 2005; Jiménez Robayo et al., 2007; Tavares Pires de Souza Sereno et al., 2008; De Assis et al., 2009).
Los estudios basados en el análisis del ADNmt y marcadores autosómicos evidenciaron un alto nivel de diversidad de matrilinajes en los caballos criollos. Por el contrario, este elevado nivel de polimorfismo no se corresponde con la diversidad de patrilinajes observado en los loci específicos del cromosoma Y en las poblaciones de esta especie. El extensivo estudio realizado por Lindgren et al. (2004) en 15 razas equinas de Europa y Asia reveló que todos los animales estudiados tenían el mismo haplotipo, no pudiéndose identificar sitios polimórficos en las secuencias analizadas (14,3 kb). Por lo tanto, debido a la ausencia de polimorfismos reportados del cromosoma, no existen trabajos sobre caracterización genéticas de razas de equinos criollos basados en marcadores del cromosoma Y.
CONCLUSIONES
Durante los últimos 15 años, mucho se ha avanzado en la genética molecular de bovinos y equinos criollos latinoamericanos. Hoy en día, existen grupos consolidados y referentes en toda Latinoamérica dedicados fundamentalmente al estudio de marcadores genéticos en estas especies. El número de trabajos publicados, como la masa crítica de investigadores es considerablemente mayor en bovinos que en equinos. Sin embargo, gran parte de las publicaciones son de índole local o regional, lo cual dificulta su acceso masivo a través de los buscadores más frecuentemente utilizados (mencionados anteriormente en este artículo). Por otra parte, también podemos citar trabajos de genética molecular realizados por consorcios internacionales que incluyen las razas criollas latinoamericanas dentro de sus paneles. Las principales conclusiones obtenidas a partir de estos quince años de trabajo son:
1. Las razas de bovinos y equinos criollos presentan altos niveles de diversidad y estructuración poblacional, que en muchos casos es superior a las reportadas para razas altamente seleccionadas.
2. El pool génico de los bovinos criollos americanos tendría al menos cuatro orígenes: dos africanos (introducidos directamente desde Iberia o siguiendo la ruta de comercio de esclavos), uno europeo mediterráneo (procedente del Cercano Oriente) y otro a partir del B. primigeneous del Mediterráneo Europeo. Un porcentaje de su pool génico está constituido por genes índicos incorporados por un proceso de introgresión mediada por machos. 3. En equinos, los análisis filogenéticos no evidenciaron grupos monofiléticos, por lo que no existe en la topología del árbol una clara relación entre las razas criollas y otras razas analizadas. Si bien estos resultados podrían ser interpretados como una mezcla de ancestros en la formación de los caballos criollos, es probable que estén indicando la retención de linajes maternos muy antiguos en las razas estudiadas.
Los altos niveles de diversidad génica y la presencia de un elevado número de alelos privativos de las razas criollas, apoyan la hipótesis que estas poblaciones constituyen importantes e irremplazables reservorios de diversidad genética para las especies bovina y equina. Además, el conocimiento de las razas autóctonas es clave para la planificación de estrategias de conservación sustentables a lo largo del tiempo.
BIBLIOGRAFÍA
1. Ajmone-Marsan, P., (2010) The GLOBALDIV Consortium. A global view of livestock biodiversity and conservation - GLOBALDIV. Anim. Genet. 41, 1-5. [ Links ]
2. Armstrong E., Postiglioni, A., Martínez, A., Rincón, G., Vega-Pla, J.L. (2006) Microsatellite analysis of a sample of Uruguayan Creole bulls (Bos taurus) Genet. Mol. Biol. 29, 267-272. [ Links ]
3. Bedoya, G, Carvajal, L.G., Bermúdez, N. R., Moreno, F. L., Márquez, M. E., Davies, S., Derr, J., Ossa Londoño, J., Ruiz A. (2001) Estructura molecular y poblacional del ganado criollo Colombiano (GCC). Revista Colombiana de Ciencias Pecuarias 14(2), 107-118. [ Links ]
4. Beja-Pereira, A., Caramelli, D., Lalueza-Fox, C., Vernesi, C., Ferrand, N., Casoli, A., Goyache, F., Royo, L. J., Conti, S., Lari, M., Martini, A., Ouragh, L., Magid, A., Atash, A., Zsolnai, A., Boscato, P., Triantaphylidis, C., Ploumi, K., Sineo, L., Mallegni, F., Taberlet, P., Erhardt, G., Sampietro, L., Bertranpetit, J., Barbujani, G., Luikart, G., Bertorelle, G. (2006) The origin of European cattle: evidence from modern and ancient DNA. Proc. Natl. Acad. Sci. U.S.A. 103, 8113-8 . [ Links ]
5. Boettcher, P.J., Tixier-Boichard, M., Toro, M.A., Simianer, H., Eding, H., Gandini, G., Joost, S., Garcia, D., Colli, L., Ajmone-Marsan, P. (2010) Objectives, criteria and methods for using molecular genetic data in priority setting for conservation of animal genetic resources. Anim. Genet. 41, 64-77. [ Links ]
6. Bovine Genome Sequencing and Analysis Consortium, Elsik, C.G., Tellam, R.L., Worley, K.C., Gibbs, R.A., Muzny, D.M., Weinstock, G.M., Adelson, D.L., Eichler, E.E., et al. (2009)The genome sequence of taurine cattle: a window to ruminant biology and evolution. Science 324, 522-8. [ Links ]
7. Bovine HapMap Consortium, Gibbs, R.A., Taylor, J.F., Van Tassell, C.P., Barendse, W., Eversole, K.A., Gill, C.A., Green, R.D., Hamernik, D.L. et al. (2009) Genome-wide survey of SNP variation uncovers the genetic structure of cattle breeds. Science 324, 528-32. [ Links ]
8. Brooks, S.A., Gabreski, N., Miller, D., Brisbin, A., Brown, H.E., Streeter, C., Mezey, J., Cook, D., Antczak, D.F. (2010) Whole-Genome SNP Association in the Horse: Identification of a Deletion in Myosin Va Responsible for Lavender Foal Syndrome. PLoS Genet. 6, e1000909. [ Links ]
9. Casas E. (2010) Capítulo 13: Genes involucrados en caracteres de producción de carne, en Genética de Animales Domésticos (Giovambattista, G., Peral-García, P.) Editorial Inte-Médica, Ciudad Autónoma de Buenos Aires - Argentina, 237-241. [ Links ]
10. Casas, E., Bennett, G.L., Bottema, C.D., Crawford, A., Kalm, E., Kappes, S.M., Kister, A., Lewin, H.A., Lien, S., Morris, C.A., Olsaker, I., Pitchford, W.S., Schmutz, S.M., Thomsen, H., Xu, N. (2001) Comprehensive linkage map of bovine chromosome 11. Anim. Genet. 32, 92 - 94. [ Links ]
11. Chowdhary, B.P., Raudsepp, T., Kata, S.R., Goh, G., Millon, L.V., Allan, V., Piumi, F., Guérin, G., Swinburne, J., Binns, M., Lear, T.L., Mickelson, J., Murray J.,, Antczak, D.F., Womack, J.E., Skow, L.C. (2003) The First-Generation Whole-Genome Radiation Hybrid Map in the Horse Identifies Conserved Segments in Human and Mouse Genomes. Genome Res. 13, 742-751. [ Links ]
12. Cothran, E.G., Santos, S.A., Mazza, M.C.M., Lear, T.L., Sereno, J.R.B. (1998) Genetics of the Pantaneiro horse of the Pantanal region of Brazil. Gen. Mol. Biol. 21, 343-349. [ Links ]
13. De Assis, J.B., DeLaat, D.M., Peixoto, M.G.C.D., Bergmann, J.A.G., Fonseca, C.G., Carvalho, M.R.S. (2009) Genetic diversity and population structure in Brazilian Mangalarga Marchador horses. Gen. Mol. Res. 8, 1519-1524. [ Links ]
14. De Luca J.C., Zufriategui, L., Picco, S.J., Ripoli, M.V., Giovambattista, G., Rojas, F.V., Dulout, F.N. (2002) Incidence of 1/29 translocation in Bolivian Creole and Brahman Yacumeño cattle. Theriogenology 58, 1273-1281. [ Links ]
15. Delgado Bermejo, J.V., Martínez Martínez, A., Camacho Vallejo, M.E., Vega Pla J.L. (2010) Capítulo 6: Conservación de Razas de especies Domésticas, en Genética de Animales Domésticos (Giovambattista, G., Peral-García, P.) Editorial Inte-Médica, Ciudad Autónoma de Buenos Aires - Argentina, 105-121. [ Links ]
16. Díaz, S., Echeverría, G., It, V., Posik, D.M., Rogberg-Muñoz, A., Peral-García, P., Vega-Pla, J.L., Giovambattista, G. (2008) Development of an ELA-DRA gene typing method based on pyrosequencing technology. Tissue Antigens 72, 464-8. [ Links ]
17. Díaz, S., Giovambattista, G., Dulout, F.N., Peral García, P. (2001) Genetic variation of the second exon of ELA-DRB genes in Argentine Creole horses. Anim. Genet. 32, 257-263. [ Links ]
18. Díaz, S., Giovambattista, G., Peral-García, P. (2005) Structure and polymorphism of the upstream regulatory region of the Major Histocompatibility Complex DRB genes in domestic horses. Int. J. Immunogenet. 32, 91-98. [ Links ]
19. Egito, A.A., Paiva, S.R., Albuquerque, M.S.M., Mariante, A.S., Almeida, L.D., Castro, S.R., Grattapaglia, D. (2007) Microsatellite based genetic diversity and relationships among ten Creole and commercial cattle breeds raised in Brazil. BMC Genet. 8, 83. [ Links ]
20. Felius M. (1995) Cattle breeds: an encyclopaedia. Editorial:C Misset bv. the Netherlands [ Links ]
21. Freeman, A.R., Meghen, C.M., MacHugh, D.E., Loftus, R.T., Achukwi, M.D., Bado, A., Sauveroche, B., Bradley, D.G. (2004) Admixture and diversity in West African cattle populations. Mol. Ecol. 13, 3477-3487. [ Links ]
22. Fries, E., Eggen, A., Womack, J. (1993) The bovine genome map. Mamm. Genome 4, 405-428. [ Links ]
23. Ginja, C., Penedo, M.C.T., Melucci, L., Quiroz, J., Martínez López, O.R., Revidatti, M.A., Martínez-Martínez, A., Delgado, J.V., Gama, L.T. (2009) Origins and genetic diversity of New World Creole cattle: inferences from mitochondrial and Y chromosome polymorphisms. Anim. Genet. 41, 128-141. [ Links ]
24. Giovambattista, G., Golijow, C.D., Dulout, F.N., Lojo, M.M. (1996) Gene frequencies of DRB3.2 locus of Argentine Creole cattle. Anim. Genet. 27, 55-56. [ Links ]
25. Giovambattista, G., Ripoli, M.V., De Luca, J.C., Mirol, P., Lirón, J.P., Dulout, F.N. (2000) Geographic distribution and frequency of taurine Bos taurus and zebu B. indicus Y chromosome incidence amongst Argentine and Bolivian Creole cattle breeds. Anim. Genet. 31, 302-5. [ Links ]
26. Giovambattista, G., Ripoli, M.V., Peral García, P., Bouzat, J.L. (2001) Indigennous domestic breeds as reservoirs of genetic diversity: The Argentine Creole Cattle. Anim. Genet. 32, 240-247. [ Links ]
27. Golijow, C.D., Giovambattista, G., Poli, M., Dulout, F.N., Lojo, M.M. (1996) K-casein gene frequencies support subdivision and historical origin of Argentine Creole cattle. Rev. Bras. Genet. 19, 583-586. [ Links ]
28. Groeneveld, L.F., Lenstra, J.A., Eding, H., Toro, M.A., Scherf, B., Pilling, D., Negrini, R., Finlay, E.K., Jianlin, H., Groeneveld, E., Weigend, S., (2010) The GLOBALDIV Consortium Genetic diversity in farm animals - a review. Anim. Genet. 41, 6-31. [ Links ]
29. Guerin, G., Bailey, E., Bernoco, D., Anderson, I., Antczak, D.F., et al. (1999) Report of the International Equine Gene Mapping Workshop: male linkage map. Anim. Genet. 30, 341-354. [ Links ]
30. Hall S.J.G., Bradley D.G. (1995) Conserving livestock breed biodiversity. Trends. Ecol. Evol. 10, 267-70. [ Links ]
31. Hall S.J.G., Ruane J. (1993) Livestock breeds and their conservation: a global overview. Conserv. Biol. 7, 815-25. [ Links ]
32. Heaton, M.P., Harhay, G.P., Bennett, G.L., Stone, R.T., Grosse, W.M., Casas, E., Keele, J.W., Smith, T.P.L., Chitko-McKown, C.G., Laegreid, W.W. (2002) Selection and use of SNP markers for animal identification and paternity analysis in U.S. beef cattle. Mamm. Genome 13, 272-281. [ Links ]
33. Hernández Herrera, D.Y., Posso Terranova, A.M., Benavides, J.A., Muñoz Flórez, J.E., Giovambattista, G., Álvarez Franco, L.Á. (2009) Polimorfismos del gen BoLA-DRB3.2 en razas criollas y colombianas. Revista Colombiana de Ciencias Pecuarias 22, 469. [ Links ]
33. Jansen, T., Forster, P., Levine, M.A., Oelke, H., Hurles, M., Renfrew, C., Weber, J., Olek, K. (2002) Mitochondrial DNA and the origins of the domestic horse. Proceedings of the National Academy of Sciences of the USA. 99(16), 10905-10910. [ Links ]
34. Jiménez Robayo, L.M., Cañón Ferreras, J., Sánchez Isaza, C.A., Sandoval, F.A., Gómez Tarazona A. (2007) Análisis de la variabilidad genética en caballos criollos llaneros colombianos mediante la utilización de microsatélites. Rev Col Cienc Pec 20, 4. [ Links ]
35. Kappes, S.M., Keele, J.W., Stone, R.T., McGraw, R.A., Sonstegard, T.S., Smith, T.P.L., Lopez-Corrales, N.L., Beattie, C.W. (1997). A second generation linkage map of the bovine genome. Genome Res. 7, 235-249. [ Links ]
36. Kelly L., A. Postiglioni, D. F. de Andrés, J. L. Vega-Plá, R. Gagliardi, R. Biagetti and J. Franco. (2002) Genetic characterisation of the Uruguayan Creole horse and analysis of relationships among horse breeds. Reserch Veterinary Sciences 72, 69-73. [ Links ]
37. Kelly L., Postiglioni, A., De Andres, D. F., Gagliardi, R., Biagetti, R., Franco, J. (2002) Variabilidad genética de los caballos criollos del Uruguay Arch. med. vet. 34(1), 13-23. [ Links ]
38. Kelly, L., Postiglioni, A., Andrés, D. (2002) Variabilidad genética de los caballos Criollos del Uruguay. Arch. Med. Vet. 34, 13-23. [ Links ]
39. Kelly, L., R. Gagliardi, R. Biagetti, A. Postiglioni, D.F. De Andrés, (1998) análisis de marcadores genéticos en una muestra de caballos criollos del uruguay (analysis of genetic markers in a sample of uruguayan criollo horses). Arch. Zootec. 178, 259-266. [ Links ]
40. Kikkawa, Y., Takada, T., Sutopo, Nomura, K., Namikawa, T., Yonekawa, H., Amano, T. (2003) Phylogenies using mtDNA and SRY provide evidence for male-mediated introgression in Asian domestic cattle. Anim. Gen. 34: 96-101. [ Links ]
41. Klein, H.S. (1999) The slave trade. Cambridge University press, Cambridge,UK, 225. [ Links ]
42. Lindgren, G., Backstrom, N., Swinburne, J., Hellborg, L., Einarsson, A., Sandberg, K., Cothran, G., Vila, C., Binns, M., Ellegren, H. (2004) Limited number of patrilines in horse domestication. Nat. Genet. 36, 335-336. [ Links ]
43. Lirón J.P., Posik, D.M., MacNeil, M.D., Mirol, P.M., Rogberg-Muñoz, A., Martínez-Martínez, A., Maciel, S., Terán-Polo, C.R., Peral-García, P., Hanotte, O., Giovambattista, G. (2008) Origen histórico y afinidad filogeográfica de los bovinos criollos americanos a través del estudio del ADN mitocondrial. IX Simposio Iberoamericano sobre Conservación y Utilización de Recursos Zoogenéticos. Mar Del Plata, Buenos Aires, Argentina. 311-315. [ Links ]
44. Lirón, J.P., Bravi, C.M., Mirol, P.M., Peral-García, P., Giovambattista, G. (2006) African matrilineages in American Creole cattle: evidence of two independent continental sources. Anim. Genet. 37, 379-382. [ Links ]
45. Lirón, J.P., Peral-García, P., Giovambattista, G. (2006) Genetic characterization of Argentine and Bolivian Creole cattle breeds assessed through with microsatellites. J. Hered. 97, 331-9. [ Links ]
46. Lirón, J.P., Ripoli, M.V., De Luca, J.C., Peral-García, P., Giovambattista, G. (2002) Análisis of Genetic Diversity and Population Structure in Argentine and Bolivian Creole Cattle using Five Loci Related to Milk Production. Genet. Mol. Biol. 25, 413-419. [ Links ]
47. Liu, Y., Qin, X., Song, X.Z., Jiang, H., Shen, Y., Durbin, K.J., Lien, S., Kent, M.P., Sodeland, M. Ren, Y., Zhang, L., Sodergren, E., Havlak, P., Worley, K.C., Weinstock, G.M., Gibbs, R.A. (2009) Bos taurus genome assembly. BMC Genomics 10,180. [ Links ]
48. MacHugh, D.E., Shriver, M.D., Loftus, R.T., Cunningham, P., Bradley, D.G. (1997) Microsatellite DNA variation and the evolution, domestication and phylogeography of Taurine and Zebu cattle (Bos taurus and Bos indicus). Genetics 146, 1071-1086. [ Links ]
49. Magee, D.A., Meghen, C., Harrison, S., Troy, C.S., Cymbron, T., Gaillard, C., Morrow, A., Maillard, J.C., Bradley, D.G. (2002) A partial African ancestry for the Creole cattle populations of the Caribbean. J.Hered. 93, 429-432. [ Links ]
50. Maillard, J.C., Kemp, S.J., Naves, M., Palin, C., Demangel, C., Accipe, A., Maillard, N., Bensaid, A. (1993) An attempt to correlate cattle breed origins and diseases associated with or transmitted by the tick Amblyomma variegatum in the French West Indies. Rev. Elev. Méd. Vét. Pays. Trop. 46, 283-290. [ Links ]
51. Martínez, R.D., Giovambattista, G., Ripoli, M.V., De Luca, J.C., Dulout, F.N. (2003) Patagonian Argentine Creole cattle polymorphism: comparison with North-West populations of this breed. Res. Vet. Sci. 74, 287-290. [ Links ]
52. Medrano, J.F., Rincón, G. (2010) Capítulo 12: Genes involucrados en la determinación de la composición y rendimiento de la leche, en Genética de Animales Domésticos (Giovambattista, G., Peral-García, P.) Editorial Inte-Médica, Ciudad Autónoma de Buenos Aires - Argentina, 209-225. [ Links ]
53. Meirelles, F.V., Rosa, A.J.M., Lobo, R.B., Raysildo, B., Garcia, J.M., Smith, L.C., Duarte, F.A.M. (1999) Is the American Zebu really Bos indicus? Genet. Mol. Biol. 22, 543-546. [ Links ]
54. Milenkovic, D., Oustry-Vaiman, A., Lear, L.T., Billault, A., Mariat, D., Piumi, F., Schibler, L., Cribiu, E., Guerin, G. (2002) Cytogenetic localization of 136 genes in the horse: Comparative mapping with the human genome. Mamm. Genome 13, 524-534. [ Links ]
55. Miretti, M.M., Dunner, S., Naves, M., Contel, E.P., Ferro, J.A. (2004) Predominant African-Derived mtDNA in Caribbean and Brazilian Creole Cattle is also Found in Spanish Cattle (Bos taurus). J. Hered. 95, 450-453. [ Links ]
56. Miretti, M.M., Ferro, J.A., Lara, M.A., Contel, E.P. (2001) Restriction fragment length polymorphism (RFLP) in exon 2 of the BoLA-DRB3 gene in South American cattle. Biochem. Genet. 39, 311-24. [ Links ]
57. Miretti, M.M., Pereira Jr., H.A., Poli, M.A., Contel, E.P., Ferro, J.A. (2002) African-Derived Mitochondria in South American Native Cattle Breeds (Bos taurus): Evidence of a New Taurine Mitochondrial Lineage. J. Hered. 93, 323-330. [ Links ]
58. Mirol, P.M., Giovambattista, G., Lirón, J.P., Dulout, F.N. (2003) African and European mitochondrial haplotypes in South American Creole cattle. Hered. 91, 248-254. [ Links ]
59. Mirol, P.M., Peral-García, P., Vega-Pla, J.L., Dulout, F.N. (2002) Phylogenetic relationships of Argentinean Creole horses and other South American and Spanish breeds inferred from mitochondrial DNA sequences. Anim. Genet. 33, 356-363. [ Links ]
60. Moreno, F., Bedoya, G., Der, J.N., Carvajal, L.G., Bermúdez, N., Zuluaga, F.N., Ossa, J., Verdugo, J., Estrada, L., Barrera, J., Scott, D. (2001) Diversidad genética y relaciones filogenéticas del ganado criollo colombiano. Revista Corpoica. 3, 17-23 [ Links ]
61. Mujica F., Obreque, V., Hinrichsen, P., Cothran, G. (2005) Recuperación, conservación y caracterización del caballo chilote. Agro Sur 33(1), 58-67. [ Links ]
62. Paredes, M., Norambuena, M.C., Molina, B. (2009) Diversidad genética de 12 LOCI microsatelitales utilizados en pruebas de paternidad equina en Chile. Arch. Zootec. 58, 111-116. [ Links ]
63. Penedo, M.C., Millon, L.V., Bernoco, D., Bailey, E., Binns, M., et al. (2005) International Equine Gene Mapping Workshop Report: a comprehensive linkage map constructed with data from new markers and by merging four mapping resources. Cytogenet. Genome Res. 111, 5-15. [ Links ]
64. Penedo, M.C., Millon, L.V., Bernoco, D., Bailey, E., Binns, M., et al. (2005) International Equine Gene Mapping Workshop Report: a comprehensive linkage map constructed with data from new markers and by merging four mapping resources. Cytogenet. Genome Res. 111, 5-15. [ Links ]
65. Peral-García, P., Díaz, S., Kienast, M.E., Villegas-Castagnasso, E.E., Scotti, M.C., Dulout, F.N. (1997) Relaciones genéticas entre seis razas de equinos americanos y la Pura Raza Criolla. Analecta Veterinaria 3, 11-14. [ Links ]
66. Peral-García, P., Díaz, S., Villegas Castagnasso, E., Kienast, M., De Luca, J., Giovambattista, G. (2007) Capitulo XV: Genética y Perfomance, en Fisiología del Ejercicio en Equinos. (Boffi F.M.) Editorial Inte-médica, Buenos Aires - Argentina. 278-298. [ Links ]
67. Peral-García, P., Golijow, C.D., Giovambattista, G., Lojo, M.M., Dulout, F.N. (1999) Analysis of ELA-DRB exón 2 polymorphism by PCR-RFLP. J.Equine Sci. 10, 13-16. [ Links ]
68. Peral-García, P., Kienast, M.E., Villegas-Castagnasso, E.E., Díaz, S. Dulout, F.N. (1996) Estudio de relaciones genéticas entre razas equinas mediante el análisis multivariado. Agrosur. 24,39-47. [ Links ]
69. Perrocheau, M., Boutreux, V., Chadi, S., Mata, X., Decaunes, P., Raudsepp, T., Durkin, K., Incarnato, D., Iannuzzi, L., Lear, T. L., Hirota, K., Hasegawa, T., Zhu, B., de Jong, P., Cribiu, E.P., Chowdhary, B.P., Guerin, G. (2006) Construction of a medium-density horse gene map. Anim. Gen. 37, 145-155. [ Links ]
70. Poli, M. A. (1986) Asociación entre factores sanguineos, transferrinas, genes Ps, Bs y aspectos productivos en bovinos criollos. RIA, XXI, N 2,67-76. [ Links ]
71. Poli, M.A. (1994) Marcadores bioquímicos y moleculares en animales domésticos. Reseña de los estudios en Argentina. Mendeliana XI, 60-63. [ Links ]
72. Poli, M.A., Antonini, A.G. (1991) Genetic structure of milk proteins in Argentinian Holstein and Argentinian Creole cattle. Hereditas 115, 177-182. [ Links ]
73. Posik, D.M., Giovambattista, G. Capítulo 3: Identificación especie específica de compuestos biológicos, en Genética de Animales Domésticos (Giovambattista, G., Peral-García, P.) Editorial Inte-Médica, Ciudad Autónoma de Buenos Aires - Argentina, 29-39. [ Links ]
74. Posik, D.M., Ripoli, M.V., Peral García, P., Giovambattista G. (2007) El Veredicto de la Genética. La identificación genética de especies y su uso en control de alimentos, conservación de especies y detección de fraudes. Ciencia Hoy 17, 51-57. [ Links ]
75. Postiglioni, A., Rincón, G., Kelly, L., Llambí, S., Fernandez, G., D'Angelo, M., Gagliardi, G., Trujillo, J., de Bethencourt, M., Guevara, K., Castellano, A. (2002) Biodiversidad genética en bovinos Criollos del Uruguay. Análisis con marcadores moleculares. Arch. Zoot. 51,195-202. [ Links ]
76. Puig, F. (1984) Polimorfismo de la albumina y la transferrina en caballos Criollo de Paso [Cuba]. Revista de Salud Animal. CU. 6(2), 285-291. [ Links ]
77. Quinteros, I.R., Miller, W.J., Tejedor, E.D., Poli, M.A., Antonini De Ruiz, A.A. (1980) Investigaciones inmunogenéticas en el Bovino Criollo Argentino. Marcadores genéticos. Analecta Veterinaria XII, 37-60. [ Links ]
78. Quinteros, I.R., Muller, A.O., Tejedor, E.D., Garcia Valenti H., Bischoff, J.R. (1973) Algunos marcadores genéticos en bovinos criollos de Argentina 1. Inmunogenética. Analecta Veterinaria 4, 1-3. [ Links ]
79. Quiroz-Valiente, J., Martínez, A., Ulloa R., Vega-Pla, J. (2006) Caracterización genética de las poblaciones bovinas Criollas de México. VII Simposio Iberoamericano sobre Conservación y Utilización de Recursos Zoogenéticos. Cochabamba -Bolivia. 220-222. [ Links ]
80. Rexroad 3rd, C.E., Owens, E.K., Johnson, J.S., Womack, J.E. (2000) A 12,000 rad whole genome radiation hybrid panel for high resolution mapping in cattle: characterization of the centromeric end of chromosome 1. Anim. Genet. 31, 262-5. [ Links ]
81. Rincón, G., D'Angelo, M., Gagliardi, R., Kelly, L., Llambí, S., Postiglioni, A. (2000) Genomic polymorphism in Uruguayan Creole cattle using RAPD and microsatellite markers. Res. Vet. Sci. 69,171-174. [ Links ]
82. Ripoli, M.V., Corva, P., Antonini, A., De Luca, J.C., Rojas, F., Dulout, F. N., Giovambattista G. (2003) Asociación entre cinco genes candidatos y producción de leche en la raza Criolla Saavedreña. Archivos de Zootecnia 52, 89-92. [ Links ]
83. Ripoli, M.V., Corva, P., Giovambattista G. (2006) Analysis of a polymorphism in the DGAT1 gene in 14 cattle breeds through PCR-SSCP methods. Res. Vet. Sci. 80, 287-290. [ Links ]
84. Ripoli, M.V., Díaz, S., Peral-García, P., Giovambattista, G. (2002) Nucleotide sequence of the upstream regulatory region of the BoLA-DRB. Eur. J. Immunogenet. 29, 537-540. [ Links ]
85. Ripoli, M.V., Lirón, J.P., De Luca, J.C., Rojas, F., Dulout, F. N., Giovambattista G. (2004) Gene Frequency Distribution of the BoLA-DRB3 in the Dairy "Saavedreño" Creole Cattle. Bioch. Genet. 42, 231-240. [ Links ]
86. Ripoli, M.V., Peral-García, P., Dulout, F.N., Giovambattista, G. (2004) Polymorphism in the Bovine BoLA-DRB3 Upstream Regulatory Regions detected through PCR-SSSP and DNA sequencing. Gene 339, 71-78. [ Links ]
87. Rouse, J.E. (1973) World cattle, III: Cattle of North America. Norman (University of Oklahoma Press), USA . [ Links ]
88. Sereno F.T.P.S, Sereno J.R.B., Vega-Pla,J.L., Kelly, L., Delgado Bermejo, J.V. (2008) Genetic diversity of Brazilian Pantaneiro horse and relationships among horse breeds. Pesq. Agropec. Bras. 43, 595-604. [ Links ]
89. Steigleder, C.S., Almeida, E.A., Weimer, T.A. (2004) Genetic diversity of a brazilian Creole cattle based on fourteen microsatellite loci. Arch. Zoot. 53, 3-11. [ Links ]
90. Thomas, H., (1998). The slave trade -the history of the Atlantic slave trade: 1440-1840. Macmillan, London. [ Links ]
91. Tryon, R.C., White, S.D., Bannasch, D.L. (2007) Homozygosity mapping approach identifies a missense mutation in equine cyclophilin B (PPIB) associated with HERDA in the American Quarter Horse. Genomics 90, 93-102. [ Links ]
92. Valberg, S.J., Ward, T.L., Rush, B., Kinde, H., Hiraragi, H., et al. (2001) Glycogen branching enzyme deficiency in quarter horse foals. J. Vet. Intern. Med. 15, 572-580. [ Links ]
93. Vázquez-Flores, F., Alonso, R., Villegas-Sepúlveda, N., Arriaga, C., Pereira-Suárez, A.L., Mancilla, R., Estrada-Chávez, C. (2006) A microsatellite study of bovine solute carrier family 11 a1 (Slc11a1) gene diversity in Mexico in relation to bovine tuberculosis. Gen. Mol. Biol. 29, 503-507. [ Links ]
94. Villalobos Cortés, A.I., Martínez, A.M., Vega-Pla, J.L., Delgado, J.V. (2009) Caracterización genética de la población bovina Guabalá mediante microsatélites. Arch. Zootec. 58, 485-488. [ Links ]
95. Villegas Castagnasso, E.E., Ripoli, M.V. (2010) Capítulo 2: Identificación individual en animales domésticos, en Genética de Animales Domésticos (Giovambattista, G., Peral-García, P.) Editorial Inte-Médica, Ciudad Autónoma de Buenos Aires - Argentina, 17-27. [ Links ]
96. Villegas-Castagnasso, E.E., Diaz, S., Giovambattista, G., Dulout, F.N., Peral-Garcia, P. (2003) Analysis of ELA-DQB Exon 2 Polymorphism in Argentine Creole Horses by PCR-RFLP and PCR-SSCP. J. Vet. Med. A. Physiol. Pathol. Clin. Med. 50, 280-285. [ Links ]
97. Vinocur, M.E., Brass, K.E., Batistella Rubin M.I., Mondino Silva C.A. (2003) Genetic variability in the brazilian criollo horse breed. Ciência Rural, Santa Maria, 33(1), 137-142. [ Links ]
98. Wade, C.M., Giulotto, E., Sigurdsson, S., Zoli, M., Gnerre, S., et al. (2009) Genome sequence, comparative analysis, and population genetics of the domestic horse. Science 326, 865-867. [ Links ]
99. Zamorano, M.J., Género, E.R., Rodero, A., Vega Pla, J.L., Rumiano, F.J. (1998) Caracterización genética del ganado bovino Criollo Argentino utilizando microsatélites. Arch. Zoot. 47, 273-277. [ Links ]
Received: 18/05/2010
Accepted: 04/06/2010














