Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
BAG. Journal of basic and applied genetics
versión On-line ISSN 1852-6233
BAG, J. basic appl. genet. vol.27 no.2 Ciudad Autónoma de Buenos Aires dic. 2016
ARTÍCULOS ORIGINALES
Mecanismos de regulación epigenética del ADN mitocondrial
Mechanisms for the epigenetic regulation of mitochondrial DNA
Sierra-Delgado J.A.1,2*, Contreras-García G.A.3
1 Médica, Candidata a Máster en Ciencias Básicas Biomédicas, Grupo de Genética Humana, Universidad Industrial de Santander, Bucaramanga, Santander, Colombia.
2 Médica, Grupo de Investigación en Medicina Genómica y Metabolismo FCV, Fundación Cardiovascular de Colombia, Bucaramanga, Santander, Colombia.
3 Médico Genetista, especialista en Bioética, Grupo de Genética Humana, Universidad Industrial de Santander, Departamento de Pediatría, Hospital Universitario de Santander, Bucaramanga, Santander, Colombia.
*Corresponding author: andreasierrad@outlook.com
Fecha de recepción: 22/02/2016
Fecha de aceptación de versión final: 27/10/2016
RESUMEN
La mitocondria es uno de los organelos celulares más importantes para el correcto funcionamiento de la maquinaria celular eucariota. Dentro de la epigenética tradicional cumple un papel primordial al formar parte del metabolismo de un carbono y el ciclo del folato; sin embargo, en mamíferos existen mecanismos epigenéticos específicos poco estudiados, que regulan la síntesis y expresión del ADN mitocondrial (ADNmt), campo denominado mitoepigenética. El ADNmt es un ADN circular de 16.6 kb, sin intrones que codifica 14 proteínas, 2 ARNr y 22 ARNt. Se encuentra empaquetado en nucleoides, compuestos por proteínas análogas a las histonas llamadas factor de transcripción mitocondrial A (TFAM). Existe evidencia de regulación epigenética del ADNmt en mamíferos desde 1984, sin embargo, su hallazgo permaneció controversial hasta el 2011, cuando se demostró la translocación de DNMT1 desde el núcleo hacia la mitocondria. Desde entonces se han descrito otros mecanismos entre los que se encuentran la presencia de 5-metilcitosinas (5mC) en patrón CpG y no-CpG, 5-hidroximeltilcitosinas (5hmC), la remodelación de nucleoides y el ARN de interferencia. De igual manera estos patrones de regulación se han correlacionado con niveles aberrantes en la transcripción de genes mitocondriales. Se ha encontrado asociación de estos cambios con entidades patológicas, como cáncer, NASH (esteatopatía no alcohólica), enfermedades neurológicas y retinopatía diabética, así como cambios ambientales e incluso envejecimiento. A partir de estos resultados y los avances recientes, se podría considerar la mitoepigenética como un campo de estudio fascinante y creciente, que puede contribuir al entendimiento de la fisiopatología de enfermedades complejas.
Palabras clave: Epigénesis Genética; Metilación de ADN; ADN mitocondrial; Mitocondria.
ABSTRACT
The mitochondrion is one of the most important cellular organelles for the right functioning of the eukaryotic cell. It plays a fundamental role in traditional epigenetics, because it takes part in one-carbon metabolism and the folate cycle. However, in mammals, the epigenetic mechanisms regulating the synthesis and expression of mitochondrial DNA (mtDNA), a field of study called mitoepigenetics, are understudied. The mtDNA is a circular DNA molecule of 16.6 kb without introns, which codes for 14 proteins, two rRNA and 22 tRNA. The mtDNA is packaged in nucleoids, composed of histone analogs called TFAM (Transcription factor A of Mitochondria). In mammals, evidence of epigenetic regulation of mtDNA has been known since 1984; however, the finding remained controversial until 2011, when the translocation of nuclear DNMT1 to the mitochondria was proved. Since then, there have been reports of other epigenetic mechanisms, which include the presence of 5-methylcytosines in CpG and Non-CpG pattern, 5-hidroximetilcytosines (5HmC), nucleoids remodeling and interference RNA. Similarly, these regulation patterns have been related to aberrant levels of transcription of mitochondrial genes. Association of this aberrant transcription with pathologies like cancer, NASH (not-alcoholic esteatopathy), neurodegenerative diseases and diabetic retinopaty, but also with environmental changes and even ageing, have been described. According to these results and the recent advances in the field, mitoepigenetics should be considered as a fascinating and growing field of study, because it might contribute to the understanding of the pathophysiology of complex diseases.
Key words: Epigenesics; Genetic; DNA methilation; Mitochondrial DNA; Mitochondria.
INTRODUCCIÓN
La mitocondria es, indudablemente, uno de los organelos celulares más importantes para el correcto funcionamiento de la maquinaria celular eucariota. Es la encargada de la producción aeróbica de ATP por medio de un proceso denominado fosforilación oxidativa, el cual es mediado por complejos enzimáticos multiproteícos en su membrana, encargados de transporte en cadena de electrones (Saraste, 1999). De igual manera, la mitocondria juega un papel fundamental en la apoptosis, la generación de especies reactivas de oxígeno y cuenta con funciones tejido específicas, como el metabolismo del colesterol, la biosíntesis del grupo hemo y la detoxificación de derivados del amonio en tejido hepático, entre otros (Skorecki et al., 2012). Sin embargo, a pesar de su rol importante en el mantenimiento de la homeostasis y el metabolismo, se conoce poco de la relación entre la mitocondria y los mecanismos epigenéticos específicos que regulan su síntesis y funcionamiento en mamíferos, un campo novedoso conocido como mitoepigenética.
Dentro de la epigenética tradicional la mitocondria cumple un papel primordial (van der Wijst y Rots, 2015). La mitocondria participa en la regulación del epigenoma al formar parte del metabolismo de un carbono a través de la producción de ATP y folato (Taylor y Tumbull, 2005; Locasale, 2013). Las mitocondrias regulan el cambio entre S-AdenosilMetionina (SAM), un donante de grupos metilo, y la síntesis de nucleótidos por la acción de la enzima mitocondrial bifuncional, el cual es un paso fundamental en la metilación del ADN (Iacobazzi et al., 2013).
ADN MITOCONDRIAL: MECANISMOS MOLECULARES
La mitocondria, junto al ribosoma, son los únicos organelos en la célula animal cuya composición incluye ácidos nucleicos. El ADN mitocondrial (ADNmt) fue descubierto en 1963 por Margit M.K. Nass- Edelson y Sylvan Nass mediante microscopía de electrones (Nass y Nass, 1963), sin embargo, su presencia se intuía aún antes debido a la similaridad ultraestructural entre la mitocondria y las bacterias. Por este motivo se propuso la teoría de la endosimbiosis, en la cual se formula que durante el proceso evolutivo células bacterianas fueron engullidas y modificadas para convertirse en organelos eucariotas, como la mitocondria (Manev et al., 2012). El ADNmt es un ADN circular de 16,6 kb, el cual representa menos de 1% del ADN total de una célula y es transmitido casi exclusivamente por linaje materno. Su composición consta de 2 cadenas, 1 ligera y 1 pesada (Figura 1), no contiene intrones y codifica 13 proteínas en total, todas parte de la cadena de transporte de electrones. El ADNmt también contiene el bucle de desplazamiento (D-Loop), una región no codificante en el ADNmt que contiene elementos que regulan la replicación y la transcripción en cis. En el D-Loop también se ubican las regiones hipervariables 1 y 2 (HVR1 y HVR2), las cuales son los sitios de mayor acúmulo de mutaciones dentro de la secuencia (Gosh et al., 2015). El genoma mitocondrial también transcribe 2 ARNr y 22 ARNt necesarios para la traducción del ADNmt (Minocherhomji et al., 2012). Otros estudios han encontrado un 14vo. polipéptido de 21 a 24 aminoácidos, llamado humanina, factor protector para el desarrollo de la enfermedad de Alzheimer, el cual es codificado por el gen MT-RNR2 (Maximov et al., 2002). El resto de elementos necesarios para el correcto funcionamiento de la mitocondria son codificados nuclearmente y son transportados por complejos proteicos dentro y fuera de la membrana mitocondrial. El ADNmt no contiene histonas, sin embargo, se encuentra recubierto por proteínas y empaquetado en agregados llamados nucleoides o mitocromosoma. El factor de transcripción mitocondrial A (TFAM) es el componente más abundante del nucleoide, es análogo a las histonas y juega un rol importante en el empaquetamiento y compactación del ADNmt. Los datos indican que la mayoría de los nucleoides tienen sólo una copia de ADNmt (Manev et al., 2012).
La replicación del ADNmt es mediada de manera exclusiva por la polimerasa γ, la cual cumple doble función: y reparar, por lo que existe una disminución en la fidelidad por una baja corrección de errores de la replicación en comparación con el genoma nuclear (Skorecki et al., 2012). Sin embargo, debido al alto número de copias de ADNmt (hasta 1.000 copias por célula), estas mutaciones no conllevan necesariamente efectos fenotípicos, sino que aumentan el nivel de heteroplasmia de la célula, definida como la divergencia entre las copias de ADNmt dentro la misma; siempre que no se supere un umbral de heteroplasmia, diferente para cada tejido y tipo de célula, no va a existir enfermedad o alteración, lo que se conoce como mitocheckpoint (Minocherhomji et al., 2012). Existe un mayor nivel de heteroplasmia en células post-mitóticas que en los tejidos que se dividen rápidamente (Manev et al., 2012).
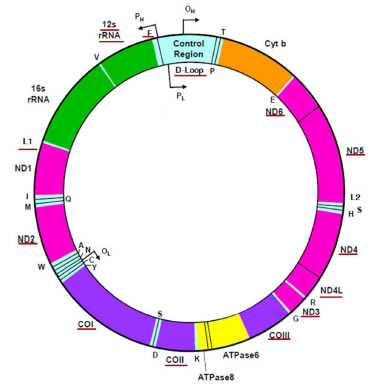
Figura 1. Diagrama del mtDNA. Se representan la posición de los 37 genes, la región de control (D-Loop) y los orígenes de replicación de las cadenas pesada (OH, PH) y ligera (OL, PL). Genes que han sido reportados con metilación o transcripción diferencial se encuentran subrayados. Modificado de www.mitomap.org (Lott et al., 2013).
MITOEPIGENÉTICA Y REGULACIÓN
En 1984, Pollack y su grupo publicaron reportes que indicaban la presencia de 5-Metilcitosinas (5mC) en ADNmt en tejido murino (Pollack et al., 1984). El equipo de Pollack observó que, en comparación con el ADN no codificante (ADNnc), el ADNmt se encontraba generalmente submetilado, con sólo un 1-5 % de las citosinas metiladas. Sin embargo, la presencia de metilación en el ADN mitocondrial fue controvertida en la comunidad científica (Manev et al, 2012; Chinnery et al, 2012), hasta que los experimentos de Shock et al. (2011) demostraron la translocación de DNMT1 (ADN Metiltransferasa-1) nuclear a la matriz mitocondrial (mtDNMT1) en fibroblastos murinos. En condiciones normales la DNMT1 es la encargada del mantenimiento de los patrones de metilación en el ADN nuclear en mamíferos. La mtDNMT1 es una isoforma de la enzima que contiene tanto el dominio catalítico de la DNMT1 como la secuencia diana mitocondrial. La migración de la DNMT hacia la mitocondria es un proceso regulado mediante la expresión de una secuencia diana conservada corriente arriba del sitio de inicio de transcripción dentro del gen codificado nuclearmente. Estudios posteriores han encontrado que la migración de mtDNMT1 está regulada positivamente por factores que responden al estrés oxidativo, como el PGC1 α (Coactivador α del receptor activado por proliferadores peroxisomales γ), el factor nuclear respiratorio 1 (NRF1), y la liberación de p53 a partir del promotor DNMT1 (Balinang, 2012). Bajo condiciones basales, la transcripción de mtDNMT1 constituye alrededor del 2 % del total de la DNMT1 (Shock et al., 2011).
Adicional a la presencia de mtDNMT1, estudios posteriores en fracciones mitocondriales provenientes de cultivos murinos y sistema nervioso central humano detectaron la presencia de DNMT3a, pero bajos niveles de mtDNMT1, lo cual podría sugerir que la localización mitocondrial de DNMT3a es tejido-específica (Chestnut et al., 2011; Wong et al., 2013). Cabe recordar que la DNMT3, en el núcleo, es la encargada de regular los patrones de metilación de novo (Shock et al., 2011). En un estudio posterior, Bellizzi et al. (2013) reportaron citosinas metiladas e hidroximetiladas en el D-Loop de las muestras analizadas, encontrando que la metilación en el ADNmt se da principalmente en un patrón no-CpG en la región promotora de la cadena pesada y en bloques de secuencias conservadas, lo que sugiere su implicación en la regulación de la replicación y/o transcripción del ADNmt. El inactivar las DNMTs 1, 3a y 3b en células madre embrionarias de ratón resulta en una reducción de la metilación CpG, pero no la metilación no-CpG, lo cual sugiere que la modificación epigenética del D-loop sólo se establece parcialmente por estas enzimas (Bellizzi et al., 2013).
Ghosh et al. (2014) analizaron 39 conjuntos de datos presentes en bases de datos públicas a fin de caracterizar el metiloma mitocondrial, encontrando que el patrón y la distribución de 5mC son relativamente constantes a través de la mitocondria, con la excepción de unos pocos loci metilados diferencialmente en diversos tejidos durante diferentes puntos en el tiempo; coincidentemente, estos loci han sido relacionados con enfermedades mitocondriales (Tabla 1). Estos resultados muestran que es plausible que la variabilidad en la metilación puede influir potencialmente en la regulación génica, con la consiguiente diversidad fenotípica.
Tabla 1. Recopilación de los genes reportados con metilación selectiva, transcriptos y funciones correspondientes. Se listan los genes que codifican para las 13 proteínas de la cadena de transporte de electrones, las dos subunidades del ribosoma mitocondrial (uno de los cuales codifica para la proteína Humanina, factor neuroprotector e inhibidor de apoptosis) y los dos ARN de transferencia que han mostrado metilación diferencial. Los principales genes reportados con cambios epigenéticos en diversas condiciones son MT-ND2, MT-ND6, que codifican subunidades del complejo NADH Deshidrogenasa y MT-RNR1, que codifica para la subunidad 12S del ribosoma mitocondrial. ECV (Enfermedad cardiovascular), NASH (Esteatopatía no alcohólica).

OTROS MECANISMOS DE REGULACIÓN
La presencia de 5-hidroximetilcitosina (5hmC) ha sido confirmada en ADNmt de mamíferos (Shock et al., 2011), en una densidad significativamente mayor en el ADNmt que en ADNnc (Sun et al., 2013). Estudios realizados por Dzitoyeva et al. (2012) encontraron presencia de proteínas TET (TET1 y TET2) por inmunoreactividad en fracciones mitocondriales de cultivos neuronales, sin embargo, no se ha confirmado de manera concluyente la actividad de dichas proteínas en la mitocondria (Sadakierska- Chudy et al., 2014; Gosh et al., 2015). La expresión coordinada de genes nucleares y mitocondriales parece ser particularmente importante durante la división celular y la biogénesis mitocondrial, los estudios sugieren que existe una correlación entre la regulación transcripcional de ADNmt mediada por 5hmC, 5mC y genes codificados en el núcleo (Minocherhomji et al., 2012). Sin embargo, no sólo la metilación tiene un componente importante en la regulación epigenética del ADNmt, las alteraciones en el contenido de TFAM determinan un cambio en el mitocromosoma y exponen al ADN a la acción de DNMTs. Se ha encontrado que las DNMTs tienen accesibilidad a diferentes sitios en ADNmt en función del nivel de ocupación de la proteína, y que existe correlación entre el nivel de empaquetamiento del nucleoide con el porcentaje de metilación e incluso con la presencia de ARN de interferencia en mitocondria (Rebelo et al., 2009 ; Chinnery et al, 2012 ). De manera curiosa, se ha descrito una regulación negativa del TFAM en amniocitos cultivados de pacientes con trisomía 21 en comparación con fetos normales (Infantino et al., 2011).
En ratas, un pequeño grupo de micro ARN (ARNmi) ha sido identificado en derivados altamente purificados de mitocondrias hepáticas; estudios sugieren que estos regulan la expresión de genes relacionados con la apoptosis, la proliferación celular y la diferenciación celular (Kalani et al., 2014). Evidencia de esta regulación por ARNmi se encontró al hallar la presencia de argonauta 2 en extractos mitocondriales (Sadakierska-Chudy et al., 2014). Das et al. (2012) también hallaron evidencia de regulación mitocondrial del ADNmt a través de ARNmi en tejido cardíaco. Un diagrama resumiendo los diversos mecanismos de control epigenético en el ADNmt se puede encontrar en la Figura 2.

Figura 2. Un modelo que resume los principales mecanismos epigenéticos que regulan la expresión del ADN mitocondrial. El ADN nuclear codifica proteínas que involucran modificaciones epigenéticas dentro del núcleo y la mitocondria. Entre los mecanismos de regulación se encuentran en primer lugar, la metilación del ADNmt, la cual, en patrón CpG está mediado por las DNMTs 1 y 3a, las cuales son importadas desde el núcleo. Se desconocen los mecanismos involucrados en la metilación No CpG. Otros mecanismos incluyen la hidroximetilación, mediada por proteínas TET cuya actividad intramitocondrial no se ha podido confirmar de manera concluyente a pesar de encontrarse en fracciones mitocondriales, la remodelación de los nucleoides, regulada por la metilación de loci mitocondriales y cambios en TFAM y por último la regulación por medio de ARNmi, producidos por argonauta 2 mitocondrial. Estudios han ligado estos cambios a desencadenantes como patologías, contaminantes ambientales y envejecimiento. TFAM: factor de transcripción mitocondrial A, ADNmt: ADN mitocondrial, 5hmC: 5-hidroximetilcitosina, 5mC: 5-metilcitosina, ARGO-2: argonauta 2 mitocondrial, DNMT1: ADN Metiltransferasa-1, DNMT3A: ADN Metiltransferasa-3A.
ROL DEL MITOEPIGENOMA EN PATOLOGÍAS
Además de la diferencia en la presencia de TFAM en amniocitos con trisomía 21, otras modificaciones epigenéticas han sido correlacionadas o ligadas a entidades clínicas; por ejemplo, se encontró un aumento de niveles de DNMT1 y DNMT3A en mitocondrias de las motoneuronas en pacientes con esclerosis lateral amiotrófica respecto a controles sanos, lo cual sugiere que existen mecanismos que pueden contribuir a la muerte celular mediada por metilación del ADNmt (Chestnut et al., 2011). Asimismo, se ha demostrado que la hipermetilación en sitios de unión de la polimerasa γ en el genoma mitocondrial durante estados hiperglucémicos podría potencialmente modular la memoria epigenética en retinopatía diabética (Ghosh et al., 2014; Kowluru et al., 2015). En estudios realizados por Feng y colaboradores (2012) también se ha observado una correlación directa entre la expresión de MT-ND2 mitocondrial y la demetilación de su secuencia de promotor en el ADNmt (D-Loop) en pacientes con cáncer colorrectal, encontrando que, en los fenotipos neoplásicos, la expresión de ND2 se aumentaba mientras que la metilación disminuía. Esta diferencia fue significativa incluso en estadios tempranos de cáncer (Feng et al., 2012).
La evidencia sugiere que no sólo las enfermedades sino también factores como la exposición ambiental o el envejecimiento pueden afectar los niveles de metilación. El envejecimiento afecta los niveles de 5-hidroximetilcitosina en el ADNmt en la corteza frontal del cerebro en ratones y disminuye los niveles de transcripción de proteínas relacionadas con el complejo I de la fosforilación oxidativa (Dzitoyeva et al., 2012). La exposición de cultivos celulares a estrés oxidativo derivado de la contaminación ambiental también ha mostrado una alteración en la metilación de los genes ND6 y 12SrRNA (Giordano et al., 2012; Iacobazzi et al., 2013) y el estrés oxidativo secundario a la exposición del etanol in vitro conlleva a un aumento en el nivel de proteína mtDNMT1 seguido por el aumento del contenido de 5mC y 5hmC en el ADNmt (Balinang, 2012). In vivo, estudios en pacientes expuestos a material particulado rico en metales muestran una metilación selectiva en los genes MT-TF y MT-RNR1 con respecto a controles con baja exposición (Byun, 2013; Byun y Baccarelli, 2014). De manera interesante, el estudio ENVIRONAGE (Environmental influence on early ageing) (Jannsen et al., 2015) evaluó una cohorte de 381 gestantes con diferentes grados de exposición a material particulado durante el curso de su embarazo. Al estudiar el nivel de metilación en el gen MT-RNR1 y el D-Loop en mitocondrias provenientes de la placenta de las mujeres, se encontró un aumento en la metilación en ambos loci. El porcentaje de metilación fue significativamente mayor cuando las gestantes se exponían al material particulado durante el primer trimestre de embarazo. La metilación aumentada también se relacionaba con disminución del número de copias del ADNmt en placenta, por lo cual los investigadores proponen que la exposición temprana a contaminantes ambientales (material particulado) podría llevar a mitofagia, disminución del ADNmt y muerte mitocondrial en el tejido placentario. Uno de los casos más claros de la correlación entre la metilación diferencial de pacientes sanos y fenotipos patológicos en humanos vino a partir de los experimentos realizados por Pirola et al. (2013), quienes estudiaron los cambios moleculares que median la transición de un estado sin daño hepático (esteatosis simple) a una enfermedad más agresiva (esteatohepatitis no alcohólica- NASH). El equipo de Pirola (2013) encontró patrones de metilación diferencial en el gen que codifica para la deshidrogenasa NADH6 (MT-ND6), el cual se encuentra altamente metilado en el hígado de pacientes con NASH, pero no en pacientes con esteatosis simple. Este efecto mostró un impacto significativo en la regulación transcripcional del MTND6: en pacientes con NASH se encontró una menor actividad transcripcional y expresión de la proteína NADH6 a comparación con la esteatosis simple. Este patrón de metilación podría correlacionarse con el hallazgo de mayor expresión de la mtDNMT1 en pacientes con NASH (Pirola et al., 2013).
Asimismo, Baccarelli y Byun (2015) estudiaron el grado de metilación del ADNmt en plaquetas como un marcador patológico en enfermedad cardiovascular (ECV). Las plaquetas, al carecer de ADN nuclear, constituyen un cultivo interesante para el estudio de la regulación del ADN mitocondrial. En este estudio los autores compararon el porcentaje de metilación de 7 genes mitocondriales (MT-CO1, MT-CO2, MT-CO3, MT-TL1, MT-ATP6, MT-ATP8, MT-MD5) por medio de pirosecuenciación de ADN tratado con bisulfito en pacientes con ECV y controles sanos. Encontraron diferencias estadísticamente significativas en la metilación de los genes MT-CO1, MTCO2, MT-CO3 y MT-TL1, en su mayoría encargados de la síntesis de ATP. Dichos genes se encuentran más metilados en pacientes que presentan ECV que en controles sanos, sin importar edad, índice de masa corporal o raza (Baccarelli y Byun, 2015). Una recopilación de los genes que se han encontrado con metilación selectiva, su transcrito y función puede encontrarse en la Tabla 1. Estos hallazgos que sugieren que la expresión de genes mitocondriales está regulada por modificaciones epigenéticas y pueden desempeñar un papel importante en la patogénesis de la progresión de enfermedades, o como biomarcador temprano de patogénesis; asimismo los estudios muestran que dichos cambios epigenéticos podrían ser susceptibles a factores ambientales y como tal, podrían utilizarse como marcadores de polución o cambio ambiental, mostrando patrones diferenciales incluso en gestación.
CONCLUSIÓN
La mitoepigenética ha sido un campo altamente controversial y pobremente estudiado desde el descubrimiento de 5mC en el ADN mitocondrial en 1984. Sin embargo, estudios recientes han probado la existencia de mecanismos epigenéticos tales como la metilación e hidroximetilación del ADNmt mediante la respectiva migración de enzimas nucleares como la DNMT a la mitocondria. También se ha comprobado la existencia de otros mecanismos epigenéticos como la remodelación de nucleoides y la presencia de miARN.
Estas alteraciones en los patrones de regulación se han correlacionado con niveles aberrantes en la transcripción y producción de ciertas proteínas mitocondriales y se ha encontrado asociación con entidades patológicas y cambios ambientales. A partir de estos resultados y los avances recientes en este campo, se podría considerar la epigenética mitocondrial como un campo de estudio fascinante, que podría dilucidar parte de los mecanismos moleculares detrás de enfermedades complejas, y que incluso podría ser usado como un biomarcador predictor en el futuro.
AGRADECIMIENTOS
Agradecimiento especial al grupo de genética Humana de la Universidad de Los Andes y a la clase de epigenética del Doctor Valeriano López, donde nació el interés por el tema.
BIBLIOGRAFÍA
1. Baccarelli A.A., Byun H.M. (2015) Platelet mitochondrial DNA methylation: a potential new marker of cardiovascular disease. Clinical epigenetics, 7 (1): 44. [ Links ]
2. Balinang J. (2012) The Regulation of Mitochondrial DNMT1 During Oxidative Stress. M.S. thesis, Virginia Commonwealth University, Richmond, Virginia. [ Links ]
3. Bellizzi D., DAquila P., Scafone T., Giordano M., Riso V., Riccio A., Passarino G. (2013) The control region of mitochondrial DNA shows an unusual CpG and non-CpG methylation pattern. DNA research, 20 (6): 537-547. [ Links ]
4. Byun H.M., Baccarelli A.A. (2014) Environmental exposure and mitochondrial epigenetics: study design and analytical challenges. Human genetics 133 (3): 247-257. [ Links ]
5. Byun H.M., Panni T., Motta V., Hou L., Nordio F., Apostoli P., Baccarelli A.A. (2013) Effects of airborne pollutants on mitochondrial DNA methylation. Part Fibre Toxicol. 10 (1): 18. [ Links ]
6. Chestnut B.A., Chang Q., Price A., Lesuisse C., Wong M., Martin L.J. (2011) Epigenetic regulation of motor neuron cell death through DNA methylation. J. Neurosci. Nov. 16; 31 (46): 16619-36. [ Links ]
7. Chinnery P.F., Elliott H.R., Hudson G., Samuels D.C., Relton C.L. (2012) Epigenetics, epidemiology and mitochondrial DNA diseases. Int. J. Epidemiol. Feb; 41 (1): 177-87. [ Links ]
8. Das S., Ferlito M., Kent O.A., Fox-Talbot K., Wang R., Liu D., Raghavachari N., Yang Y., Steenbergen C. (2012) Nuclear miRNA regulates the mitochondrial genome in the heart. Circulation research 110 (12): 1596-1603. [ Links ]
9. Dzitoyeva S., Chen H., Manev H. (2012) Effect of aging on 5-hydroxymethylcytosine in brain mitochondria. Neurobiology of aging 33 (12): 2881- 2891. [ Links ]
10. Feng S., Xiong L., Ji Z., Cheng W., Yang H. (2012) Correlation between increased ND2 expression and demethylated displacement loop of mtDNA in colorectal cancer. Mol. Med. Rep. 6: 125-130. [ Links ]
11. Ghosh S., Sengupta S., Scaria V. (2014) Comparative analysis of human mitochondrial methylomes shows distinct patterns of epigenetic regulation in mitochondria. Mitochondrion. Jul 22, pii: S1567-7249 (14). [ Links ]
12. Ghosh S., Singh K.K., Sengupta S., Scaria V. (2015) Mitoepigenetics: The different shades of grey. Mitochondrion 25: 60-66. [ Links ]
13. Giordano M., Cristiani C., Crocco P., DAquila P., De Rango F., Pisani F., Scafone T., Tallaro F., Rose G., Passarino G., Bellizzi D. (2012) Methylation of the human mitochondrial 12S rRNA gene is correlated with aging. In 12th International FISV Congress, Rome, Italy (Vol. 9). [ Links ]
14. Iacobazzi V., Castegna A., Infantino V., Andria G. (2013) Mitochondrial DNA methylation as a next-generation biomarker and diagnostic tool. Mol. Genet. Metab. Sep-Oct; 110 (1-2): 25-34. [ Links ]
15. Infantino V., Castegna A., Iacobazzi F., Spera I., Scala I., Andria G., Iacobazzi V. (2011) Impairment of methyl cycle affects mitochondrial methyl availability and glutathione level in Downs syndrome. Molecular genetics and metabolism 102 (3): 378-382. [ Links ]
16. Janssen B.G., Byun H.M., Gyselaers W., Lefebvre W., Baccarelli A.A., Nawrot T.S. (2015) Placental mitochondrial methylation and exposure to airborne particulate matter in the early life environment: An ENVIRONAGE birth cohort study. Epigenetics 10 (6): 536-44. [ Links ]
17. Kalani A., Kamat P.K., Voor M.J., Tyagi S.C., Tyagi N. (2014) Mitochondrial epigenetics in bone remodeling during hyperhomocysteinemia. Mol. Cell Biochem. Oct; 395 (1-2): 89-98. [ Links ]
18. Kowluru R.A., Kowluru A., Mishra M., Kumar B. (2015) Oxidative stress and epigenetic modifications in the pathogenesis of diabetic retinopathy. Progress in retinal and eye research 48: 40-61. [ Links ]
19. Locasale J.W. (2013) Serine, glycine and the one-carbon cycle: cancer metabolism in full circle. Nature Reviews Cancer 13 (8): 572-583. [ Links ]
20. Lott M.T., Leipzig J.N., Derbeneva O., Xie H.M., Chalkia D., Sarmady M., Procaccio V., Wallace D.C. (2013) mtDNA variation and analysis using MITOMAP and MITOMASTER. Current Protocols in Bioinformatics 1 (123): 1.23.1-26. PMID: 25489354 URL: http://www.mitomap.org. [ Links ]
21. Manev H., Dzitoyeva S., Chen H. (2012) Mitochondrial DNA: A Blind Spot in Neuroepigenetics. Biomol. Concepts Apr.; 3 (2): 107-115. [ Links ]
22. Maximov V., Martynenko A., Hunsmann G., Tarantul V. (2002) Mitochondrial 16S rRNA gene encodes a functional peptide, a potential drug for Alzheimers disease and target for cancer therapy. Medical hypotheses 59 (6): 670-673. [ Links ]
23. Minocherhomji S., Tollefsbol T.O., Singh K.K. (2012) Mitochondrial regulation of epigenetics and its role in human diseases. Epigenetics Apr.; 7 (4): 326-34. [ Links ]
24. Nass M.M., Nass S. (1963) Intramitochondrial fibers with DNA characteristics I. Fixation and electron staining reactions. The Journal of cell biology 19 (3): 593-611. [ Links ]
25. Pirola C.J., Gianotti T.F., Burgueño A.L., Rey-Funes M., Loidl C.F., Mallardi P., Martino J.S., Castaño G.O., Sookoian S. (2013) Epigenetic modification of liver mitochondrial DNA is associated with histological severity of nonalcoholic fatty liver disease. Gut. Sep; 62 (9): 1356-63. [ Links ]
26. Pollack Y., Kasir J., Shemer R., Metzger S., Szyf M. (1984) Methylation pattern of mouse mitochondrial DNA. Nucleic acids research 12 (12): 4811-4824. [ Links ]
27. Rebelo A.P., Williams S.L., Moraes C.T. (2009) In vivo methylation of mtDNA reveals the dynamics of protein–mtDNA interactions. Nucleic acids research 37 (20): 6701-6715. [ Links ]
28. Sadakierska-Chudy A., Frankowska M., Filip M. (2014) Mitoepigenetics and drug addiction. Pharmacol. Ther. Nov; 144 (2): 226-233. [ Links ]
29. Saraste M. (1999) Oxidative phosphorylation at the fin de siecle. Science 283 (5407): 1488-1493. [ Links ]
30. Shock L.S., Thakkar P.V., Peterson E.J., Moran R.G., Taylor S.M. (2011) DNA methyltransferase 1, cytosine methylation, and cytosine hydroxymethylation in mammalian mitochondria. Proc. Natl. Acad. Sci. USA Mar 1; 108 (9): 3630-5. [ Links ]
31. Skorecki K., Mandel H. (2012) DNA mitocondrial y enfermedades y rasgos hereditarios. En: Longo D.L. (Ed.) Harrison Principios de Medicina Interna, Capítulo 18, parte 3. McGraw-Hill Interamericana Editores 18a edición. [ Links ]
32. Sun Z., Terragni J., Borgaro J.G., Liu Y., Yu L., Guan S., Zheng Y. (2013) High-resolution enzymatic mapping of genomic 5-hydroxymethylcytosine in mouse embryonic stem cells. Cell reports 3 (2): 567-576. [ Links ]
33. Taylor R.W., Turnbull D.M. (2005) Mitochondrial DNA mutations in human disease. Nat. Rev. Genet. May; 6 (5): 389-402. [ Links ]
34. Van der Wijst, M. G., & Rots, M. G. (2015). Mitochondrial epigenetics: an overlooked layer of regulation?. Trends in Genetics, 31(7), 353-356. [ Links ]
35. Wong M., Gertz B., Chestnut B.A., Martin L.J. (2013) Mitochondrial DNMT3A and DNA methylation in skeletal muscle and CNS of transgenic mouse models of ALS. Front Cell Neurosci. Dec. 25 (7): 279. [ Links ]














