Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
BAG. Journal of basic and applied genetics
versión On-line ISSN 1852-6233
BAG, J. basic appl. genet. vol.29 no.1 Ciudad Autónoma de Buenos Aires jun. 2018
ARTÍCULOS ORIGINALES
La arquitectura genética como herramienta de análisis del mapa genotipo-fenotipo
Genetic architecture as an analysis tool of the genotype-phenotype map
Petino Zappala M.A.1,*, Fanara J.J.1
1 Universidad de Buenos Aires, Facultad de Ciencias Exactas y Naturales, Departamento de Ecología, Genética y Evolución, Laboratorio de Evolución, CONICET-Universidad de Buenos Aires, Instituto de Ecología, Genética y Evolución de Buenos Aires (IEGEBA), Buenos Aires, Argentina.
* Autor correspondiente: mapz@ege.fcen.uba.ar
Fecha de recepción: 06/02/2017
Fecha de aceptación de versión final: 27/04/2018
RESUMEN
El estudio de la relación entre el genotipo y el fenotipo es de gran importancia para las investigaciones en genética y en las ciencias de la vida en general. A diferencia de la concepción tradicional de esta relación como un conjunto invariante de parámetros, el enfoque actual utiliza la arquitectura genética, una herramienta realista y dinámica que permite elucidar el mapa genotipo-fenotipo, ahora considerado una estructura en evolución. De las complejas relaciones entre los elementos del mapa genotipo-fenotipo surgen diversas propiedades emergentes que pueden explicar distintos fenómenos evolutivos. Además, algunas de estas propiedades promueven la acumulación de variabilidad genética en poblaciones naturales, la cual constituye el sustrato de procesos evolutivos como la selección natural. La caracterización y análisis de la arquitectura genética de caracteres adaptativos constituye una herramienta eficaz para comprender los procesos genéticos subyacentes al cambio evolutivo.
Palabras clave: Plasticidad; Canalización; Modularidad; Variabilidad genética críptica; “Evolvabilidad”.
ABSTRACT
Studying the relationship between genotype and phenotype is of great importance for genetics and life science studies in general. In contrast with the traditional view of this relationship as an invariant set of parameters, the current approach incorporates the concept of genetic architecture, a realistic and dynamic tool that allows to elucidate the genotype-phenotype map, which is now regarded as an evolving structure. From the complex relationships between the elements in the genotype-phenotype map several emergent properties arise that can explain different evolutionary phenomena. Moreover, some of these properties promote the accumulation of genetic variability in natural populations, which constitutes the substrate to evolutionary processes such as natural selection. The characterization and analysis of the genetic architecture of adaptive traits constitutes a powerful tool to understand the genetics underpinnings of evolution.
Key words: Plasticity; Canalization; Modularity; Cryptic genetic variability; Evolvability.
INTRODUCCIÓN
La biodiversidad es el resultado de una larga historia de complejas interacciones entre las especies y su ambiente, a lo largo de la cual aquéllas han evolucionado desarrollando adaptaciones manifestadas a través del fenotipo, que les permiten vivir y reproducirse. Comprender las bases del surgimiento y mantenimiento de esta diversidad es uno de los desafíos con los que se enfrentan la genética, la ecología y la biología evolutiva (Badyaev, 2011; Futuyma, 2013).
Determinar las relaciones entre genotipo y fenotipo y cómo éstas se modifican a lo largo de la evolución constituye un objetivo fundamental para la genética en particular y para las ciencias de la vida (biología, biomedicina, veterinaria, etc.) en general. El enfoque tradicional apuntaba a un vínculo estático en el que el genotipo era un “plano” (blueprint) para el fenotipo, y en el que las asociaciones entre estos dos factores estaban restringidas para un tiempo y espacio dado. Un ejemplo de esto es, para el caso de la genética de poblaciones, la heredabilidad, un parámetro poblacional circunscripto a las condiciones en las cuales dicho parámetro fue estimado. Sin embargo en 1991 Alberch planteó el concepto de mapa genotipo-fenotipo (Alberch, 1991; Houle et al., 2010), el cual incorpora niveles intermedios de organización de la información como el transcriptoma, proteoma y metaboloma, conectados mediante relaciones que se modifican en función del ambiente. A diferencia del modelo anterior, el mapa genotipo-fenotipo no es determinista, sino que se considera que el genotipo codifica un espectro de posibles fenotipos para cada carácter. Los distintos caracteres serán a su vez interdependientes y además tendrán una mayor o menor capacidad de permanecer inalterables frente a distintas perturbaciones, i.e. robustez (Kitano, 2004; Felix, 2015; DeWitt, 2016). Esta perspectiva de la relación entre la información genética (genotipo) y la manifestación de tal información (fenotipo) es capaz de explicar fenómenos relevantes en campos de creciente desarrollo experimental y conceptual como la biología evolutiva del desarrollo (evodevo), la genómica ecológica, la genética cuantitativa, entre otros, que no podrían derivarse del enfoque tradicional.
ARQUITECTURA GENÉTICA
El concepto del mapa genotipo-fenotipo se usa como metáfora para comprender las múltiples formas en las que la información genética influye en el fenotipo de un organismo (Pigliucci, 2010). Una forma de estudiar este sistema de relaciones genotipo-fenotipo (en cuanto a sus componentes y mecanismos de cambios) es a través del estudio de la arquitectura genética de caracteres complejos (Hansen, 2006). En este sentido, los procesos de adaptación, el surgimiento de variación a nivel poblacional y los mecanismos de especiación sólo pueden ser abordados cabalmente mediante la comprensión de la arquitectura genética de los caracteres adaptativos y estudiando su evolución (Badyaev, 2011).
Dilucidar la arquitectura genética de un carácter no sólo consiste en identificar los loci involucrados en los procesos subyacentes a la manifestación fenotípica de dicho carácter, sino también caracterizar sus propiedades variacionales, es decir, los efectos contexto-dependientes que contribuyen y/o modifican la expresión del carácter. Según Hansen (2006), la principal razón para estudiar la arquitectura genética es que permite comprender cómo las propiedades variacionales determinan el potencial evolutivo de los caracteres en cuestión.
Un análisis pormenorizado de la arquitectura genética requiere de la colaboración de distintas ramas de la biología, como la genética, la genómica, la bioinformática, la ecología y la biología evolutiva. Desde el punto de vista genético poblacional y evolutivo, descifrar la arquitectura genética de caracteres complejos implica determinar (Mackay, 2001; Boyle et al., 2017): i) la proporción de los loci involucrados en el carácter que son variables; ii) las bases moleculares de la variación entre alelos (polimorfismos de nucleótidos individuales o SNPs [single nucleotide polymorphisms]); iii) la plasticidad fenotípica y los efectos contexto-dependientes de caracteres adaptativos; iv) las fuerzas evolutivas que han gobernado el cambio evolutivo; y v) si la arquitectura genética varía a nivel filogenético. Si bien esta es una tarea compleja y que requiere de mucho esfuerzo, promete en el trayecto, nuevas miradas sobre el estudio de la variación natural (Anholt et al., 2003; Manolio et al., 2009; Mackay y Moore, 2014).
CARACTERIZACIÓN DE LA ARQUITECTURA GENÉTICA
Base genética La base genética de un carácter se refiere a todos los loci involucrados en los procesos que subyacen al establecimiento de ese carácter y sus efectos en él. Muchos de los caracteres de relevancia para estos campos son continuos y su base genética está constituida por muchos loci de efecto pequeño (Flint y Mackay, 2009; Paaby y Gibson, 2016). Este hecho dificulta su estudio, ya que los análisis de mutagénesis tradicionalmente utilizados involucran variantes de gran efecto, que usualmente no se presentan en la naturaleza en condiciones en las que sería esperable que un gran cambio fenotípico resultase deletéreo. Una de las técnicas que constituyó un avance sustancial en estos estudios fue el mapeo por QTL (Quantitative Trait Locus mapping), que buscaba identificar regiones específicas del genoma que pudiesen explicar las diferencias fenotípicas entre líneas, poblaciones o especies (Mackay, 2001; Fanara et al., 2002; Buckler et al., 2009; Carreira et al., 2016). Actualmente, los problemas de detección de variabilidad y su resolución, inherentes al protocolo de mapeo por QTL, se resuelven en gran medida con las técnicas de asociación del genoma completo (Genome Wide Association Studies, GWAS). Los protocolos metodológicos que se desarrollaron a partir del GWAS involucran una gran cantidad de líneas cuyo ADN está completamente secuenciado, proveyendo un mapa muy denso de sitios polimórficos que constituye la variabilidad genética (genómica) natural (Huang et al., 2014; Korte y Farlow, 2013). Con estos elementos y considerando el gran poder de cómputo disponible en la actualidad, se pueden realizar análisis de asociación entre las variabilidades genética y fenotípica, obteniéndose como resultado loci candidatos responsables de la variación fenotípica detectada, los que posteriormente pueden ser corroborados mediante la utilización de otras técnicas (Morozova et al., 2014).
Propiedades variacionales
Tanto una búsqueda (screening) de mutantes puntuales como la realización de un GWAS permiten llegar a una lista de variantes nucleotídicas asociadas a un cierto cambio fenotípico. Sin embargo, esta lista no aporta información acerca de los procesos que subyacen a dicho cambio de las relaciones entre las distintas variantes, ni acerca de los efectos que un cambio fenotípico pueda tener en otros caracteres, es decir, las propiedades variacionales de la arquitectura genética. Éstas involucran a lo que se denomina la arquitectura funcional, es decir la colección de caminos metabólicos (pathways) que conducen de la base genética al carácter mediante el mapa genotipo-fenotipo. Estas relaciones pueden caracterizarse por medio del estudio de distintos fenómenos emergentes de la estructura del mapa, los cuales se describen a continuación.
Pleiotropía y modularidad
Una variante genética es pleiotrópica cuando afecta a la vez a más de un carácter. Las complejas y flexibles redes génicas que actúan a lo largo de distintos procesos del desarrollo que subyacen a varios caracteres explican la generalidad de la pleiotropía que puede afectar, paralelamente, a distintos estadios ontogenéticos (Flint y Mackay, 2009; Lavagnino et al., 2008). El estudio de la interacción entre estos procesos (también conocido como integración fenotípica) permite una visión realista de los organismos estudiados y las dinámicas que siguen las poblaciones a lo largo de la evolución (Goswami et al., 2014). Cheverud (1996) afirma que esta integración fenotípica estructura la pleiotropía, de forma que la selección favorecerá una arquitectura del desarrollo caracterizada por la respuesta coordinada de caracteres relacionados funcionalmente. Por otro lado, si dos caracteres presentan combinaciones que resultan en óptimos de fitness, puede favorecerse una integración entre ellos por medio de alguna forma de pleiotropía como consecuencia de la selección correlacional (Schluter y Nychka, 1994; Sinervo y Svensson, 2002).
Los procesos pleiotrópicos que ocurren dentro de los individuos también pueden producir soluciones de compromiso (trade-offs), debido a condicionamientos y limitaciones de los caracteres por interacciones negativas (Lailvaux y Husak, 2014). Éstos se generan cuando para un genotipo determinado un aumento del fitness, como consecuencia de un cambio en un carácter, se asocia a una disminución del fitness por un cambio concomitante en otro carácter. Según el modelo de adquisición y asignación de recursos (acquisition-allocation model) (van Noordwijk y de Jong, 1986), estos compromisos entre caracteres se deberían a la necesidad de “repartir” los recursos disponibles entre varias funciones vitales, lo cual hace imposible optimizar todos los caracteres a la vez. Si bien el efecto de los loci pleiotrópicos puede determinar la existencia de correlaciones significativas entre caracteres, una falta de correlación no implica que no existan sectores del genoma afectándolos a la vez. El modelo de adquisición y asignación de recursos permite explicar las posibles faltas de correlación estadística entre caracteres, dado que tanto la adquisición como la asignación de recursos presentan variaciones dependiendo de factores genéticos, ambientales y de la relación entre ambos (Glazier, 1999; Roff y Fairbairn, 2007). La multiplicidad de loci afectando a los caracteres (muchas veces a más de dos de ellos) y las interacciones entre ellos, complejizan aún más estas relaciones (Chippindale et al., 2003; Roff y Fairbairn, 2007; Paaby y Rockman, 2014a). Por otro lado, los loci pleiotrópicos que tengan efectos distintos sobre dos caracteres no causarán necesariamente una correlación significativa entre ellos (Hansen, 2006). También debe considerarse que los efectos sobre los distintos caracteres pueden deberse a distintos polimorfismos dentro del mismo gen pleiotrópico, los que pueden no estar en desequilibrio de ligamiento (Flint y Mackay, 2009). Esto último constituye una forma de modularidad.
Un sistema es modular si se puede dividir en múltiples conjuntos de partes fuertemente interactivas, si bien las partes (módulos) son relativamente autónomas entre sí (Melo et al., 2016). La modularidad impone un límite a la pleiotropía: mediante el surgimiento de módulos a distintos niveles (anatómicos, genéticos, ontogenéticos, etc.) se evita que las alteraciones en un módulo comprometan a otros, dado que la pleiotropía ocurre mayormente de forma intramodular (Wagner y Zhang, 2011). Este fenómeno conocido como pleiotropía modular (Figura 1), puede ser visto como un compromiso entre una integración completa e inflexible y caracteres completamente descoordinados e independientes (Goswami et al., 2014). Al favorecer o restringir ciertas combinaciones de caracteres, los efectos de la pleiotropía determinarán qué trayectorias evolutivas puede seguir una población o especie (Roff y Fairbairn, 2007). Contrariamente, la existencia de módulos relaja estos condicionamientos, promueve el mantenimiento de variabilidad genética y fomenta la divergencia fenotípica (Raff, 1996; Yang, 2001; Melo et al., 2016). Además, la modularidad facilita la aparición de novedades evolutivas pues los módulos pueden ser cooptados para nuevas funciones (Wilkins, 2002; Parter et al., 2008; Shubin et al., 2009; Badyaev, 2011) mediante cambios en la expresión de los genes que constituyen el módulo (Stern, 2000).

Figura 1. Esquema de pleiotropía modular. Los módulos representan grupos de caracteres co-afectados por grupos de loci intramodularmente pleiotrópicos. Las conexiones intermodulares son menos frecuentes que las intramodulares. Adaptado de Wagner y Zhang (2011).
Plasticidad Fenotípica e Interacción Genotipo-Ambiente
La necesidad de los organismos de responder a cambios ambientales es uno de los factores que promueven la evolución. Para un dado carácter, cuando distintos ambientes se asocien a óptimos fenotípicos diferentes, distintas estrategias serán seleccionadas en función de los desafíos que presupone la heterogeneidad ambiental. Una de ellas es la plasticidad fenotípica, es decir la capacidad de un genotipo de producir diferentes fenotipos en respuesta a la variación ambiental (Schlichting y Pigliucci 1998; van Gestel y Weissing 2016). Esta plasticidad tiene base genética y además puede presentar variabilidad, lo que se denomina “interacción genotipo-ambiente” (Figura 2) (Callahan, 2005; Mensch et al., 2008; GTExConsortium, 2015); esta variabilidad puede sustentar una eventual evolución en la plasticidad fenotípica (DeWitt y Scheiner, 2004). La alteración del fenotipo en relación al ambiente favorece una mayor diversificación, teniendo el organismo la capacidad de explotar distintos nichos y soportar un mayor rango de condiciones ambientales (Chevin et al., 2013; Forsman y Wennersten, 2016).
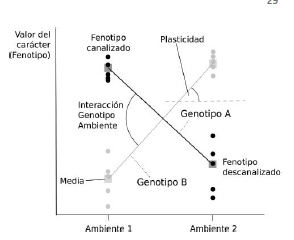
Figura 2. Esquema de los distintos aspectos del fenotipo considerados en este trabajo. La figura representa dos normas de reacción (línea oscura y línea clara) que indican el valor del carácter (fenotipo) frente a dos ambientes para los genotipos A y B. En el caso del genotipo A, la expresión del carácter (fenotipo) en el ambiente 2 muestra un efecto de descanalización, puesto que en este ambiente se observa un cambio tanto en la media fenotípica como en la variabilidad del carácter, que se representa mediante la mayor dispersión de puntos respecto al ambiente 1. Para el genotipo B se observa el patrón opuesto. Ambas normas de reacción son plásticas (plasticidad fenotípica) dado que los fenotipos que se expresan en los dos ambientes son diferentes. La falta de plasticidad está representada por la línea punteada para el genotipo B que revelaría que la expresión fenotípica es independiente del ambiente. Dado que se están analizando dos genotipos es posible establecer si hay interacción genotipo-ambiente mediante una prueba de paralelismo de las normas de reacción.
En un organismo con una alta tasa migratoria, como por ejemplo la que se ha informado en Drosophila melanogaster (Lange et al., 1990), la plasticidad fenotípica y su variación podrían formar parte de una estrategia que permite la supervivencia y adecuación en distintos ambientes sin la necesidad de cambios genéticos (Meyers y Bull, 2002; Bellard et al., 2012). En los últimos años se ha puesto en relevancia la importancia de considerar la plasticidad fenotípica y la interacción genotipo-ambiente en estudios relacionados con la genética y la biología evolutiva: e.g. vinculados a la adaptación (Davidson et al., 2011; Del Pino et al., 2012; Richter et al., 2012; Carreira et al., 2013; Nunes et al., 2014; DeWitt, 2016), resistencia a condiciones de estrés (Fallis et al., 2014; Biddle et al., 2016), capacidad invasora (Richards et al., 2006; Keller y Taylor, 2008) así como en procesos de especiación (Pfennig et al., 2010; Wennersten y Forsman, 2012; Levis y Pfennig, 2016) entre otros aspectos. Por otro lado, un alelo puede tener efectos en el fitness en algunos ambientes, y en otros ser “invisible” a la selección natural (lo que se conoce como “neutralidad condicional”), lo cual favorece la acumulación de variabilidad genética (Paaby y Gibson, 2016).
Epístasis, canalización y variabilidad genética críptica
El papel de la epístasis (Wright, 1931) en la arquitectura genética de los caracteres cuantitativos ha sido controvertido, si bien en los últimos años se incrementó notablemente la importancia que la epístasis detenta para explicar las interacciones moleculares no lineales que sustentan el mapa genotipo-fenotipo (Phyllips, 2008; Mackay, 2014). En este sentido, la generalización de las interacciones epistáticas llevó a cambiar el paradigma previo que consideraba a los genes como miembros de cascadas genéticas o pathways lineales por una idea de redes génicas susceptibles al ambiente, las cuales otorgan a la vez flexibilidad y robustez a la relación entre genotipo y fenotipo (Greenspan, 2001; Sambandan et al., 2006). Dados los efectos del acervo genético en la relación entre genotipo y fenotipo, es evidente que este factor puede tener a nivel poblacional consecuencias importantes desde el punto de vista genético y evolutivo. Ciertamente, podría condicionarse el destino de una mutación en función del acervo (background) genético poblacional y el contexto ambiental (Woods et al., 2011; Chandler et al., 2013; Miton y Tokuriki, 2016), lo cual aumentaría la posibilidad de incompatibilidades entre poblaciones divergentes (Mackay, 2014). Las eventuales interacciones podrían condicionar los fenotipos que se manifiestan y, por lo tanto, las trayectorias evolutivas futuras (Blount et al., 2008; Chandler et al., 2013).
Las interacciones epistáticas pueden ser seleccionadas como un mecanismo de buffer ante condiciones ambientales relativamente estables, provocando un efecto de canalización del fenotipo (Waddington, 1942; Yamamoto et al., 2009) alrededor de un óptimo adaptativo (Figura 2). Como resultado de estas interacciones, es posible determinar dos grupos de variantes resultantes: las que forman parte de la expresión del carácter y que están bajo proceso selectivo y otras cuya manifestación es nula o despreciable respecto a los parámetros adaptativos en un determinado tiempo y espacio. Este segundo tipo de variantes puede acumularse debido a que su efecto permanece oculto de la selección natural, generando una variabilidad genética críptica u oculta (Gibson y Wagner, 2000; Gibson y Dworkin, 2004; Paaby y Rockman, 2014b). Bajo diferentes condiciones genéticas o ambientales que causen modificaciones en la red de interacciones epistáticas, la variabilidad genética críptica podría expresarse y por lo tanto, estar sujeta a los efectos de la selección natural (Flatt, 2005; Gibson, 2009). Durante este proceso, se produce un efecto de descanalización, es decir, aumento de la varianza fenotípica y cambio en la media fenotípica como producto de la “liberación” de variabilidad genética que estaba oculta (Gibson y Wagner, 2000; Dworkin, 2005; Chandler et al., 2013; Lavagnino y Fanara, 2016). El estudio del grado de canalización de distintos fenotipos puede realizarse a través del cálculo del Coeficiente de Variación Ambiental (CVA) que surge de la división entre la varianza del carácter y su media; al ser una variable estandarizada, pueden compararse incluso CVAs para distintos caracteres (Dworkin, 2005; Lavagnino y Fanara, 2016).
La plasticidad en el nivel de canalización podría surgir en combinación con distintos niveles de plasticidad de las medias como estrategia ecológica, especialmente si las funciones de fitness difieren entre ambientes (DeWitt y Scheiner, 2004). Podría pensarse que un caso extremo sería el de las especies invasoras, las cuales enfrentan un importante grado de incertidumbre en cuanto a las nuevas condiciones ambientales. La descanalización y/o la plasticidad en el nivel de canalización sería, en el caso de las especies invasoras, una estrategia que resultaría adaptativa (Carroll, 2008). Los fenómenos de canalización y descanalización son un mecanismo que permitiría explicar la aparente paradoja entre la gran robustez que presentan los sistemas biológicos frente a las perturbaciones de distinto tipo y los numerosos ejemplos de radiaciones adaptativas abruptas con cambios morfológicos drásticos en tiempos evolutivos relativamente cortos (Stanley, 1998; Futuyma, 2013). Estos fenómenos entraban en contradicción con los modelos clásicos, que postulaban una disminución de la variabilidad genética poblacional por selección estabilizadora interrumpidos por períodos de evolución direccional ante cambios de nicho (Paaby y Gibson, 2016). El modelo alternativo propone una sustancial acumulación de variabilidad genética críptica frente a un proceso de selección, generándose un fenotipo canalizado. Ante una modificación en el ambiente o drásticos cambios en el background genético (por ejemplo, por efectos de deriva) puede expresarse esta variabilidad genética escondida, conllevando una rápida aparición de novedades evolutivas (Pigliucci, 2008). Este modelo conecta los procesos macro y microevolutivos sin necesidad de incorporar hipótesis ad-hoc, respetando los principios y mecanismos propuestos por la genética de poblaciones y biología del desarrollo, entre otras áreas del conocimiento (Paaby y Gibson, 2016).
“Evolvabilidad” (del inglés, “evolvability”)
Se define como “evolvabilidad” a la capacidad de evolucionar. Hansen (2006) y Schlichting (2008) la relacionan con la habilidad del sistema genético para producir y mantener variantes genéticas que le permitirán seguir trayectorias evolutivas diversas. La “evolvabilidad” es, por lo tanto, una propiedad emergente del mapeo entre genotipo y fenotipo. El estudio de la arquitectura genética, es decir de todos los elementos genéticos que contribuyen o podrían contribuir en el futuro a la expresión fenotípica (Figura 3), es el único medio para abordar las preguntas relativas a este concepto. Resulta necesario en este punto diferenciar los conceptos de “evolvabilidad” y heredabilidad, que refieren a aspectos distintos de la arquitectura genética de un carácter. La heredabilidad, ya sea en sentido amplio o reducido, refiere a la habilidad de una población de responder a la selección desde la variabilidad genética preexistente (Figura 3). La “evolvabilidad” expresa el potencial evolutivo de una dada arquitectura genética, involucrando tanto la variabilidad genética (visible, críptica y neutra) como las características que favorecen su generación y mantenimiento (Le Rouzic y Carlborg 2008). En consecuencia, todas las propiedades variacionales que mencionamos en las secciones anteriores condicionan la “evolvabilidad”. La base genética de un carácter, definida en principio por las regiones del genoma cuya variabilidad implica un efecto sobre el fenotipo, se organiza en redes de interacciones epistáticas con distinta modularidad que determinan el grado de canalización y plasticidad del fenotipo. Estas propiedades favorecen el mantenimiento de variabilidad genética (operando a distintos niveles de organización de la información genética) que luego será la materia prima del cambio evolutivo (Le Rouzic y Carlborg, 2008; Paaby y Gibson, 2016). De hecho, muchos trabajos demuestran que las respuestas evolutivas rápidas que pueden observarse en la naturaleza dependen no sólo de esta variabilidad genética (en sentido amplio, ver Figura 3) (Barrett y Schluter, 2008; Hayden et al., 2011; Rohner et al., 2013) sino también de la capacidad de responder en forma plástica ante cambios ambientales (Davidson et al., 2011; Lin et al., 2016) y de los procesos de canalización y descanalización que pudieran acontecer (Flatt, 2005; Le Rouzic y Carlborg, 2008; Paaby y Gibson, 2016), involucrando alteraciones en la función y/o expresión de genes a causa de factores epigenéticos, es decir, no relacionados a variaciones en la secuencia del ADN (Richards, 2006; Feinberg, 2007; Zhang et al., 2013; Duncan et al., 2014).

Figura 3. Representación de la arquitectura genética de un carácter. El triángulo representa la base genética que conforma la arquitectura genética de un carácter en una especie o población. Las variantes alélicas ingresan por mutación (parte superior del triángulo); las que presentan un efecto inmediato (beneficioso o perjudicial) sobre el fenotipo se colorean en negro, mientras que las que no tienen un efecto inmediato sobre el fenotipo se encuentran representadas en color claro. Las variantes con efectos beneficiosos deben ingresar y propagarse (debido a su baja frecuencia alélica) a través de la población antes de ser fijadas (vértice del triángulo) por selección natural. La mayor parte de la variabilidad genética no se traduce en variabilidad fenotípica y se considera “neutral”. A nivel fenotípico sólo una parte de la variación es “visible” a los efectos inmediatos de la selección natural, mientras que el resto (denominado “carga genética”) es “críptico” o “neutro”, aunque este límite no es estático. Por efecto de perturbaciones tanto ambientales como genéticas puede producirse un cambio de diversidad genética visible (que está bajo efecto de la selección natural) a críptica y/o neutra y viceversa. Modificado de Le Rouzic y Carlborg (2008).
En resumen, para dilucidar la evolución de la “evolvabilidad” necesariamente se debe caracterizar y comprender los mecanismos y procesos subyacentes a la evolución de la arquitectura genética de caracteres adaptativos (Pigliucci, 2008).
CONCLUSIONES
A diferencia del enfoque clásico de la genética de poblaciones que considera una arquitectura genética invariante, el estudio de las propiedades variacionales y su evolución permite definir relaciones más realistas entre genotipo y fenotipo. Este tipo de estructura puede explicar fenómenos complejos que son relevantes para comprender las dinámicas macro y microevolutivas, y entender los cambios en la capacidad de evolucionar (Hansen, 2006).
La concepción de la arquitectura genética descansa en las intrincadas estructuras que la subyacen y su estudio requiere considerar aspectos del fenotipo que aún son habitualmente relegados o simplificados. Dada la gran complejidad inherente a los fenómenos biológicos, siguen siendo usuales los estudios que involucran líneas mutagenizadas en background genéticos controlados o mutantes de gran efecto que no representan la situación esperable en la naturaleza (Rockman, 2012). Además, la mayoría de los trabajos se enfocan exclusivamente en el estudio de las medias fenotípicas, obviando información sobre otros aspectos del fenotipo, como su varianza o plasticidad (DeWitt y Scheiner, 2004; DeWitt, 2016). Estas metodologías no sólo limitan la posibilidad de inferencia, sino que eliminan la variabilidad que representa el objeto mismo de estudio (Gasch et al., 2016). Sin embargo, en la actualidad es posible abordar estudios más realistas y complejos a partir del desarrollo de métodos de secuenciación de genomas completos y el aumento del poder de cómputo. Sólo considerando a la variabilidad como objeto de estudio y no como mero ruido estadístico se podrán caracterizar las complejas estructuras que moldean el mapa genotipo-fenotipo y su evolución para comprender cabalmente el surgimiento de la diversidad biológica y los procesos inherentes a los mecanismos de adaptación a los diferentes escenarios ecológicos.
AGRADECIMIENTOS
Expresamos nuestro agradecimiento a los Lic. Victoria Ortiz, Ignacio Satorre y Nicolás Flaibani por la lectura y sugerencias de este artículo, así como a los editores del Journal of Basic and Applied Genetics y a dos revisores anónimos por sus correcciones. Agradecemos el financiamiento del FONCyT (proyecto PICT 2012-0640).
BIBLIOGRAFÍA
1. Alberch P. (1991) From genes to phenotype: dynamical systems and evolvability. Genética 84: 5-11. [ Links ]
2. Anholt R.R.H., Dilda C.L., Chang S., Fanara J.J., Kulkarni N.H., Ganguly I., Rollmann S.M., Kamdar K.P., Mackay T.F.C. (2003) The genetic architecture of odor-guided behavior in Drosophila: epistasis and the transcriptome. Nat. Genet. 35: 180-184. [ Links ]
3. Badyaev A.V. (2011) Origin of the fittest: link between emergent variation and evolutionary change as a critical question in evolutionary biology. Proc. R. Soc. B 278: 1921-1929. [ Links ]
4. Barrett R.D., Schluter D. (2008) Adaptation from standing genetic variation. Trends Ecol. Evol. 23: 38-44. [ Links ]
5. Bellard C., Bertelsmeier C., Leadley P., Thuiller W., Courchamp F. (2012) Impacts of climate change on the future of biodiversity. Ecol. Lett. 15: 365-377. [ Links ]
6. Biddle A., Gammon L., Liang X., Costea D.E., Mackenzie I.C. (2016) Phenotypic plasticity determines cancer stem cell therapeutic resistance in oral squamous cell carcinoma. EBioMedicine 4: 138-145. [ Links ]
7. Blount Z.D., Borland C.Z., Lensk R.E. (2008) Historical contingency and the evolution of a key innovation in an experimental population of Escherichia coli. PNAS 105: 7899-7906. [ Links ]
8. Boyle E.A., Li Y.I., Pritchard J.K. (2017) An expanded view of complex traits: from polygenic to omnigenic. Cell 169: 1177-1186. [ Links ]
9. Buckler E.S., Holland J.B., Bradbury P.J., Acharya C.B., Brown P.J., Browne C., Ersoz E., Flint-Garcia S., Garcia A., Glaubitz J.C., Goodman M.M., Harjes C., Guill K., Kroon D.E., Larsson S., Lepak N.K., Li H., Mitchell S.E., Pressoir G., Peiffer J.A., Rosas M.O., Rocheford T.R., Romay M.C., Romero S., Salvo S., Sanchez Villeda H., da Silva H.S., Sun Q., Tian F., Upadyayula N., Ware D., Yates H., Yu J., Zhang Z., Kresovich S., McMullen M.D. (2009) The genetic architecture of maize flowering time. Science 325 (5941): 714-718. [ Links ]
10. Callahan H.S. (2005) Using artificial selection to understand plastic plant phenotypes. Integr. Comp. Biol. 45: 475-485. [ Links ]
11. Carreira V.P., Imberti M., Mensch J., Fanara J.J. (2013) Geneby- temperature interactions and candidate plasticity genes for morphological traits in Drosophila melanogaster. Plos One. 8: e70851. [ Links ]
12. Carreira V.P., Mensch J., Hasson E., Fanara J.J. (2016) Natural genetic variation and candidate genes for morphological traits in Drosophila melanogaster. Plos One 11: e0160069. [ Links ]
13. Carroll S.P. (2008) Facing change: forms and foundations of contemporary adaptation to biotic invasions. Mol. Ecol. 17: 361-372. [ Links ]
14. Chandler C.H., Chari S., Dworkin I. (2013) Does your gene need a background check? How genetic background impacts the analysis of mutations, genes, and evolution. Trends Genet. 29: 358-366. [ Links ]
15. Cheverud J.M. (1996) Developmental integration and the evolution of pleiotropy. Integr. Comp. Biol. 36: 44-50. [ Links ]
16. Chevin L.M., Collins S., Lefèvre F. (2013) Phenotypic plasticity and evolutionary demographic responses to climate change: taking theory out to the field. Funct. Ecol. 27: 967-979. [ Links ]
17. Chippindale A.K., Ngo A.L., Rose M.R. (2003) The devil in the details of life-history evolution: instability and reversal of genetic correlations during selection on Drosophila development. J. Genet. 82: 133-145. [ Links ]
18. Davidson A.M., Jennions M., Nicotra A.B. (2011) Do invasive species show higher phenotypic plasticity than native species and, if so, is it adaptive? A meta-analysis. Ecol. Lett. 14: 419-43. [ Links ]
19. Del Pino F., Salgado E., Godoy-Herrera R. (2012) Plasticity and genotype×environment interactions for locomotion in Drosophila melanogaster larvae. Behav. Genet. 42: 162- 169. [ Links ]
20. DeWitt T.J. (2016) Expanding the phenotypic plasticity paradigm to broader views of traits space and ecological function. Curr. Zool. 62: 463-473. [ Links ]
21. DeWitt T.J., Scheiner S.M. (2004) Phenotypic plasticity: functional and conceptual approaches. Oxford University Press, New York, USA. [ Links ]
22. Duncan E.J., Gluckman P.D., Dearden P.K. (2014) Epigenetics, plasticity, and evolution: How do we link epigenetic change to phenotype? J. Exp. Zool. B. Mol. Dev. Evol. 322 (4): 208-20. [ Links ]
23. Dworkin I. (2005) A study of canalization and developmental stability in the sternopleural bristle system of Drosophila melanogaster. Evolution 59: 1500-1509. [ Links ]
24. Fallis L.C., Fanara J.J., Morgan T.J. (2014) Developmental thermal plasticity among Drosophila melanogaster populations. J. Evol. Biol. 27: 557-564. [ Links ]
25. Fanara J.J., Robinson K.O., Rollmann S.M., Anholt R.R.H., Mackay T.F.C. (2002) Vanaso is a candidate Quantitative trait gene for Drosophila olfactory behavior. Genetics 162: 1321-1328. [ Links ]
26. Feinberg A.P. (2007) Phenotypic plasticity and the epigenetics of human disease. Nature 447: 433-440. [ Links ]
27. Félix M.A. ( 2015) Pervasive robustness in biological systems. Nat. Rev. Genet. 16: 483-496. [ Links ]
28. Flatt T. (2005) The evolutionary genetics of canalization. Quart. Rev. Biol. 80: 287-316. [ Links ]
29. Flint J., Mackay T.F.C. (2009) Genetic architecture of quantitative traits in mice, flies, and humans. Genome Res. 19: 723-733. [ Links ]
30. Forsman A., Wennersten L. (2016) Inter-individual variation promotes ecological success of populations and species: evidence from experimental and comparative studies. Ecography 39: 630-648. [ Links ]
31. Futuyma D.J. (2013) Evolution. Sinauer Associates, Sunderland, MA, USA. [ Links ]
32. Gasch A.P., Payseur B.A., Pool J.E. (2016) The power of natural variation for model organism biology. Trends Genet. 32: 147-154. [ Links ]
33. Gibson G. (2009) Decanalization and the origin of complex disease. Nat. Rev. Genet. 10: 134-140. [ Links ]
34. Gibson G., Dworkin I. (2004) Uncovering cryptic genetic variation. Nat. Rev. Genet. 5: 681-690. [ Links ]
35. Gibson G., Wagner G. (2000) Canalization in evolutionary genetics: a stabilizing theory? Bioessays 22: 372-380. [ Links ]
36. Glazier D.S. (1999) Trade-offs between reproductive and somatic (storage) investments in animals: A comparative test of the Van Noordwijk and De Jong model. Evol. Ecol. 13: 539-555. [ Links ]
37. Goswami A., Smaers J.B., Soligo C., Polly P.D. (2014) The macro evolutionary consequences of phenotypic integration: from development to deep time. Phil. Trans. R. Soc. B 369: 20130254. [ Links ]
38. Greenspan R.J. (2001) The flexible genome. Nat. Rev. Genet. 2: 383-387. [ Links ]
39. GTEx Consortium (2015) The Genotype-Tissue Expression (GTEx) pilot analysis: multitissue gene regulation in humans. Science 348: 648-660. [ Links ]
40. Hansen T.F. (2006) The evolution of genetic architecture. Annu. Rev. Ecol. Evol. Syst. 37: 123-157. [ Links ]
41. Hayden E.J., Ferrada E., Wagner A. (2011) Cryptic genetic variation promotes rapid evolutionary adaptation in an RNA enzyme. Nature 474: 92-95. [ Links ]
42. Houle D., Govindaraju D.R., Omholt S. (2010) Phenomics: the next challenge. Nat. Rev. Genet. 11: 855-866. [ Links ]
43. Huang W., Massouras A., Inoue Y., Peiffer J., Ràmia M., Tarone A.M., Turlapati L., Zichner T., Zhu D., Lyman R.F., Magwire M.M., Blankenburg K., Carbone M.A., Chang K., Ellis L.L., Fernandez S., Han Y., Highnam G., Hjelmen C.E., Jack J.R., Javaid M., Jayaseelan J., Kalra D., Lee S., Lewis L., Munidasa M., Ongeri F., Patel S., Perales L., Perez A., Pu L., Rollmann S.M., Ruth R., Saada N., Warner C., Williams A., Wu Y.Q., Yamamoto A., Zhang Y., Zhu Y., Anholt R.R., Korbel J.O., Mittelman D., Muzny D.M., Gibbs R.A., Barbadilla A., Johnston J.S., Stone E.A., Richards S., Deplancke B., Mackay T.F.C. (2014) Natural variation in genome architecture among 205 Drosophila melanogaster Genetic Reference Panel lines. Genome Res. 24: 1193-1208. [ Links ]
44. Keller S.R., Taylor D.R. (2008) History, chance and adaptation during biological invasion: separating stochastic phenotypic evolution from response to selection. Ecol. Lett. 11: 852-866. [ Links ]
45. Kitano H. ( 2004) Biological robustness. Nat. Rev. Genet. 5: 826-837. [ Links ]
46. Korte A., Farlow A. (2013) The advantages and limitations of trait analysis with GWAS: a review. Plant Methods 9: 29. [ Links ]
47. Lailvaux S.P., Husak J.F. (2014) The life history of wholeorganism performance. Quart. Rev. Biol. 89: 285-318. [ Links ]
48. Lange B.W., Langley C.H., Stephan W. (1990) Molecular evolution of Drosophila metallothionein genes. Genetics 126: 921-932. [ Links ]
49. Lavagnino N., Anholt R.R.H., Fanara J.J. (2008) Variation in genetic architecture of olfactory behaviour among wildderived populations of Drosophila melanogaster. J. Evol. Biol. 21: 988-996. [ Links ]
50. Lavagnino N., Fanara J.J. (2016) Changes across development influence visible and cryptic natural variation of Drosophila melanogaster olfactory response. Evolutionary Biology 43: 96-108. [ Links ]
51. Le Rouzic A., Carlborg O. (2008) Evolutionary potential of hidden genetic variation. Trends Ecol. Evol. 23: 33-37. [ Links ]
52. Levis N.A., Pfennig D.W. (2016) Evaluating “plasticity-first” evolution in nature: key criteria and empirical approaches. Trends Ecol. Evol. 31: 563-574.
53. Lin Y., Chen Z.X., Oliver B., Harbison S.T. (2016) Microenvironmental gene expression plasticity among individual Drosophila melanogaster. G3: Genes, Genomes, Genetics 6: 4197-4210. [ Links ]
54. Mackay T.F.C. (2001) The genetic architecture of quantitative traits. Ann. Rev. Genet. 35: 303-339. [ Links ]
55. Mackay T.F.C. (2014) Epistasis and quantitative traits: using model organisms to study gene-gene interactions. Nat. Rev. Genet. 15: 22-33. [ Links ]
56. Mackay T.F.C., Moore J.H. (2014) Why epistasis is important for tackling complex human disease genetics. Genome Med. 6: 42. [ Links ]
57. Manolio T.A., Collins F.S., Cox N.J., Goldstein D.B., Hindorff L.A., Hunter D.J., McCarthy M.I., Ramos E.M., Cardon L.R., Chakravarti A., Cho J.H., Guttmacher A.E., Kong A., Kruglyak L., Mardis E., Rotimi C.N., Slatkin M., Valle D., Whittemore A.S., Boehnke M., Clark A.G., Eichler E.E., Gibson G., Haines J.L., Mackay T.F.C., McCarroll S.A., Visscher P.M. (2009) Finding the missing heritability of complex diseases. Nature 461: 747-753. [ Links ]
58. Melo D., Porto A., Cheverud J.M., Marroig G. (2016) Modularity: genes, development, and evolution. Annu. Rev. Ecol. Evol. Syst. 47: 463-486. [ Links ]
59. Mensch J., Lavagnino N., Carreira V.P., Massaldi A., Hasson E., Fanara J.J. (2008) Identifying candidate genes affecting developmental time in Drosophila melanogaster: pervasive pleiotropy and gene-by-environment interaction. BMC Dev. Biol. 8: 78. [ Links ]
60. Meyers L.A., Bull J.J. (2002) Fighting change with change: adaptive variation in an uncertain world. Trends Ecol. Evol. 17: 551-557. [ Links ]
61. Miton C.M., Tokuriki N. (2016) How mutational epistasis impairs predictability in protein evolution and design. Protein Sci. 25: 1260-1272. [ Links ]
62. Morozova T.V., Mackay T.F.C., Anholt R.R.H. (2014) Genetics and genomics of alcohol sensitivity. Mol. Genet. Genom. 289: 253-269. [ Links ]
63. Nunes A.L., Orizaola G., Laurila A., Rebelo R. (2014) Rapid evolution of constitutive and inducible defenses against an invasive predator. Ecology 95: 1520-1530. [ Links ]
64. Paaby A., Gibson G. (2016) Cryptic Genetic Variation in Evolutionary Developmental Genetics. Biology 5: 28. [ Links ]
65. Paaby A.B., Rockman M.V. (2014a) The many faces of pleiotropy. Trends Genet. 29: 66-73. [ Links ]
66. Paaby A.B., Rockman M.V. (2014b) Cryptic genetic variation, evolution’s hidden substrate. Nat. Rev. Genet. 15: 247- 258.
67. Parter M., Kashtan N., Alon U. (2008) Facilitated variation: How evolution learns from past environments to generalize to new environments. PLoS Comput. Biol. 4: e1000206. [ Links ]
68. Pfennig D.W., Wund M.A., Snell-Rood E.C., Cruickshank T., Schlichting C.D., Moczek A.P. (2010) Phenotypic plasticity’s impacts on diversification and speciation. Trends Ecol. Evol. 25: 459-467.
69. Phillips P.C. (2008) Epistasis the essential role of gene interactions in the structure and evolution of genetic systems. Nat. Rev. Genet. 9: 855-867. [ Links ]
70. Pigliucci M. (2008) Isevolvability evolvable? Nat. Rev. Genet. 9: 75-82. [ Links ]
71. Pigliucci M. (2010) Genotype-phenotype mapping and the end of the genes as blueprint metaphor. Philos. Trans. R. Soc. Lond. B Biol. Sci. 365: 557-566. [ Links ]
72. Raff R.A. (1996) The shape of life: Genes, development, and the evolution of animal form. University of Chicago, Chicago, IL, USA. [ Links ]
73. Richards E.J. (2006) Inherited epigenetic variation-revisiting soft inheritance. Nat. Rev. Genet. 7: 395-401. [ Links ]
74. Richards C.L., Bossdorf O., Muth N.Z., Gurevitch J., Pigliucci M. (2006) Jack of all trades, master of some? On the role of phenotypic plasticity in plant invasions. Ecol. Lett. 9: 981-993. [ Links ]
75. Richter S., Kipfer T., Wohlgemuth T., Guerrero C.C., Ghazoul J., Moser B. (2012) Phenotypic plasticity facilitates resistance to climate change in a highly variable environment. Oecologia 169: 269-279. [ Links ]
76. Rockman M.V. (2012) The QTN program and the alleles that matter for evolution: All that’s gold does not glitter. Evolution 66: 1-17.
77. Roff D.A., Fairbairn D.J. (2007) The evolution of trade-offs: where are we? J. Evol. Biol. 20: 433-447. [ Links ]
78. Rohner N., Jarosz D.F., Kowalko J.E., Yoshizawa M., Jeffery W.R., Borowsky R.L., Lindquist S., Tabin C.J. (2013) Cryptic variation in morphological evolution: HSP90 as a capacitor for loss of eyes in cavefish. Science 342: 1372- 1375. [ Links ]
79. Sambandan D., Yamamoto A., Fanara J.J., Mackay T.F.C., Anholt R.R.H. (2006) Dynamic genetic interactions determine odor-guided behavior in Drosophila melanogaster. Genetics 174: 1349-1363. [ Links ]
80. Schlichting C.D. (2008) Hidden reaction norms, cryptic genetic variation, and evolvability. Ann. N. Y. Acad. Sci. 1133: 187-203. [ Links ]
81. Schlichting C.D., Pigliucci M. (1998) Phenotypic Evolution: a reaction norm perspective. Sinauer Associates, Sunderland, MA, USA. [ Links ]
82. Schluter D., Nychka D. (1994) Exploring fitness surfaces. Am. Nat. 143: 597-616. [ Links ]
83. Shubin N., Tabin C., Carroll S. (2009) Deep homology and the origins of evolutionary novelty. Nature 457: 818-823. [ Links ]
84. Sinervo B., Svensson E. (2002) Correlational selection and the evolution of genomic architecture. Heredity 89: 329-338. [ Links ]
85. Stanley S.M. (1998) Macroevolution: pattern and process. Johns Hopkins University Press, Baltimore, MD, USA. [ Links ]
86. Stern D.L. (2000) Perspective: evolutionary developmental biology and the problem of variation. Evolution 54: 1079-1091. [ Links ]
87. van Gestel J., Weissing F.J. (2016) Regulatory mechanisms link phenotypic plasticity to evolvability. Sci. Rep. 6: 24524. [ Links ]
88. van Noordwijk A.J., de Jong G. (1986) Acquisition and allocation of resources: Their influence on variation in Life History tactics. Am. Nat. 128: 137-142. [ Links ]
89. Waddington C.H. (1942) Canalization of development and the inheritance of acquired characters. Nature 150: 563-565. [ Links ]
90. Wagner G.P., Zhang J. (2011) The pleiotropic structure of the genotype-phenotype map: the evolvability of complex organisms. Nat. Rev. Genet. 12: 204-213. [ Links ]
91. Wennersten L., Forsman A. (2012) Population-level consequences of polymorphism, plasticity and randomized phenotype switching: A review of predictions. Biol. Rev. 87: 756-767. [ Links ]
92. Wilkins A.S. (2002) The evolution of developmental pathways. Sinauer Associates, Sunderland, MA, USA. [ Links ]
93. Woods R.J., Barrick J.E., Cooper T.F., Shrestha U., Kauth M.R., Lenski R.E. (2011) Second-order selection for evolvability in a large Escherichia coli population. Science 331: 1433-1436. [ Links ]
94. Wright S. (1931) Evolution in mendelian populations. Genetics 16: 97-159. [ Links ]
95. Yamamoto A., Anholt R.R.H., Mackay T.F.C. (2009) Epistatic interactions attenuate mutations affecting startle behaviour in Drosophila melanogaster. Genet. Res. 91: 373-382. [ Links ]
96. Yang A.S. (2001) Modularity, evolvability, and adaptive radiations: A comparison of the hemi- and holometabolous insects. Evol. Dev. 3: 59-72. [ Links ] [ Links ]














