Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Multequina
versión On-line ISSN 1852-7329
Multequina vol.9 no.2 Mendoza 2000
ARTICULO ORIGINAL
Estudios evolutivos y poblacionales en el género Prosopis utilizando marcadores bioquímicos y moleculares
Evolutive and poblational studies in the genus Prosopis using biochemical and molecular markers
Beatriz O. Saidman1 *, Cecilia F. Bessega1#, Laura Ferreyra1, Norma Julio2 y Juan C. Vilardi1 *
1 Departamento de Ecología, Genética y Evolución, Facultad de Ciencias Exactas y Naturales, Universidad de Buenos Aires. 1428. Buenos Aires.
2 Cátedra de Genética de Poblaciones y Evolución. Facultad de Ciencias Exactas, Físicas y Naturales, Universidad Nacional de Córdoba.
* Miembro de la Carrera del Investigador Científico. Consejo Nacional de Investigaciones Científicas y Tecnológicas (CONICET).
#Becaria del Consejo Nacional de Investigaciones Científicas y Tecnológicas (CONICET). Parte de los resultados presentados en el presente trabajo integran las tesis doctorales de C. F. Bessega, L. I. Ferreyra y N. Julio y su participación en este trabajo fue equivalente. ISSN 0327-9375
RESUMEN
La forma en que se distribuye la variabilidad genética dentro y entre poblaciones depende de diferentes factores entre los que se puede mencionar el sistema reproductivo y la estrategia adaptativa. La estructura de las poblaciones, a su vez, establece restricciones a los procesos evolutivos tendientes a la adaptación creciente al ambiente y/o la diferenciación específica.
Los marcadores bioquímicos y moleculares constituyen herramientas para evaluar la proporción de la diversidad genética en entidades de complejidad creciente. Esta información permite hacer inferencias acerca de los mecanismos evolutivos que podrían haber conducido al escenario actual.
En el género Prosopis se realizaron estudios de estructura poblacional y se analizó la diferenciación genética en 15 especies de la Sección Algarobia, Strombocarpa y Monilicarpa utilizando las técnicas de isoenzimas, RAPD, RFLP y Secuenciación de ADN.
En siete especies de la sección Algarobia se analizó, a partir de datos provenientes de electroforesis de isoenzimas, el sistema de apareamiento, pudiendo demostrarse que aunque éstas son mayormente exógamas, pueden presentar hasta un 28% de autofecundación, con un promedio del 19%.
La variabilidad genética en especies de Algarobia es alta, pero tiende a ocurrir dentro de las poblaciones. Por esta razón con muestras de pocas poblaciones se cubre la mayor parte de la variación genética detectada por isoenzimas y por la técnica de RAPD.
Con excepción de P. kuntzei, las especies de Algarobia son muy afines entre sí independientemente de su nivel de ploidía y distribución geográfica y las mismas no se agrupan de acuerdo con las series.
En las especies estudiadas de la sección Strombocarpa la variabilidad es mucho menor que en Algarobia. Estas especies se diferencian mucho entre sí y se separan claramente de las de Algarobia.
Finalmente P. argentina (secc. Monilicarpa) es la especie más diferenciada de las estudiadas hasta el presente.
La hibridación natural es un hecho frecuente entre especies de la sección Algarobia. La técnica de RAPD ha permitido obtener bandas marcadoras de especie que podrían utilizarse para inferir los progenitores putativos de híbridos naturales.
Finalmente los resultados obtenidos por las técnicas de RFLP de mitocondrias y cloroplastos, así como la de secuenciación de un fragmento de este último plástido han permitido estudios cladísticos que confirman la alta afinidad entre especies de Algarobia y brindan evidencias de que las mismas tendrían un origen común.
Palabras clave: Isoenzimas; RAPD; Secuenciación de ADN; Estructura Poblacional.
SUMMARY
The way in which genetic variability is distributed within and among populations depends on different factors, outstanding among these are the reproductive system and the adaptive strategy. Population structure, in turn, imposses restrictions to those evolutionary processes tending to a growing adaptation to the environment and/or specific differentiation.
Studies on population structure were made for the Prosopis genus and the genetic differentiation was analyzed for 15 species of Algarobia, Strombocarpa and Monilicarpa Sections using isoenzyme techniques, RAPD, RFLP and DNA sequenciation. The linkage system was analyzed in 7 species of the Algarobia Section from data obtained from isoenzyme electrophoresis, which showed that although these species are mostly exogamous they can reach up to 28% of self-fecundation, with an average of 19%. This behaviour and the limited capacity for pollen and seed dispersal determine an excess of homozygotes in the populations.
Except for P. kuntzei, Algarobia species are closely related, independently of their ploidy level and geographical distribution, and are not grouped according to series.
Species in the Strombocarpa Section exhibit much lower variability than those in Algarobia.
P. argentina (Monilicarpa Section) is the most distinctly differentiated of the species studied to date. Natural hybridation is frequent among species of the Algarobia Section. This phenomenon and the great similarity among these species make their recognition difficult.
The results obtained using RFLP techniques in mitocondria and cloroplasts confirm the great affinity among Algarobia species, and provide evidence that they would have a common origin.
Key words: Isoenzymes; RADP; DNA sequenciation; Population structure.
INTRODUCCIÓN
El género Prosopis ha despertado gran interés en los últimos años dado que constituye un recurso promisorio para reforestación y recuperación de suelos empobrecidos en regiones áridas y semiáridas. Desde el punto de vista ecológico estas especies cumplen importantes funciones, que incluyen fijación de nitrógeno, control de la erosión del suelo y estabilización de médanos y presentan tolerancia a la sequía y a suelos salinos. Desde el punto de vista económico sus usos son muy diversos: alimentos y forraje, combustible, leña, elaboración de alcohol, fabricación de muebles y producción de taninos y exhiben una excelente adaptación a sistemas agroforestales y silvopastoriles.
La provincia biogeográfica chaqueña en la Argentina constituye un importante centro de polimorfismo de este grupo (Burkart, 1976). De las 44 especies que comprende el género, unas 28 pueden encontrarse en este país, ocupando diferentes provincias biogeográficas pertenecientes al dominio Chaqueño (para una revisión de las regiones biogeográficas en Sudamérica ver Cabrera y Willink, 1980). La explotación racional de especies nativas requiere de un amplio conocimiento de la taxonomía, biología, ecología y genética. A partir de los estudios pioneros sobre taxonomía (ver Burkart, 1976), se realizaron estudios sobre las relaciones entre especies del grupo basados en diversas técnicas, tales como cromatografía, citogenética, electroforesis de proteínas seminales e isoenzimas, que fueron revisados por Hunziker et al. (1986). Durante los últimos años los avances de la biología molecular permitieron incorporar nuevos marcadores útiles en estudios poblacionales y filogenéticos para resolver problemas relacionados con la estructura genética, sistema de fecundación, identifica ción de híbridos naturales y relaciones entre las diferentes especies del género (Saidman, 1985, 1993; Saidman y Vilardi, 1987, 1993, Bessega et al., 2000a, 2000b, 2000c).
MATERIAL Y MÉTODO
Estudios alozímicos
La mayor parte de los estudios acerca de la variación genética en especies de Prosopis se basan en electroforesis de isoenzimas. Hasta el presente se estudiaron isoenzimáticamente 19 especies, 13 pertenecientes a la sección Algarobia, 5 de Strombocarpa y P. argentina de la sección Monilicarpa (Tabla 1).
Tabla 1. Especies y poblaciones de Prosopis analizadas hasta el presente mediante isoenzimas, RAPD, RFLP o secuenciación
Table 1. Species and populations of Prosopis analyzed up to the present time by means of insoenzymes, RAPD, RFLP or sequenciation

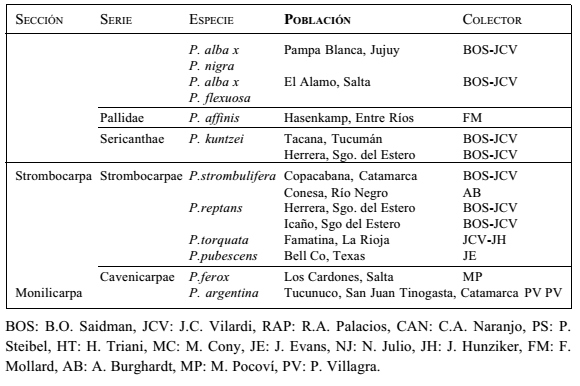
Dentro de la sección Algarobia se analizaron además poblaciones que comprendían híbridos naturales interespecíficos. Esta técnica permitió estimar parámetros para cuantificar (a) la variabilidad genética dentro de las poblaciones, (b) la estructura genética, (c) el sistema de fecundación y (d) la diferenciación genética entre poblaciones y especies.
La variabilidad fue cuantificada por medio de diferentes índices incluyendo la proporción de loci polimórficos (P), la heterocigocidad media (H), el número efectivo de alelos (ne), el número total de alelos (A). Los resultados obtenidos hasta el presente muestran diferencias importantes entre las especies de las secciones Algarobia y Strombocarpa en el grado de variabilidad dentro de las poblaciones. En la primera la variabilidad fue mucho mayor (H= 0.21±0.01, P= 51.5±1.9, ne= 1.30, A= 1.81) que en las especies de Strombocarpa (H= 0.07±0.01, P= 15.3±2.3, ne= 1.06, A= 1.15).
La frecuente ocurrencia de híbridos naturales interespecíficos e introgresantes entre especies de Algarobia se consideró inicialmente como una posible explicación de la mayor variabilidad genética en esta sección. Por este motivo se realizaron estudios de estructura poblacional y se estimaron los coeficientes FST jerárquicos y no jerárquicos y el índice de fijación FIS (Wright, 1951, 1978). La hipótesis a evaluar era que si las barreras reproductivas entre las especies eran débiles, las especies simpátricas podrían mantener un alto flujo génico, reduciendo la diferenciación entre ellas y aumentando su variabilidad. Los resultados de estos análisis indicaron que la diferenciación entre poblaciones coespecíficas, cuantificada por medio del coeficiente FST, era menor que entre poblaciones de distintas especies que cohabitaban en una misma localidad. La interpretación de estos resultados es que a pesar de la capacidad de hibridar en simpatría, el flujo génico efectivo entre distintas especies no sería significativo (Saidman et al., 1998a; Ferreyra, 2001). Algunos loci, como Adh-1, Adh-2, Idh-1 e Idh- 2 serían los que más contribuyen a la diferenciación entre las especies.
El índice F IS indicó que en la mayoría de las especies estudiadas de Algarobia había un significativo exceso de homocigotas con respecto a lo esperado para poblaciones panmícticas (FIS > 0) (Montoya et al., 1994; Julio, 2000; Ferreyra, 2001; Bessega, 1997; 2001; Bessega et al., 2000c).
Este resultado era contradictorio con ciertas evidencias que sugerían que estas especies serían de fecundación cruzada obligada (Burkart, 1976; Simpson, 1977; Simpson et al., 1977). Por este motivo se realizó un estudio de los parámetros del sistema de fecundación en poblaciones naturales de siete especies por medio de los coeficientes de exocruza t (Ritland y Jain, 1981). El método de análisis fue el de Brown y Allard (1970) utilizando el programa MLTR (versión mejorada del programa MLT; MLTR, Ritland) Éste se basó en la segregación de variantes alozímicas, agrupando las muestras poblacionales en familias constituidas por semillas provenientes de una misma planta madre (medio hermanas) (Bessega et al., 2000c). Los coeficientes de exocruza a nivel multilocus (t m ) y de loci individuales (t s ) indicaron que la proporción de autofecundación variaba de 0.72 a 1.0, lo que implica que, aunque las especies son principalmente exógamas, puede ocurrir hasta un 28% de autofecundación, con un promedio del 15%. Esta tasa de autofecundación podría explicar valores de FIS promedio de aproximadamente 0.08, mientras que los valores estimados de F IS en las mismas poblaciones oscilaron entre 0.07 y 0.48, con un promedio de 0.27 (Bessega et al., 2000b). La diferencia entre el valor de FIS esperado en función de la tasa estimada de exocruza y los valores observados se interpretó como el producto de subestructuración de las poblaciones (Bessega et al., 2000c). Dicha subestructuración podría deberse a que la dispersión del polen en especies de Algarobia sería limitada (Genisse et al., 1990; Bessega et al. , 2000c), lo que favorecería el cruzamiento entre árboles vecinos.
Al estimar los coeficientes F IS en las plantas madres, Bessega et al. (2000c) observaron que éstos eran menores que en las semillas y propusieron que esto podría deberse a selección en favor de los heterocigotas.
Una información importante para planes de conservación de germoplasma y explotación racional de especies promisorias se relaciona con la distribución de la variación genética entre y dentro de regiones y poblaciones. Esta información permite tomar decisiones acerca de la mejor estrategia de muestreo para bancos de germoplasma y de selección de caracteres benéficos heredables. P. chilensis es una de las especies más promisorias para programas de reforestación (Burkart, 1976). Su alta variabilidad morfológica aparentemente asociada a la diversidad climática y edáfica, así como su alta productividad de frutos (Roig, 1993; Cony, 1993; Karlin y Diaz, 1998a,b) alientan el desarrollo de programas de selección. En esta especie se realizó un análisis jerárquico de la distribución de la variabilidad a partir de un muestreo que involucraba 12 poblaciones pertenecientes a 3 regiones geográficas: Provincia Biogeográfica Chaqueña (Córdoba y San Luis), Provincia Biogeográfica del Monte (La Rioja, Catamarca y San Juan) y Valle Central de Chile (Julio, 2000). Los resultados indicaron que la mayoría de la variación (˜87% de la diversidad total) ocurre dentro de las poblaciones. La diferenciación entre las poblaciones dentro de cada región fue baja (˜13%), mientras que no hay divergencia entre regiones.
El mismo método estadístico permitió evaluar cómo se distribuye la variación genética entre y dentro de especies de la sección Algarobia. Para ello se consideraron dos niveles jerárquicos: especie y población. Bessega et al. (2000b) estudiando dos especies argentinas (P. flexuosa y P. ruscifolia) y una especie norteamericana (P. glandulosa) y Ferreyra (2001) estudiando seis especies argentinas obtuvieron conclusiones similares. Más del 60% de la diversidad total ocurre dentro de las poblaciones, la menor diversidad (13%) se verifica entre poblaciones de la misma especie y la diferenciación entre especies es intermedia (16-24%) entre las anteriores. Curiosamente, la inclusión de especies muy aisladas geográficamente en el estudio realizado por Bessega et al. (2000b) no se ve acompañado de un incremento de la diferenciación genética entre especies. En otros estudios (Saidman et al. , 1997) se observó también que poblaciones tetraploides de P. juliflora de Colombia mostraban alta similitud genética con especies diploides argentinas (P. caldenia y P. ruscifolia). Estos resultados son una fuerte evidencia en contra de la hibridación como explicación de la alta similitud genética observada entre especies de Algarobia. Por lo tanto, aunque no puede descartarse que la hibridación haya jugado un papel en las primeras etapas de la diversificación específica de este grupo, es muy probable que la alta variabilidad compartida y la escasa diferenciación entre estas especies se deba a que el efecto fundador haya tenido poca importancia durante la especiación, como consecuencia de tasas de crecimiento poblacional altas. En contraste, la evolución de rasgos morfológicos podría haber sido rápida como respuesta a la adaptación a distintos nichos ecológicos, promoviendo claras diferencias entre las especies.
A diferencia de lo observado en Algarobia, las especies estudiadas de Strombocarpa están muy diferenciadas genéticamente entre sí. Sólo dos especies de esta sección, P. reptans y P. strombulifera, son muy afines entre sí, y, por una serie de criterios independientes, se sugiere que podrían constituir variedades o subespecies más que especies (Hunziker et al., 1986; Saidman et al. , 1996). Las especies estudiadas de Strombocarpa y la única especie de Monilicarpa, P. argentina, se diferencian claramente de las de Algarobia.
En conclusión, los estudios alozímicos han contribuido a la interpretación de las relaciones entre las especies de este género y a la caracterización de la estructura poblacional. Sin embargo, estos marcadores no aportan loci que puedan utilizarse para identificar taxonómicamente las especies de la sección Algarobia analizadas hasta el momento.
Estudios basados en marcadores moleculares
Dado que muchas de las especies económicamente más importantes pertenecientes a la sección Algarobia hibridan naturalmente, sería fundamental contar con una herramienta que permitiera identificar los posibles progenitores de dichos híbridos. Estos marcadores serían útiles también para evaluar los resultados de cruzamientos interespecíficos controlados con el objeto de combinar propiedades de interés agroforestal de distintas especies.
Una técnica relativamente sencilla que permite analizar simultáneamente muchos loci se basa en el análisis de polimorfismos de fragmentos de ADN amplificados al azar (RAPD). Utilizando este método se analizaron poblaciones naturales de P. flexuosa, P. alba, P. nigra, P. ruscifolia y P. vinalillo e híbridos naturales determinados morfológicamente como P. alba x P. flexuosa y P. alba x P. nigra (Tabla 1 y Saidman et al., 1998a).
El análisis de los patrones de bandas obtenidos mediante esta técnica permitió por primera vez obtener marcadores moleculares que diferencian P. alba, P ruscifolia y P. nigra. (Ferreyra et al., inédito) Asimismo, en las poblaciones híbridas (Saidman et al., 1998a) se pudo reconocer los posibles progenitores. Una de ellas (P. alba x P. flexuosa) mostró patrones correspondientes a lo esperado por introgresión hacia uno de los progenitores (P. flexuosa). Sin embargo la otra población (P. alba x P. nigra) mostró patrones complejos donde estaban presentes bandas adicionales, no presentes en ninguno de sus progenitores putativos.
Los resultados obtenidos son también importantes para interpretar el posible origen de P. vinalillo. Según Burkart (1976) esta especie se habría originado como un híbrido entre P. alba y P. ruscifolia. Los marcadores RAPD obtenidos hasta el momento indican que esta especie tiene bandas de P. ruscifolia y de P. nigra, pero en ningún caso se observaron bandas de P. alba (Ferreyra et al., inédito), de modo que los resultados presentes no apoyarían la hipótesis de Burkart.
Los resultados obtenidos hasta el momento son promisorios y permiten suponer que el análisis de un mayor número de cebadores ("primers") permitirá la caracterización inequívoca de éstas y otras especies de la sección Algarobia.
Los RAPD fueron utilizados también para estudios de estructura poblacional y diferenciación genética entre poblaciones. Los resultados obtenidos por RAPD en tres especies argentinas (P. flexuosa, P. nigra y P. alba) y dos norteamericanas (P. glandulosa y P. velutina) se compararon con los obtenidos por estudios alozímicos (Bessega et al., 2000a). Los RAPD y las isoenzimas fueron coincidentes en mostrar alta variabilidad genética. Cuando se consideraron todas las poblaciones ambos marcadores produjeron estimas altas de la diferenciación genética medida a través del coeficiente F ST, lo cual sugeriría que el flujo génico sería escaso. Al analizar solamente poblaciones de P. glandulosa, los datos de isoenzimas, de acuerdo con lo esperado para poblaciones coespecíficas, mostraron una reducción de la estima del FST, compatible con un flujo génico mayor. Sin embargo, este efecto no se evidenció para los marcadores RAPD.
Estos marcadores, mostraron valores más altos de diferenciación genética entre las poblaciones coespecíficas de P. glandulosa que los obtenidos a partir de las isoenzimas y podrían resultar más útiles para realizar estimas de la variabilidad dentro de estas especies. Por otra parte, las isoenzimas fueron más útiles para diferenciar P. glandulosa de P. velutina, en base a los sistemas GOT y PRX.
Un método objetivo que permite estimar las afinidades entre especies y que no se ve afectado por el ambiente o problemas de desarrollo se basa en la comparación de fragmentos de ADN obtenidos por digestión con endonucleasas de restricción (RFLP) y/o por reacción en cadena de la polimerasa (PCR) y secuenciación de segmentos de ADN específicos. Mediante este tipo de técnicas es posible además comparar genes nucleares y citoplasmáticos, que poseen diferentes sistemas de transmisión, lo que permite realizar inferencias con respecto a los mecanismos evolutivos involucrados.
Se aplicaron las técnicas de RFLP de ADN mitocondrial y ribosómico y PCR de un espaciador no transcripto de ADNr en las especies P. ruscifolia, P. flexuosa, P. chilensis, P. kuntzei (Secc. Algarobia), P. ferox, P. reptans y P. pubescens (Secc. Strombocarpa) y en híbridos P. chilensis x P. flexuosa. Para cada tipo de genoma se realizaron análisis independientes de agrupamiento a partir de las similitudes entre especies. Los resultados de ambos son coherentes con la clasificación morfológica, salvo por el hecho de que P. pubescens aparece aislada y no se agrupa con otras especies de su misma serie. Los datos de ADNr, en concordancia con los estudios alozímicos, agrupan cercanamente las especies de Algarobia, mientras que las de Strombocarpa están mucho más dispersas. Los resultados del ADNmt, a diferencia de los isoenzimáticos, son consistentes con la morfología por cuanto separa las especies de distintas series (Saidman et al., 1998b).
Mediante la técnica de RFLP se realizó un análisis cladístico basado en el ADN del cloroplasto (ADNcp) en una especie de Strombocarpa (P. reptans) y 10 especies de Algarobia (Serie Ruscifoliae: P. ruscifolia y P. vinalillo; Ser. Chilenses: P. alba, P. glandulosa, P. caldenia, P. flexuosa, P. alpataco y P. nigra; Ser. Pallidae: P. affinis; Ser. Sericanthae: P. kuntzei). Acacia aromo se utilizó como grupo externo. Se analizaron los fragmentos obtenidos por digestión con 10 enzimas de restricción utilizando como sondas dos regiones de ADNcp de Nicotiana tabacum. La primera región estudiada corresponde a 17.4 kb. de la región SSC del cloroplasto mientras que la otra región corresponde a 10.3 kb. del IR.
No se observó variación intraespecífica ni diferenciación entre los patrones de restricción de las especies de la Sección Algarobia, excepto P. kuntzei que presentó patrones característicos diferenciales con respecto a las otras especies (Bessega, 2001; Bessega et al. , inédito).
Se analizó además la secuencia nucleotídica de un fragmento 1500 pb de ADNcp. El fragmento se obtuvo por la amplificación por PCR, utilizando cebadores universales de la región espaciadora intergénica trnT-trnD. Los resultados permitieron construir un único árbol de máxima parsimonia que mostró que P. reptans y P. kuntzei se separan tempranamente entre sí y del resto de las especies estudiadas. Las especies de la sección Algarobia no mostraron, a diferencia de los estudios de ADNmt una topología en el cladograma acorde con lo esperado a partir de las series propuestas en base a la morfología (Bessega, 2001; Bessega et al. , inédito).
Visto en su conjunto, las evidencias bioquímicas y moleculares coinciden en mostrar una gran afinidad entre las especies de Algarobia, con la excepción de P. kuntzei. Las distancias genéticas entre las especies analizadas de las series Ruscifoliae, Chilenses y Pallidae, en algunos casos son tan bajas como las registradas en otros géneros para poblaciones pertenecientes a subespecies o razas geográficas de la misma especie. Estos resultados sugieren que las mismas podrían estar en una etapa temprana de especiación. P. reptans (Strombocarpa) y P. kuntzei (serie Sericanthae) se habrían separado tempranamente del resto de las especies analizadas hasta el presente (Saidman y Vilardi, 1987; Pocoví, 1992; Bessega, 2001; Bessega et al., inédito).
AGRADECIMIENTOS
El material argentino analizado fue determinado gentilmente por RA. Palacios (UBA), el material de Norteamérica fue cedido por el Dr. CJ De Loach (Grassland Research Station- USDA/ARS), las poblaciones de P. argentina y parte de P. flexuosa fueron cedidas por P. Villagra y M. Cony (IADIZA, Argentina). Este trabajo fue realizado gracias al apoyo financiero de la Agencia Nacional de Promoción Científica y Tecnológica (PICT 6628), Consejo Nacional de Investigaciones Científicas y Técnicas (PIP 0722/98) y Universidad de Buenos Aires (PIP UBACYT X086).
BIBLIOGRAFÍA
1. BESSEGA, C., 1997. Estudios isoenzimáticos en especies Americanas del Género Prosopis (Leguminosae). Tesis de Licenciatura. FCEyN. Universidad de Buenos Aires. [ Links ]
2. BESSEGA C., 2001. Estructura poblacional y relaciones filogenéticas (distancia y parsimonia) en especies del género Prosopis(Leguminosae). Tesis de Doctorado. FCEyN. Universidad de Buenos Aires. [ Links ]
3. BESSEGA, C., B.O. SAIDMAN & J. C. VILARDI, 2000a. Isozyme and RAPD Studies in Prosopis glandulosa and P. velutina (Leguminosae, Mimosoideae). Genetics and Molecular Biology 23 (3):1-5. [ Links ]
4. BESSEGA, C., L. FERREYRA, B. O. SAIDMAN & J. C. VILARDI, 2000b. Unexpected low genetic differentiation among allopatric species of section Algarobia of Prosopis (Leguminosae). Genetica 109:255-266. [ Links ]
5. BESSEGA, C., L. I. FERREYRA, N. JULIO, S. MONTOYA, B. O. SAIDMAN & J. C. VILARDI, 2000c. Mating system parameters in species of genus Prosopis (Leguminosae). Hereditas 132:19-27. [ Links ]
6. BESSEGA, C., M. CLEMENTE, J. C. VILARDI & B. O. SAIDMAN. Phylogenetic relationships among species of Genus Prosopis (Fabaceae) based on cpDNA variation. Inédito [ Links ]
7. BROWN, A. H. D. & R. W. ALLARD, 1970. Estimation of the mating system in openpollinated maize populations using isozyme polymorphisms. Genetics 66:133-145. [ Links ]
8. BURKART A., 1976. A monograph of the genus Prosopis (Leguminosae subfam. Mimosoidae). Journal Arnold Arboretum 57:219-525 [ Links ]
9. CABRERA, A. L. & A. WILLINK, 1980. Biogeografía de América Latina. Monografías Científicas de la OEA. vol. 13. Buenos Aires, Argentina. [ Links ]
10. CONY, M., 1993. Reforestación racional de zonas áridas y semiáridas con árboles de múltiples propósitos. Interciencia 20:249-253. [ Links ]
11. FERREYRA, L., 2001. Estudio de la variabilidad y la diferenciación genética por medio de técnicas de isoenzimas y RAPD en poblaciones naturales de especies e híbridos del Género Prosopis (Leguminosae, Mimosoidae). Tesis de Doctorado. FCEyN. Universidad de Buenos Aires. [ Links ]
12. FERREYRA, L. I., C. BESSEGA, B. O. SAIDMAN & J. C. VILARDI. First report on RAPD patterns able to differentiate some species of section Algarobia (Prosopis, Leguminosae). Inédito. [ Links ]
13. GENISSE, J., R. A. PALACIOS, P. S. HOC, R. CARRIZO, L. MOFFAT, M. P. MOM, M. A. AGULLO, P. PICCA & S. TORREGOSA, 1990. Observaciones sobre la biología floral de Prosopis (Leguminosae, Mimosoidae). II Fases florales y visitantes en el distrito Chaqueño Serrano. Darwiniana 30:71-85. [ Links ]
14. HUNZIKER, J. H., C. A. NARANJO, R. A. PALACIOS, L. POGGIO & B. O. SAIDMAN, 1986. Studies on the taxonomy, genetic variation and biochemistry of Argentine species of Prosopis. Forest Ecology and Management 16 (1-4):301-315. [ Links ]
15. JULIO, N. B., 2000. Estudios alozímicos sobre variabilidad, estructura y diferenciación genética en Prosopis chilensis (Leguminosae, Mimosoideae) y especies relacionadas. Tesis de Doctorado. Cs. Biológicas, Universidad Nacional de Córdoba. [ Links ]
16. KARLIN, U. & R. DIAZ, 1988a. Otros posibles usos. En: Prosopis en la Argentina.Documento preliminar elaborado para el Primer Taller Internacional sobre recursos genéticos y Conservación de Germoplasma de Prosopis. Pp: 237. FAO, FCA-UNC y FCEyNUBA.
17. KARLIN, U. & R. DIAZ, 1988b. Sistemas agroforestales. En: Prosopis en la Argentina. Documento preliminar elaborado para el Primer Taller Internacional sobre recursos genéticos y Conservación de Germoplasma de Prosopis. Pp: 235. FAO, FCA-UNC y FCEyN-UBA.
18. MONTOYA, S., B. O. SAIDMAN, J. C. VILARDI & C. BESSEGA, 1994. Diferenciación y flujo genético entre especies de la Sección Algarobia, Género Prosopis (Leguminosae). XXIV Congreso de la Sociedad Argentina de Genética: 17. [ Links ]
19. POCOVÍ, M. I., 1992. Estudios isoenzimáticos en especies de Prosopis (Leguminosae, subf. Mimosoidae). Tesis de Licenciatura. Facultad de Ciencias Naturales. Universidad Nacional de Salta. [ Links ]
20. RITLAND, K. & S. JAIN, 1981. A model for the estimation of outcrossing rate and gene frequencies using n independent loci. Heredity 47:35-52. [ Links ]
21. ROIG, F. A., 1993. Aportes a la Etnobotánica del Género Prosopis. En: Contribuciones Mendocinas a la quinta Reunión Regional para América Latina y el Caribe de la Red de Forestación del CIID. Pp: 99-121. Unidades de Botánica y Fisiología vegetal. IADIZA. [ Links ]
22. SAIDMAN, B. O., 1985. Estudio de la variación alozímica en el género Prosopis. Tesis Doctoral, FCEyN. Universidad de Buenos Aires. [ Links ]
23. SAIDMAN, B. O., 1993. Las isoenzimas en el estudio de la variación genética y las afinidades entre especies de Prosopis. Bol. Genét. Inst. Fitotéc. Castelar 16:25-37. [ Links ]
24. SAIDMAN, B. O. & J. C. VILARDI., 1987. Analysis of the genetic similarities among seven species of Prosopis (Leguminosae: Mimosoideae). Theoretical Applied Genetics 75:109-116. [ Links ]
25. SAIDMAN, B. O. & J. C. VILARDI, 1993. Genetic variability and germplasm conservation in the genus Prosopis. En: Puri, S. (ed).Nursery Technology of Forest Tree Species of Arid and Semiarid Regions. Pp. 187-198. Winrock-oxford & IBH Publishing Co. PVT. LTD., New Delhi, Bombay, Calcuta. [ Links ]
26. SAIDMAN, B. O., J. C. VILARDI, M. I. POCOVÍ & N. ACRECHE, 1996. Isozyme studies in Argentine species of the Section Strombocarpa, Genus Prosopis (Leguminosae). J. Genetics 75:139-149. [ Links ]
27. SAIDMAN, B. O., J. C. VILARDI, S. MONTOYA & L. POGGIO, 1997. Genetic Variability and Ploidy level in species of Prosopis (Leguminosae). Bol. Soc. Argent Bot. 32 (3-4): 217-225. [ Links ]
28. SAIDMAN, B. O., C. BESSEGA, L. FERREYRA & J. C. VILARDI, 1998a. Random amplified polymorphic DNA (RAPDS) variation in hybrid swarms and pure populations of genus Prosopis. In: Bruns, S., S. Mantell, C. Tragärdh & A. V. Viana (eds.) Recent Advances in Biotechnology for Tree Conservation and Management. Pp: 122-134. International Foundation for Sciences. Stockholm. ISBN: 91 85798 460. [ Links ]
> 29. SAIDMAN, B. O., J. C. VILARDI, S. MONTOYA, M. J. DIEGUEZ & H. E. HOPP, 1998b. Molecular markers: a tool for the understanding of the relationships among species of Prosopis (Leguminosae, mimosoidae). In: Puri, S. (Ed.). Tree Improvement: Applied Research and Technology Transfer. Science Publishers Inc. U. S. A. Chapter 21:311- 324. [ Links ]
30. SIMPSON, B.B., 1977. Breeding system of dominant perennial plants of two disjuncts warm desert ecosystems. Oecologia 27:203-226. [ Links ]
31. SIMPSON, B. B., J. L. NEFF & A. R. MOLDENKE, 1977. Prosopis flowers as a resource. En: BB Simpson (ed). Mesquite: Its biology in two desert ecosystems, US/IBP synthesis Series Ch 5:84-107. Dowden Hutchinson and Ross, Stroudsburg. [ Links ]
32. WRIGHT, S., 1951. The genetical structure of populations. Annals Eugenics 15:323-354. [ Links ]
33. WRIGHT, S., 1978. Evolution and the genetics of Populations, Vol 4. In: Variability within and among natural populations. University of Chicago Press, Chicago. [ Links ]
Recibido: 01/12/2000
Aceptado: 14/12/2000














