Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista de la Facultad de Ciencias Agrarias. Universidad Nacional de Cuyo
On-line version ISSN 1853-8665
Rev. Fac. Cienc. Agrar., Univ. Nac. Cuyo vol.43 no.2 Mendoza July/Dec. 2011
ARTÍCULOS ORIGINALES
Ligamiento genético entre variables asociadas a calidad del fruto de tomate y polipéptidos expresados en dos estados de madurez
Genetic linkage among tomato fruit quality traits and polypeptides expressed at two ripening stages
Mariana Gallo 1, 4, Gustavo Rubén Rodríguez 2, 4, Roxana Zorzoli 3, 4, Guillermo Raúl Pratta 2, 4
1 Licenciatura en Biotecnología. Universidad Nacional de Rosario.
2 Consejo Nacional de Investigaciones Científicas y Técnicas (CONICET).
3Consejo de Investigaciones de la Universidad Nacional de Rosario.
4Cátedra de Genética. Facultad de Ciencias Agrarias. Universidad Nacional de Rosario. C. C. 14. Zavalla. Argentina. S2125ZAA. gpratta@unr.edu.ar
Recepción: 26/05/2011
Aceptación: 28/10/2011
RESUMEN
Los perfiles de polipéptidos proveen información sobre la constitución genética de un individuo y su expresión, y son útiles como marcadores moleculares. El objetivo del trabajo fue detectar ligamiento entre los perfiles de polipéptidos del pericarpio en dos estados de madurez y caracteres cuantitativos y de calidad de los frutos, analizando 21 genotipos de tomate. Se obtuvieron los perfiles polipéptidos en los estados verde y rojo maduro de frutos de 18 líneas endocriadas recombinantes (RILs, recombinant inbred lines), derivadas de un cruzamiento interespecífico entre el cultivar Caimanta de S. lycopersicum y la entrada LA722 de S. pimpinellifolium, que se incluyeron como testigos experimentales junto a su F1. En estos 21 genotipos se evaluaron también vida poscosecha, peso, firmeza, porcentaje de reflectancia, índice cromático, forma, pH, acidez titulable, contenido de sólidos solubles, espesor de pericarpio y número de lóculos de los frutos. Los perfiles mostraron polimorfismo entre los estados de madurez dentro de un mismo genotipo y entre genotipos para un mismo estado de madurez. Algunos polipéptidos segregaron de forma mendeliana (1:1) y, por análisis de un único punto, mostraron ligamiento con caracteres de calidad del fruto. Se detectaron loci de caracteres cuantitativos (QTLs, quantitative trait loci) asociados a número de lóculos, peso, pH, firmeza y vida poscosecha de los frutos.
Palabras clave: Solanum sección Lycopersicon; Recursos fitogenéticos; Mejoramiento vegetal; Marcadores moleculares; QTLs (loci de caracteres cuantitativos).
ABSTRACT
Polypeptide profiles are informative about the individual gene constitution and expression, and become useful tools as molecular markers. The objective of this work was to detect genetic linkage among polypeptide profiles of the pericarp at two ripening stages and fruit quality traits in 21 tomato genotypes. Polypeptide profiles were obtained from mature green and red ripe fruits of 18 recombinant inbred lines (RILs) derived from an interspecific cross between the cultivar Caimanta of S. lycopersicum and the accession LA722 of S. pimpinellifolium, parents that were included with their hybrid as experimental testers. These 21 genotypes were also evaluated for fruit shelf life, weight, firmness, reflectance percentage, chroma index, shape index, pH, titratable acidity, soluble solids content, pericarp width, and number of locules. Polypeptide profiles were polymorphic among ripening stages within genotypes and among genotypes within the ripening stages. Some polypeptides segregated in the Mendelian expected proportion 1:1, and showed genetic linkeage by single point analysis with fruit quality traits. Hence, eight quantitative trait loci (QTLs) associated to number of locules, weight, pH, firmness and fruit shelf life, were detected in this report.
Keywords: Solanum section Lycopersicon; Plant genetic resources; Plant breeding; Molecular markers; QTL (quantitative trait loci).
INTRODUCCIÓN
El tomate cultivado (Solanum lycopersicum, 2n = 2x = 24) es miembro de la pequeña sección Lycopersicon dentro del gran género Solanum, familia Solanaceae (10). Esta sección consta de 12 especies, las cuales muestran un alto grado de homosecuencialidad (5), aunque estudios más recientes detectaron cambios en la estructura cromosómica (2). El centro de origen del tomate es la región andina que hoy comparten Colombia, Ecuador, Perú, Bolivia y Chile. En esta área crecen espontáneamente las diversas especies de la sección. También en esta zona, S. lycopersicum muestra su mayor variación (10). El antepasado más probable del tomate cultivado es el tomate pequeño silvestre conocido como cherry, S. lycopersicum var. cerasiforme (4).
Las especies emparentadas con S. lycopersicum han aportado resistencias a insectos, enfermedades y a condiciones ambientales adversas tales como sequía y salinidad (4). Estas especies también suelen presentar variabilidad para las características de calidad de los frutos como sabor, aroma, coloración y textura, dado que en su hábitat nativo estos atributos los harían más atractivos a los predadores, constituyendo una estrategia que aseguraría la dispersión de las semillas (8). Por lo tanto, el uso de las especies silvestres constituye una alternativa para ampliar la base genética del cultivo. Algunos autores (13, 19) demostraron que también constituyen una fuente de variabilidad genética para prolongar su vida poscosecha sin afectar otros atributos de calidad de los frutos.
Zorzoli et al. (20) comenzaron una selección antagónica-divergente conducida según el método genealógico para el peso y la vida poscosecha de los frutos en una generación F2 proveniente de un cruzamiento entre S. lycopersicum cv. Caimanta, que posee corta vida poscosecha, y la entrada LA722 de S. pimpinellifolium. Como resultado de seis ciclos de selección, se generó un conjunto de líneas endocriadas recombinantes (RILs, recombinant inbred lines) divergentes en sus valores de peso y vida poscosecha de los frutos (15). Por otro lado, Pratta et al. (13) caracterizaron por marcadores moleculares AFLP estas RILs y detectaron loci de caracteres cuantitativos (QTLs) asociados a caracteres de calidad.
Como herramientas postgenómicas, los perfiles de polipéptidos son metodologías que ayudan a caracterizar genotipos. Estos perfiles de polipéptidos han sido exitosamente utilizados en diversas especies como marcadores moleculares (3, 7, 16). Aunque son menos polimórficos que los marcadores de ADN, pueden brindar rápidamente información sobre la presencia de variabilidad genética o de transcriptos intermedios para caracteres asociados al proceso biológico bajo estudio, ya que refieren a variabilidad para regiones genómicas efectivamente expresadas.
De hecho, Pratta et al. (12) utilizaron la técnica de SDS-PAGE y detectaron diferencias en el estado verde maduro del fruto en una banda correspondiente a un polipéptido de 34 kDa, que fue abundante en el genotipo homocigota para el gen mutante para la madurez nor de S. lycopersicum, pero estuvo ausente en los genotipos cv. 'Platense Italiano' de S. lycopersicum y las entradas LA1673 y LA1385 de S. lycopersicum var. cerasiforme. También, Rodríguez et al. (16) caracterizaron genotipos segregantes derivados de un cruzamiento entre el mismo homocigota para el gen nor y la entrada LA1385 de S. lycopersicum var. cerasiforme, detectando polipéptidos asociados a la duración de los estados de madurez, al peso y a la vida poscosecha de los frutos.
Objetivo
• Evaluar los perfiles de polipéptidos del pericarpio en dos estados de madurez en líneas endocriadas recombinantes de tomate y determinar sus asociaciones con caracteres de calidad del fruto.
MATERIALES Y MÉTODOS
Los experimentos se realizaron en el Campo Experimental J. F. Villarino y en el Laboratorio de cultivo in vitro y biología molecular de la Cátedra de Genética, Facultad de Ciencias Agrarias, Universidad Nacional de Rosario, ubicado en la localidad de Zavalla, Santa Fe.
Se caracterizaron 18 RILs obtenidas por Rodríguez et al. (15) y se incluyeron como testigos experimentales los progenitores del cruzamiento, S. lycopersicum cv. Caimanta, la entrada LA722 de S. pimpinellifolium y su F1 (Caimanta x LA722). La siembra se realizó en placas de Petri en cámara a 25 ± 2°C, humedad relativa ambiente y 16 horas de luz/8 horas de oscuridad. Las semillas germinadas se sembraron en vasos de plástico con tierra común en invernáculo hasta que las plántulas adquirieran un tamaño adecuado para su transplante al terreno (aproximadamente, a los 45 días de la siembra). Durante ese período se las regó con agua suficiente para evitar estrés osmótico. Los plantines (10 por genotipo) se dispusieron en 7 surcos siguiendo un diseño completamente aleatorizado. Debido a pérdidas durante el cultivo, se trabajó con un promedio de 9 plantas por genotipo, ya que el número final de plantas ensayadas fue de 198.
Los perfiles de polipéptidos de los frutos al estado verde maduro y rojo maduro fueron obtenidos por Gallo et al. (6), quienes detallaron un enfoque proteómico para el análisis de los polimorfismos entre genotipos y estados de madurez (tabla 1).
Tabla 1. Análisis de la segregación mendeliana 1:1 en el conjunto de RILs para los polipéptidos del pericarpio en los estados verde maduro (VM) y rojo maduro (RM).
Table 1. Mendelian segregation 1:1 analysis in the set of 18 RILs for pericarp polypeptides at green mature (VM) and red ripe (RM) fruits.

Para la caracterización fenotípica, en todos los materiales se evaluó la forma del fruto (F) a través del cociente entre la altura y el diámetro, el peso ( P, en g) y la vida poscosecha (VP) como el número de días transcurrido desde la cosecha hasta el ablandamiento del fruto, siguiendo la técnica propuesta por Schuelter et al. (17). Para estos caracteres, el número total de frutos medidos fue de 1850.
El color fue medido con un cromámetro a través del índice cromático a/b (donde a y b son las absorbencias a longitudes de onda de 540 nm y 675 nm, respectivamente) y del porcentaje de refectancia (L). Para evaluar las variables relacionadas con el color, se tomaron tres lecturas en la zona ecuatorial de cada fruto (número total = 136 frutos). También se evaluó la dureza del pericarpio de los frutos (Du) con un durómetro con una puntera de 0,10 cm2, tomándose dos lecturas en la zona ecuatorial de cada fruto (número total = 407 frutos). El contenido en sólidos solubles (SS, en °Brix) se evaluó con un refractómetro en el jugo homogeneizado de los frutos, sin semillas ni endocarpio (número total = 156 frutos). Se tomó una medición por homogeneizado y se realizaron 5 homogeneizados por genotipo (número total de homogeneizados = 105). La acidez se midió a través del pH y la acidez titulable (AT, de acuerdo con 16) en los mismos 5 homogeneizados que se utilizaron para medir SS, y se calculó el cociente SS/AT. Finalmente, se midió con un calibre el número de lóculos (NL) y el espesor del pericarpio (EP, en mm) , en un total de 386 frutos.
Para los caracteres cuantitativos, se obtuvo el valor medio de cada genotipo y se utilizó la prueba de Shaphiro-Wilk para verificar la distribución normal (18). Aquellos caracteres que no presentaron distribución normal fueron transformados por diferentes funciones y las comparaciones entre genotipos se hicieron por un análisis de la variancia (ANOVA) a un criterio de clasificación. Las diferencias de los valores medios de cada carácter se probaron a través del test de Duncan. El Grado de Determinación Genética (GDG) para estos caracteres se estimó a partir de un ANOVA sólo entre RILs (9). Para las variables en las cuales no fue posible lograr un ajuste a la distribución normal, la comparación entre genotipos fue hecha con la prueba no paramétrica de Kruskal-Wallis, aunque no fue posible calcular el GDG. Con el fin de estimar las asociaciones entre las RILs, se realizó un análisis multivariado de agrupamiento con la distancia Euclídea promedio, calculada a partir de todas las variables analizadas según el método UPGMA, incluyendo los progenitores y la F1.
Con la prueba no paramétrica del chi-cuadrado se determinó un modelo de herencia mendeliana 1:1 en los polipéptidos polimórficos informados por Gallo et al. (6), (tabla 1). Para detectar ligamiento entre los caracteres cuantitativos y los perfiles de polipéptidos, se utilizaron aquéllos que segregaron de forma mendeliana, descartándose el resto ya que podrían detectarse QTLs espurios si se considerasen marcadores que no ajustan a segregaciones mendelianas (9). Para identificar posibles QTLs asociados a caracteres de calidad del fruto se realizó un ANOVA a un criterio de clasificación por el método de un único punto (single point analysis), en el que la fuente de variación fue la presencia/ausencia de cada polipéptido que segregó en forma mendeliana, y la variable dependiente fue el carácter bajo estudio (9).
RESULTADOS
Los valores medios de las variables analizadas y los valores estimados del grado de determinación genética (GDG) para cada uno de los caracteres cuantitativos de calidad de fruto se muestran en la tabla 2.
Tabla 2. Valores medios de los caracteres cuantitativos del fruto y su grado de determinación genética (GDG).
Table 2. Mean valúes of fruit quality quantitative traits and their degree of genetic determination (GDG).

Las variables VP, P, L y a/b no tuvieron una distribución normal (W < 0,84, p < 0,001). La transformación de VP y P por la función logarítmica y de a/b por función cuadrática permitió obtener una distribución normal. Ninguna transformación ajustó la variable L, por lo que las comparaciones entre genotipos se realizaron por el test Kruskal-Wallis. Si bien se detectaron diferencias significativas entre RILs, no pudo calcularse el GDG para L por no cumplirse los supuestos teóricos para esta estimación.
El análisis de agrupamiento mostró una correlación cofenética de 0,89 y las distancias entre genotipos variaron entre 0,32 y 2,31 (figura).
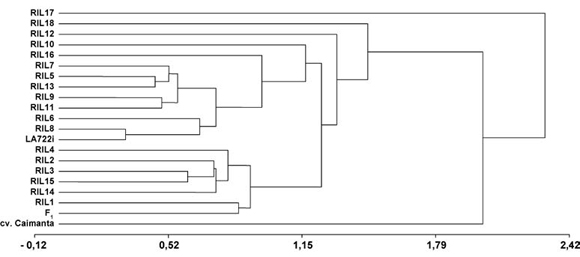
Figura. Dendrograma obtenido por análisis jerárquico en el cual se muestra el agrupamiento de las RILs y sus progenitores (cv. Caimanta, LA722 y la F1 interespecífica) por caracteres cuantitativos de calidad de fruto.
Figure. Dendrogram obtained by Hierarquical Cluster showing the grouping of RILs by means of fruit quality quantitative traits.
Se observa que la RIL17, único genotipo cuyos frutos fueron de color amarillo, se aparta del resto de los genotipos y forma un grupo único. Luego, el cv. Caimanta, de mayor peso y tamaño, se separa del resto de los genotipos, constituyendo también un grupo en forma individual. De igual manera, en un tercer nivel de agrupamiento se encuentra la RIL18, de forma ovoide, separada del resto de las RILs, LA722 y la F1 interespecífica, genotipos que se separan sólo en un quinto nivel (en un cuarto nivel se aparta la RIL12).
De un total de 24 bandas de polipéptidos polimórficas detectadas en el pericarpio de frutos al estado verde maduro, 18 (el 75%) segregaron en la proporción mendeliana 1:1 (tabla 1).
Caí: cv. Caimanta, Pimpi: accesión LA722, F,: híbrido interespecífico (Caimanta x LA722), RIL: línea endocriada recombinante derivada del híbrido interespecífico. F: forma del fruto (cociente entre la altura y el diámetro), P: Peso (en g), NL: número de lóculos, a/b: cociente entre las absorbencias a longitudes de onda de 540 nm (a) y 675 nm (b), L: porcentaje de reflectancia, Du: dureza, VP: vida poscosecha (en días), SS: contenido en sólidos solubles (en °Brix), AT: acidez titulable, SS/AT: cociente entre contenido en sólidos solubles y acidez titulable, EP: espesor del pericarpio (en cm). Valor F: cuadrado medio del tratamiento/cuadrado medio del error, * p<0,05, ** p<0,01, S/D: sin datos, NC: no corresponde.
Cai: cv. Caimanta, Pimpi: LA722, F : interspecific hybrid (Caimanta x LA722), RIL: recombinant inbred lines derived of the interspecific hybrid. Fruit traits: F: shape Índex (height/diameter ratio), P: weight (in g), NL: number of locules, a/b: chroma Índex, absorbency at a 540 nm wavelength (a) and absorbency at a 675 nm wavelength (b) ratio, L: reflectance percentage, Du: firmness, VP: shelf life (in days), SS: soluble solids content (in °Brix), AT: titratable acidity, SS/AT: soluble solids content and titratable acidity ratio, EP: pericarp width (in cm). F Valué: treatment mean square/error mean square, * p<0.05, ** p<0.01, S/D: no data available, NC: no corresponding.
El origen de cada banda, es decir, si proviene del padre Caimanta o LA722, o bien se trata de un comportamiento de novo (banda que siendo monomórfica para presencia o ausencia en los padres mostró polimorfismo -ausencia o presencia, respectivamente- en F1 o en RILs) fue informado por Gallo et al. (6) y se observa en la tabla 1. En el estado rojo maduro, 16 de un total de 23 bandas polimórficas ajustaron a la segregación 1:1 (aproximadamente 70 %, tabla 1. Cabe aclarar que en el estado verde maduro se analizaron sólo 17 líneas, ya que no se contó con muestras representativas de la RIL 2.
A partir del análisis de la varianza de los caracteres cuantitativos como variable dependiente y aquellos polipéptidos que mostraron segregación mendeliana como fuente de variación, se obtuvieron asociaciones entre los valores medios de los caracteres cuantitativos evaluados y los efectos fenotípicos sobre los caracteres asociados de la presencia / ausencia en el perfil electroforético de cada marcador de polipéptido (tabla 3).
Tabla 3. Análisis de ligamiento genético (asociación detectada por el método de un único punto a través de análisis de la variancia a un criterio de clasificación) entre polipéptidos con segregación mendeliana 1:1 y caracteres cuantitativos.
Table 3. Genetic linkage (association detected by single point analysis through one-way ANOVA) among polymorphic polypeptides adjusting to Mendelian segregation 1:1 and fruit quality quantitative traits.

Del total de 34 bandas que mostraron segregación mendeliana (1:1), se observaron cuatro asociaciones estadísticamente significativas entre los perfiles electroforéticos del estado verde maduro y los caracteres número de lóculos (con los polipéptidos de 89 y 70 kDa), pH (con el polipéptido 78 kDa) y peso (con el polipéptido de 70 kDa), así como cuatro asociaciones significativas entre los perfiles electroforéticos del estado rojo maduro y los caracteres dureza (con los polipéptido de 73, 59 y 48 kDa) y vida poscosecha (con el polipéptido de 48 kDa).
DISCUSIÓN
Los resultados encontrados indican la existencia de una amplia variación genética, tanto para la expresión de polipéptidos en dos estados fisiológicos de la madurez del fruto como en la determinación de caracteres de importancia comercial asociados a la madurez. La gran variación encontrada en los caracteres fenotípicos se debió a causas genéticas ya que los valores de GDG fueron, en general, altos (tabla 2). Esta elevada variabilidad genética se debería a la recombinación entre genes aportados por ambos progenitores. Para los caracteres fenotípicos, el diferente comportamiento mostrado por las RILs en relación con cv. Caimanta (que agrupó solo), LA722 y su F1 se observa en el agrupamiento obtenido, dado que algunas de ellas agruparon en forma independiente (RIL17, RIL18, RIL12, RIL10, entre otras), en tanto que un grupo (RILs 1, 2, 14 y 15) lo hizo con la F1 y otro (5, 6, 9 y 13, entre otras) agrupó con LA722. Los caracteres más discriminantes entre grupos fueron color, peso y forma. Resultados similares se han encontrado en trabajos anteriores para este conjunto de RILs (13, 15) y otros genotipos de tomate (4).
Por otro lado, el número de bandas de polipéptidos detectadas por estado de madurez es coincidente con informes previos (11, 12, 16), en tanto que el porcentaje de bandas que ajustaron a la segregación mendeliana esperada fue elevado (mayor al 70%) en comparación con trabajos previos (13, 16), destacándose en el conjunto de RILs la presencia de bandas de polipéptidos de novo (con comportamiento distinto al observado en los testigos experimentales) y de segregaciones transgresivas, ya que algunas RILs mostraron valores superiores o inferiores a los de sus padres para muchos caracteres. Como ejemplo de segregaciones transgresivas, reportadas por Pratta et al. (13), se encuentran las RILs 7, 17 y 18 para vida poscosecha, la RIL 16 para contenido en sólidos solubles y las RILs 17 y 18 para dureza, las que superaron en todos los casos el límite superior determinado por el padre de mayor valor para tales caracteres.
En este trabajo, la metodología empleada para estimar el ligamiento entre atributos fenotípicos y moleculares fue el análisis de un único punto, que presenta la ventaja de poder ser aplicada aun sin contar con un mapa cromosómico de la población (9). El polipéptido de 89 kDa en VM tuvo un comportamiento de novo en RILs (ausencia en algunas de ellas, mientras que estuvo presente en los tres testigos) y dicha ausencia se asoció a un menor NL, por lo que su presencia podría emplearse para seleccionar frutos de mayor tamaño (1). En el caso del polipéptido de 78 kDa en VM, también mostró la ausencia como comportamiento de novo en las RILs, y se asoció a una disminución del pH.
Para el polipéptido de 73 kDa en RM, cuya ausencia se relacionó con una mayor Du, fue aportado por el cv. Caimanta (el padre de menor valor para este carácter) y su presencia fue dominante en la F1 (6). Para el polipéptido de 59 kDa en RM, su ausencia se asoció a una mayor dureza, también fue aportado por Caimanta pero el modo de herencia en la F1 indicó que la ausencia fue la alternativa dominante (6). El comportamiento de ambos polipéptidos explicaría el hecho de que la F1 sea intermedia para Du entre ambos padres y sus efectos en las RILs fueron los esperados según el padre que los aportó (su presencia se asoció a una menor Du).
Para el polipéptido de 70 kDa del VM se obtuvo asociación con NL y con P, caracteres que están genéticamente asociados tal como propusieron Alpert & Tanksley (1). Otra asociación importante fue entre el polipéptido de 48 kDa del estado RM, con efectos significativos simultáneos sobre VP y Du. En ambos casos, al estar el mismo marcador asociado a más de un atributo fenotípico, se puede establecer que hay efecto pleiotrópico del polipéptido sobre ambos caracteres, que indica que el mismo gen los determina con su expresión o bien que el gen que codifica a este polipéptido se encuentra estrechamente ligado a dos loci distintos, cada uno de ellos con efectos sobre uno de ambos caracteres cuantitativos.
Las asociaciones polipéptido / caracteres fueron las esperadas: la presencia de la banda de 70 KDa se correspondió con un mayor P y un mayor NL, y la ausencia de la banda de 48 KDa se correspondió con una mayor VP y una mayor Du. Al observar el origen de las bandas de la tabla 3, estos dos polipéptidos tienen comportamiento de novo en la F1. Para el caso del polipéptido de 70 kDa, no está presente en los progenitores y sí lo está en la F1. El polipéptido de 48 kDa se comporta de manera opuesta, está presente en los progenitores y no lo está en la F1.
Estos comportamientos de novo podrían deberse a reorganizaciones cromosómicas que se dan frecuentemente en cruzamientos interespecíficos y que ayudarían a superar el estrés genómico que se produce al combinarse dos entidades genéticas evolutivamente alejadas (2, 14). Los resultados encontrados en este trabajo sugieren que tales reorganizaciones pueden producir efectos fenotípicos importantes, tales como los encontrados por las bandas con comportamientos de novo sobre los caracteres de calidad del fruto de tomate.
Este conjunto de RILs se generó por el método genealógico en forma antagónica-divergente (15) y puede observarse que aquellas RILs que se seleccionaron para alto P expresan el polipéptido de 70 kDa, por ejemplo la RIL 1 con un peso de 40 g. Sin embargo, la RIL 8 es de bajo peso de los frutos pero presenta la banda, lo que insinuaría que no existe ligamiento absoluto entre el polipéptido y el QTL sino que un cierto porcentaje de recombinación ocurrió en esta región cromosómica durante los sucesivos ciclos de autofecundación. Por otro lado, la ocurrencia de bandas de novo asociadas a la VP de los frutos podría explicar la segregación transgresiva fijada en algunas RILs (16), ya que ambas situaciones representan comportamientos nuevos -no presentes en ninguno de los progenitores- en los niveles molecular y fenotípico, respectivamente.
CONCLUSIONES
En frutos en dos estados de madurez de 18 líneas endocriadas recombinantes de tomate se encontraron bandas de polipéptidos cuya herencia responde a una segregación mendeliana 1:1, por lo que marcan loci únicos. La variancia genética para caracteres cuantitativos de calidad de fruto fue significativa en este grupo de materiales. Al analizar en conjunto los atributos moleculares y fenotípicos, se detectaron QTLs para peso, número de lóculos, pH, dureza y vida poscosecha de los frutos. Los programas de mejoramiento pueden beneficiarse por la incorporación de técnicas moleculares a fin de detectar QTLs que pudieran auxiliar en la selección de materiales y en la planificación de los cruzamientos.
1. Alpert, K.; Tanksley, S. D. 1996. High-resolution mapping and isolation of a yeast artificial chromosome contig containing fw2.2: a major fruit weight quantitative locus in tomato. Proceedings of the National Academy of Sciences 93: 15503-15507. [ Links ]
2. Anderson, L. K.; Covey, P. A.; Larsen, L. R.; Bedinger, P.; Stack, S. M. 2010. Structural differences in chromosomes distinguishing species in the tomato clade. Cytogenetic and Genome Research 129: 24-34. [ Links ]
3. De Luca, S.; Creus, J.; O'Grazi, D.; Dondini, L. Bregoli, A.; Serafín-Fracassini, D. 2000. Suber vegetative stages and cells cycle in Helianthus tuberosus protein pattern and their modification by spermidine. Plant Physiology 156: 17-25. [ Links ]
4. Foolad, M. R. 2007. Genome mapping and molecular breeding of tomato. International Journal of Plant Genomics. Doi: 10.1155/2007/64358. [ Links ]
5. Galiana-Balaguer, L.; Roselló, S.; Nuez, F. 2006. Characterization and selection of balanced sources of variability for breeding tomato (Lycopersicon) internal quality. Genetic Resources and Crop Evolution 53: 907-923. [ Links ]
6. Gallo, M.; Rodríguez, G. R.; Zorzoli, R.; Picardi, L. A.; Pratta, G. R. 2010. Proteómica de la madurez del tomate: Identificación de dos estados de madurez del fruto por perfiles proteicos totales del pericarpio en RILs de tomate. Revista de la Facultad de Ciencias Agrarias. Universidad Nacional de Cuyo. Mendoza. Argentina. 42(2): 119-133. [ Links ]
7. Garello, G.; Barthe, P.; Bonelli, M.; Bianco-Trinchant, J.; Le Page-Degivry, M. T. 2000. Abscisic acid regulated responses of dormant and non-dormant embryos of H. Annus: Role of ABA-inducible proteins. Plant Physiology and Biochemics 38: 473-482. [ Links ]
8. Grierson, D.;Fray, R. 1994. Control of ripening in transgenic tomatoes. Euphytica 79: 251-263. [ Links ]
9. Kearsey, M.; Pooni, H. 1996. The genetical analysis of quantitative traits. Great Britain. Chapman and Hall. 381 p. [ Links ]
10. Peralta, I. E.; Spooner, D. M. 2001. GBSSI gene phylogeny of wild tomatoes (Solanum L. section Lycopersicon [Mill.] Wettst. subsection Lycopersicon). American Journal of Botany 88: 1888-1902. [ Links ]
11. Pereira da Costa, J. H.; Rodríguez, G. R.; Pratta, G. R.; Zorzoli, R.; Picardi L. A. 2009. Characterization of tomato germoplasm by pericarp protein profiles and morphologic and biochemical fruit traits. Fruits, Vegetable and Cereal Science and Biotechnology 3: 48-53. [ Links ]
12. Pratta, G. R.; Zorzoli, R.; Picardi, L. A.; Valle, E. M.; Carrillo, N. 2001. Characterization of tomato genotypes that differ in their fruit shelt-life by analysis of total pericarp protein patterns at two ripening stages. Acta Horticulturae 546: 483-487. [ Links ]
13. Pratta, G. R.; Rodriguez, G. R.; Zorzoli, R.; Valle, E. M.; Picardi, L. A. 2011. Phenotypic and molecular characterization of selected tomato recombinant inbred lines derived from a cross Solanum lycopersicum x S. pimpinellifolium. Journal of Genetics 90: 229-237. [ Links ]
14. Rieseberg, L. H. 2001. Polyploid evolution: Keeping the peace at genomic reunions. Current Biology 11: 925-928. [ Links ]
15. Rodríguez, G. R.; Pratta, G. R.; Zorzoli, R.; Picardi, L. A. 2006. Recombinant lines obtained from an interspecific cross between Lycopersicon species selected by fruit weight and fruit shelf life. Journal of the American Society for Horticultural Sciences 131: 651-656. [ Links ]
16. Rodríguez, G. R.; Sequin, L.; Pratta, G. R.; Zorzoli, R.; Picardi L. A. 2008. Protein profiling in F1 and F2 generations of two tomato genotypes differing in ripening time. Biologia Plantarum 52: 548-552. [ Links ]
17. Schuelter, A. R.; Finger, F. L.; Casali, V. W. D.; Brommonschenkel, S. H.; Otoni, W. C. 2002. Inheritance and genetic linkage analysis of a firm-ripening tomato mutant. Plant Breeding 121: 338-342. [ Links ]
18. Shapiro, S. S.; Wilk, M. B. 1965. An analysis of variance test for normality (complete samples). Biometrica 52: 591-611. [ Links ]
19. Zorzoli, R.; Pratta, G. R.; Picardi, L. A. 1998. Efecto de los mutantes nor y rin y de genes silvestres sobre características del fruto en Lycopersicon. Mendeliana 13: 12-19. [ Links ]
20. Zorzoli, R.; Pratta, G. R.; Picardi, L. A. 2000. Variabilidad genética para la vida postcosecha y el peso de los frutos en tomate para familias F3 de un híbrido interespecífico. Pesquisa Agropecuaria Brasileira 35: 2423-2427. [ Links ]














