Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Revista de la Facultad de Ciencias Agrarias. Universidad Nacional de Cuyo
versão On-line ISSN 1853-8665
Rev. Fac. Cienc. Agrar., Univ. Nac. Cuyo vol.45 no.2 Mendoza dez. 2013
ARTÍCULOS ORIGINALES
Utilización de marcadores morfológicos y moleculares AFLP en la identificacíon de germo plasma nativo y cultivado de Elymus scabrifolius (Poaceae)
Utilization of morphological and AFLP molecular markers in the identification of native and cultivated germplasm of Elymus scabrifolius (Poaceae)
Pablo A. Tomas 1, Alexandra M. Gottlieb 2, 3, Gustavo E. Schrauf 4, Lidia Poggio 2, 3
1 Cátedra de Mejoramiento Vegetal y Animal. Facultad de Ciencias Agrarias.Universidad Nacional del Litoral. R. P. Kreder 2805. Esperanza, Santa Fe, Argentina. S3080HOF. patomas@fca.unl.edu.ar
2 Consejo Nacional de Investigaciones Científicas y Técnicas (CONICET). Av. Rivadavia 1917. Buenos Aires, Argentina. C1033AAJ.
3 Dpto. de Ecología, Genética y Evolución. Facultad de Ciencias Exactas y Naturales. Universidad de Buenos Aires. Int. Güiraldes 2160. Ciudad Universitaria. Buenos Aires, Argentina. C1428EGA.
patomas@fca.unl.edu.ar
4 Cátedra de Genética. Facultad de Agronomía. Universidad de Buenos Aires. Av. San Martín 4453. Buenos Aires, Argentina. C1417DSE.
Originales: Recepción: 26/03/2012 - Aceptación: 20/04/2013
Resumen
Elymus scabrifolius es una gramínea perenne nativa de Sudamérica con gran potencial como recurso forrajero para ambientes con limitantes edáficas. En el presente trabajo se analizó la utilización de caracteres morfológicos y marcadores moleculares AFLP para la identificación genotípica de seis accesiones, un cultivar comercial y siete híbridos artificiales de esta especie. Ambos tipos de marcadores permitieron diferenciar a los materiales analizados en los respectivos dendrogramas, aunque las relaciones entre materiales variaron según el tipo de marcador. El Análisis de Componentes Principales permitió identificar las variables más relevantes para la diferenciación morfológica. Los híbridos se diferenciaron morfológicamente de ambos parentales, excepto un híbrido que se agrupó con su material paterno. Aunque en el análisis de los marcadores AFLP los híbridos se agruparon con uno de sus parentales, se pudo corroborar su origen híbrido mediante el registro de bandas paternas y polimórficas entre parentales. Se concluye que las metodologías empleadas para caracterizar los materiales analizados de E. scabrifolius serían de gran utilidad para el manejo eficiente de colecciones de germoplasma como así también para su utilización en programas de mejoramiento genético.
Palabras clave: Elymus; Identificación de germoplasma; Morfología; Marcadores moleculares; AFLP
Abstract
Elymus scabrifolius is a South American perennial grass exhibiting great potential as a forage resource, especially for environments with edaphic limitations. In the present work we use morphological traits and AFLP molecular markers to genotypically identify six E. scabrifolius accessions, one commercial cultivar and seven artificial hybrids of this species. Both types of marker systems resulted in an accurate differentiation of the interrogated E. scabrifolius germplasm in the dendrograms. Moreover, relationships between the tested germplasm tend to vary according to the type of marker system used. Principal Component Analysis was used to identify the traits that best described the morphological variation. The morphological study revealed that almost all hybrids were different from their parents, with exception of one hybrid that was clustered with their own male parent. In the AFLP marker analysis, all the hybrids were clustered with one of their own parents. However, hybrid origin of those individuals was verified by the presence of paternal bands and polymorphic bands. In conclusion, the use of different methodologies to characterize and analyze E. scabrifolius materials has the potential to become of great benefit for the efficient management of germplasm collections and also for their utilization in breeding programs.
Keywords: Elymus; Germplasm identification; Morphology • molecular markers; AFLP
Introducción
Elymus L. (Poaceae, Triticeae) es el género más ampliamente distribuido y diverso dentro de la tribu Triticeae, incluyendo numerosas especies con caracteres agronómicos deseables como tolerancia a diversos tipos de estrés y calidad forrajera (8, 31). Este género está representado en Sudamérica por siete especies, seis de las cuales son nativas de Argentina (25) y solo dos de ellas, Elymus scabrifolius (Döll) J. H. Hunz. y E. scabriglumis (Hack.) Á. Löve, presentan potencial como recurso de interés agronómico.
E. scabrifolius es una gramínea alotetraploide perenne (2n = 4x = 28) ampliamente distribuida en la región templado-cálida de Argentina y Uruguay (15). Es una especie de reproducción preferentemente autógama, dado que los individuos se encuentran aislados en poblaciones naturales y que la dehiscencia del polen ocurre al momento de la antesis, aunque la excersión de anteras y estigmas posibilitaría eventualmente la ocurrencia de polinización cruzada (P. A. Tomas, comunicación personal). Esta especie es valorada como un potencial recurso forrajero por presentar tolerancia a diversas limitantes edáficas y elevada calidad forrajera (19, 32, 33). Fue considerada una especie en riesgo debido a la reducción de su hábitat natural (1), siendo incluida en programas de colecta, conservación y caracterización de germoplasma para ambientes marginales. Actualmente, existe un material comercial de E. scabrifolius (El Triunfo, Universidad Nacional del Litoral - Universidad de Buenos Aires, Cereagro SRL) y una importante colección de materiales silvestres, líneas elite e híbridos tanto en jardín introductorio como en el banco de germoplasma de INTA Pergamino (34).
La utilidad de una colección de germoplasma depende de la caracterización disponible, la cual permite un mejor aprovechamiento de la diversidad genética y un uso eficiente de los recursos. La correcta identificación y caracterización de los genotipos promisorios es una instancia previa a su introducción en programas de mejora genética (7, 18).
Por otro lado, la generación de nueva variabilidad a partir del germoplasma disponible generalmente implica la obtención de híbridos artificiales entre materiales selectos, cuya identificación en estados iniciales del desarrollo es de gran importancia para el mejoramiento de especies perennes, reduciendo el tiempo y los costos en el mantenimiento de líneas madre (5, 6, 17, 28).
La identificación genotípica ha sido tradicionalmente basada en caracteres morfológicos y fisiológicos. Sin embargo, la limitada variación entre materiales muy relacionados genéticamente y el efecto del ambiente dificultan la diferenciación y afecta su precisión en colecciones extensas (5). Por el contrario, las técnicas de marcadores moleculares de ADN presentan la ventaja de independizarse del estado de desarrollo de la planta y de la influencia del ambiente en la identificación varietal (12, 20).
La técnica de polimorfismos en la longitud de los fragmentos de restricción (Amplified Fragment Length Polymorphism, AFLP) (30) ha sido empleada con éxito en análisis de diversidad e identificación genotípica en forrajeras perennes (4, 22), incluyendo diversas especies de Elymus (13, 16, 23, 27, 29).
Objetivos
• Evaluar y comparar el uso de caracteres morfológicos y marcadores AFLP en la identificación genotípica de líneas e híbridos en el manejo del germoplasma de E. scabrifolius y su perspectiva de aplicación en programas de mejoramiento genético.
Materiales y métodos
Material vegetal
Se analizaron seis accesiones, un cultivar comercial y siete híbridos intraespecíficos de E. scabrifolius (tabla 1, pág. 88), pertenecientes a la colección de germoplasma de la Facultad de Ciencias Agrarias, Universidad Nacional del Litoral (Santa Fe, Argentina). Las accesiones corresponden a ejemplares únicos colectados en poblaciones silvestres y conservados como planta individual en la mencionada colección.
Tabla 1. Materiales de E. scabrifolius incluidos en el presente estudio.
Table 1. Materials of E. scabrifolius included in the present study.

El material comercial fue provisto por la empresa Cereagro SRL y es multiplicado en el jardín introductorio de la colección como población mejorada de autógama. Los individuos analizados en este estudio fueron obtenidos a partir de semillas cosechadas de las accesiones y del cultivar comercial, en tanto que los híbridos intra-específicos corresponden a ejemplares únicos obtenidos mediante cruzamientos dirigidos, realizando emasculación y polinización manual, seguidas de ensobrado para prevenir la llegada de polen no deseado. Las semillas fueron germinadas en cámaras húmedas con alternancia de temperatura según Zabala et al. (32). Las plántulas fueron colocadas en macetas y transplantadas luego de dos meses al jardín introductorio de la colección (31°27' S, 60°56' W), ubicadas como plantas individuales espaciadas a 70 cm entre sí en un diseño completamente aleatorizado.
Caracterización morfológica
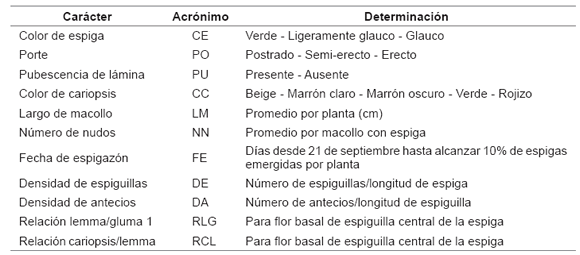
En las plantas individuales de cada material se realizaron determinaciones para 11 caracteres morfológicos (tabla 2) mayormente asociados a la etapa reproductiva para mejorar la precisión y repetibilidad del fenotipo. Para los caracteres cuantitativos se calculó el promedio de cinco determinaciones para cada individuo analizado.
Tabla 2. Caracteres morfológicos evaluados en materiales de E. scabrifolius.
Table 2. Morphological characters evaluated in E. scabrifolius materials.
Caracterización molecular
El ADN genómico total fue extraído a partir de hojas jóvenes de cada planta según Doyle & Doyle (11) con modificaciones menores. Cada muestra fue cuantificada por comparación con estándar de peso molecular (Lambda EcoRI/HindIII, Promega Corp.) mediante electroforesis en gel de agarosa 0,8% (p/v) en solución tampón TAE 1X, y luego almacenada a 4°C hasta su uso.
El análisis AFLP fue realizado según Gottlieb et al. (14). Para cada muestra, 250 ng de ADN genómico total fueron digeridos con enzimas EcoRI y MseI, siendo luego ligados a los adaptadores correspondientes. La reacción de preamplificación por PCR utilizó cebadores con una base selectiva (EcoRI+A y MseI+C), en tanto que para la reacción de amplificación selectiva se emplearon combinaciones de cebadores con tres bases selectivas.
Ocho combinaciones de cebadores selectivos fueron analizadas en forma preliminar en cuatro accesiones; fueron seleccionadas las siguientes cinco combinaciones para ser utilizadas en todos los materiales, por su repetibilidad y practicidad para el análisis: EcoRI+AGC/MseI+CTC; EcoRI+AGG/MseI+CAC; EcoRI+AGG/MseI+CTC; EcoRI+ACT/MseI+CTA; EcoRI+AAC/MseI+CAG.
Los productos de amplificación fueron separados por electroforesis en geles desnaturalizantes de poliacrilamida 6% (p/v) con 5 M de urea. La electroforesis fue realizada en una cuba S2 (GIBCO BRL Sequencing System, Life Technologies) en tampón TBE 1X por 4 hs a 55 W. En cada gel fue incluido por duplicado un estándar de peso molecular de rango 30-330 pb (Life Technologies). Los geles fueron teñidos con nitrato de plata según Bassam & Caetano-Anollés (3) y luego digitalizados. Los patrones de bandas fueron registrados en el rango de 30-330 pares de bases.
Análisis de datos
Con los datos morfológicos se analizó en primera instancia la capacidad de cada variable para discriminar entre accesiones. Los caracteres cualitativos fueron registrados como variable categórica, en tanto que los caracteres cuantitativos (variables y cocientes) fueron analizados por ANVA y las medias comparadas por Tukey (p < 0,05), previo test de normalidad y homogeneidad de varianza.
En el caso de los híbridos, por tratarse de ejemplares únicos no se contó con repeticiones como para ser evaluados, pero sí fueron incluidos en el análisis multivariado.
En segunda instancia se construyó una matriz de 42 unidades taxonómicas operacionales (OTUs) x 11 caracteres morfológicos. Se realizó un análisis de agrupamiento con los valores previamente estandarizados, utilizando la distancia de Manhattan y la media aritmética no ponderada (UPGMA, unweighted pair-groupmethod with arithmetic averages) para la confección del dendrograma correspondiente. Se realizó además un Análisis de Componentes Principales (ACP) para identificar patrones de variación y los caracteres de mayor peso para la variabilidad observada.
Los datos moleculares fueron registrados como presencia [1] o ausencia [0] para cada banda. Solo fueron utilizados para el análisis los fragmentos polimórficos, siendo considerados como tal aquellos con ausencia de banda en al menos un individuo. El análisis de agrupamiento se realizó a partir de las distancias genéticas estimadas con el coeficiente de Dice (10) considerando D = 1 - S. Para la representación gráfica mediante un dendrograma se utilizó el algoritmo UPGMA a partir de los valores de distancia genética tomados como D' = √ D por producir la menor distorsión.
Con el fin de determinar la utilidad del análisis molecular para la verificación del origen híbrido de los materiales producto de cruzamientos, se registraron tanto las bandas consideradas diagnósticas (presentes exclusivamente en todos los individuos de un material), como así también la presencia de bandas paternas (presentes solo en el donante de polen) y polimórficas entre parentales, correspondientes a cada cruzamiento y presentes en el híbrido.
Las distorsiones entre la matriz de distancia y las respectivas representaciones gráficas fueron estimadas mediante el Coeficiente de Correlación Cofenética (CCC) (26). Todos los análisis de datos fueron realizados utilizando el software estadístico Infostat v2011p (9).
Resultados
Análisis morfológico
Tanto las determinaciones realizadas para caracteres cualitativos como el análisis de varianza para caracteres cuantitativos evidenciaron la existencia de considerable variabilidad entre los materiales analizados (tabla 3, pág. 91).
Tabla 3. Evaluación de materiales de E. scabrifolius utilizando caracteres morfológicos. Para caracteres cuantitativos se presentan los promedios y valor p (** = p < 0,01; ns = diferencias no significativas).
Table 3. Assessment of E. scabrifolius materials using morphological traits. For quantitative traits, average and p value are showed (** = p < 0.01; ns = no significant differences).

Mediante el análisis conjunto con técnicas numéricas, los caracteres empleados permitieron diferenciar los individuos pertenecientes a cada material analizado. En el dendrograma (CCC = 0,846) los individuos de cada material formaron grupos definidos. Las distancias genéticas entre individuos de diferentes materiales fueron mayores a las distancias entre ejemplares de un mismo material, excepto para la accesión G113, la cual presentó mayor variación entre sus individuos. Si bien los materiales G401 y G912 presentaron la menor distancia genética entre sí, sus individuos se agruparon por accesión.
La gran mayoría de los individuos híbridos se diferenciaron de sus dos respectivos parentales. El híbrido H42 presentó la mayor distancia genética respecto de sus parentales. Los híbridos H15, H54 y H59, que comparten como progenitor el cultivar comercial ET, se agruparon juntos, en tanto que H191 y H192, que comparten ambos parentales, mostraron escasa distancia entre sí. A diferencia de los híbridos mencionados, H41 se agrupó junto a su parental paterno G113 (figura 1a).

Figura 1. a) Dendrograma basado en datos morfológicos, realizado utilizando la distancia de Manhattan y el algoritmo UPGMA.
b) Dendrograma basado en datos moleculares, realizado utilizando el coeficiente de Dice y el algoritmo UPGMA.
Figure 1. a) Dendrogram based on morphological data, using Manhattan distances and the UPGMA algorithm.
b) Dendrogram based on molecular data, using the Dice's coefficient and UPGM algorithm.
En el ACP las dos primeras componentes explicaron el 54,7% de la variación total observada (CCC = 0,926); se advirtió un patrón de variación coincidente con lo observado en el análisis de agrupamiento (datos no presentados). La magnitud de los vectores principales de los caracteres analizados sobre cada eje son presentados en la tabla 4. Los caracteres Porte, Color de cariopsis, Largo de macollo y Número de nudos fueron los de mayor relevancia para la Componente Principal 1, mientras que para la Componente Principal 2, Color de espiga y Pubescencia de lámina fueron los caracteres que mejor explicaron la dispersión observada. Adicionalmente, se realizó un ACP solamente con esos seis caracteres de mayor relevancia, pero no se observó una adecuada diferenciación de las accesiones G401, G404 y G912 (datos no presentados).
Tabla 4. Vectores principales y proporción de la variación total explicada sobre las dos primeras componentes del ACP para los caracteres morfológicos utilizados para clasificar los materiales analizados de E. scabrifolius. CCC = 0,926.
Table 4. Eigenvectors and proportion of total variance explained by the first two principal components for morphological traits used to classify the E. scabrifolius analyzed materials. CCC = 0.926.

Análisis molecular
Las cinco combinaciones de cebadores AFLP utilizadas generaron 328 bandas, 50 de las cuales (15,2%) fueron polimórficas (tabla 5, pág. 94) y permitieron diferenciar todos los materiales analizados. El 42% de las bandas polimórficas variaron solamente entre materiales, mientras que las restantes variaron tanto entre como dentro de los materiales analizados.
Tabla 5. Número y tipo de bandas obtenidas mediante cada combinación de cebadores AFLP en materiales de E. scabrifolius.
Table 5. Number and type of bands obtained for each AFLP primer combination in analyzed materials of E. scabrifolius.

En el dendrograma correspondiente (CCC = 0,904) los individuos se agruparon según el material al cual pertenecían (figura 1b, pág. 92). Los coeficientes de distancia genética entre materiales fueron variables, con rango de 0,21 (entre G101 y G113) a 0,62 (entre G404 y ET) y promedio de 0,37. No obstante, la distancia entre los materiales siempre fue mayor que dentro de los materiales. Los individuos de una misma accesión o cultivar presentaron entre sí distancias genéticas muy bajas, variando de 0,03 (para G214) a 0,13 (para G404) y en promedio 0,07.
Adicionalmente, se intentó determinar el mínimo número de ensayos a realizar para una adecuada diferenciación entre los materiales analizados, lo cual se logró utilizando solamente las dos combinaciones de cebadores que mayor número de polimorfismos presentaron (datos no presentados). Por otro lado, el patrón de agrupamiento entre materiales fue diferente al observado para datos morfológicos.
Todos los híbridos se agruparon próximos a alguno de sus materiales parentales en el dendrograma (figura 1b, pág. 92). Mediante el registro de bandas polimórficas entre los respectivos parentales fue posible corroborar el origen por cruzamientos para los siete híbridos analizados. Sóoo pudieron ser identificadas dos bandas diagnósticas, correspondientes a los materiales ET y G214, las cuales estuvieron presentes en los híbridos en que participaron esos materiales.
Según el cruzamiento, para el conjunto de las cinco combinaciones de cebadores AFLP variaron tanto el número de bandas polimórficas entre parentales (entre 3 y 22) como el número de bandas paternas (entre 1 y 9). Del mismo modo, la cantidad de bandas paternas y polimórficas entre parentales generadas por cada par de cebadores varió según el nivel de polimorfismos observados en cada combinación (tabla 5, pág. 94). Para verificar el origen de todos los híbridos aquí analizados fueron necesarias al menos dos combinaciones de cebadores.
Discusión
En este estudio se analizó la utilización de caracteres morfológicos y marcadores moleculares AFLP en la identificación genotípica en diferentes materiales de Elymus scabrifolius.
Utilizando 11 caracteres morfológicos determinados mayormente sobre estructuras reproductivas fue posible identificar todos los materiales de E. scabrifolius analizados. La gran mayoría de los caracteres evaluados individualmente, tanto cuantitativos como cualitativos, evidenciaron marcadas diferencias entre materiales, permitiendo su caracterización y posterior elección como fuente de variabilidad. Por otro lado, en el análisis multivariado las distancias genéticas entre individuos de una misma accesión o cultivar fueron considerablemente menores a las distancias entre individuos de diferentes materiales, lo que sugiere que las diferencias morfológicas existentes entre materiales son consistentes y permiten una adecuada identificación de los mismos. De modo similar, numerosos estudios señalan la utilidad de los caracteres reproductivos para la identificación genotípica y el manejo de los materiales en colecciones de germoplasma en diversas especies (5, 7, 18).
Seis de los siete híbridos analizados se diferenciaron claramente, tanto de sus parentales como también de los demás materiales, en el dendrograma basado en caracteres morfológicos. Los híbridos H191 y H192 se agruparon juntos, lo cual era esperable dado que comparten ambos parentales. El híbrido H42 se diferenció marcadamente del resto de los ejemplares, indicando una diferenciación fenotípica considerable para el producto de ese cruzamiento. Los híbridos H15, H54 y H59, que comparten como parental el material comercial ET, se agruparon próximos entre sí, lo cual indicaría que ET transmitiría una morfología particular a sus híbridos, aunque diferenciable del fenotipo parental, independientemente de su participación como donante o receptor de polen. A diferencia de los híbridos antes mencionados, H41 se agrupó próximo a los individuos de su parental paterno G113, lo que indicaría que el fenotipo de este último, al menos para los caracteres analizados, fue claramente dominante sobre la morfología del otro parental.
El ACP basado en datos morfológicos permitió determinar la importancia relativa de las variables consideradas en el análisis. Seis de los 11 caracteres analizados en este estudio presentaron efecto significativo sobre las dos primeras componentes, aunque utilizando solamente esos seis caracteres no fue posible diferenciar tres de las accesiones de E. scabrifolius analizadas. Esto coincide con estudios que reportan una menor diferenciación entre materiales en ACP respecto del análisis de agrupamiento (2, 35), posiblemente debido a la capacidad relativa de discriminación de los caracteres morfológicos respecto de cada material. En función de los resultados aquí presentados, realizar una caracterización de materiales de E. scabrifolius utilizando un reducido número de caracteres podría disminuir sensiblemente la eficiencia de identificación genotípica.
Los marcadores AFLP presentaron considerable información sobre la variación genética en materiales de E. scabrifolius, siendo el primer estudio a nivel de marcadores de ADN realizado en la especie. Aunque solo el 15% de las bandas fueron polimórficas y únicamente dos de ellas fueron exclusivas de algún material, los datos moleculares permitieron una clara discriminación entre los materiales analizados, los cuales se caracterizan por presentar distintas combinaciones de alelos compartidos entre dos o más materiales, más que en la presencia de alelos propios. Asimismo, los valores estimados de distancia genética entre materiales y porcentaje de polimorfismos generados en este estudio fueron consistentes con los niveles de diferenciación observados en otras especies de Elymus autógamas utilizando marcadores AFLP (23, 27), los cuales reportaron distancias promedio de 0,25 a 0,41 con porcentajes de polimorfismo entre 9 y 35%.
Por otro lado, utilizando solamente las dos combinaciones de cebadores más informativas también fue posible la diferenciación de los materiales aquí analizados. Estudios en otras gramíneas autógamas mencionan el uso de pocas combinaciones de cebadores AFLP para una adecuada identificación de germoplasma (24, 36). Por consiguiente, una selección de las combinaciones de cebadores más informativos sería de utilidad para el uso eficiente de herramientas moleculares en la caracterización de germoplasma de especies con bajo porcentaje de bandas polimórficas como E. scabrifolius.
La diversidad genética dentro de los materiales fue muy baja, en concordancia con el modo de reproducción propuesto para E. scabrifolius, aunque más de la mitad de las bandas polimórficas variaron tanto entre como dentro de los materiales.
Estos resultados sugieren, por un lado, que los ejemplares originales que integran la colección de germoplasma tienen algún grado de heterosis y, por otro lado, que diferentes materiales serían heterocigotos para diferentes loci analizados. Este último aspecto también debería ser considerado en planes de conservación de la diversidad genética de la especie.
En este estudio se observaron diferencias entre los agrupamientos derivados de datos morfológicos y aquellos obtenidos de datos moleculares. Este tipo de contraste entre las distancias genéticas obtenidas a partir de datos morfológicos y moleculares ha sido reportado para numerosas especies (5, 18, 21), lo cual estaría relacionado con el tipo de variación que permite analizar cada tipo de dato. Los marcadores moleculares anónimos como AFLP estarían reflejando la variación genética para regiones no codificantes, en tanto que el análisis del fenotipo se basa en caracteres producto de la expresión génica. En este estudio, el número de bandas polimórficas analizadas es demasiado reducido como para permitir una adecuada asociación con la variación fenotípica observada. Como consecuencia, se contaría con una baja cobertura del genoma mediante los marcadores generados, lo cual no reflejaría la compleja base genética de los caracteres morfológicos aquí analizados. Por ello, las relaciones observadas entre materiales para ambos tipos de datos deberían ser consideradas complementariamente.
El análisis de datos moleculares ha sido empleado en diversas especies para la identificación de genotipos híbridos a partir de la distancia genética con sus parentales en un dendrograma. Sin embargo, algunos estudios demostraron que los híbridos tienden a agruparse con uno de sus parentales en los dendrogramas, variando hacia uno u otro parental según el híbrido (17, 28, 29). En concordancia con esas observaciones, todos los híbridos de E. scabrifolius analizados en este estudio se agruparon con alguno de sus respectivos parentales el análisis de datos moleculares. El número de bandas paternas varió entre 30 y 47% del total de bandas polimórficas entre parentales según el cruzamiento, siendo la distancia genética entre los híbridos y cada parental menor que la distancia entre sus parentales, lo que explica en parte el patrón de agrupamiento observado en los híbridos.
Además del análisis de agrupamiento, la simple sumatoria de bandas paternas y polimórficas entre parentales en los híbridos resultó una herramienta útil en la identificación de los ejemplares de E. scabrifolius producto de cruzamientos. Cada combinación de cebadores AFLP mostró diferente número de bandas paternas y polimórficas entre parentales según el híbrido. Sin embargo, ninguna combinación de cebadores por sí sola permitió identificar todos los híbridos de E. scabrifolius, debido principalmente a la similitud genética entre parentales en algunos cruzamientos. La identificación de híbridos mediante bandas paternas resulta limitada cuando el número de bandas informativas es bajo debido a dicha similitud genética entre parentales (6, 17). Los resultados del presente estudio sugieren que es necesario un testeo inicial de los cebadores para determinar el menor número de amplificaciones a realizar en la identificación de híbridos de E. scabrifolius.
Conclusión
Los resultados aquí presentados sugieren que la caracterización morfológica en estadios reproductivos y el uso de marcadores moleculares AFLP en materiales de E. scabrifolius serían herramientas de gran utilidad en el manejo del germoplasma de la especie. Las accesiones analizadas presentaron considerables niveles de variación genética, acordes al origen, sistema de polinización y forma de multiplicación de los materiales. El uso de aquellos caracteres morfológicos más informativos junto a la caracterización molecular permitiría optimizar la conservación de la diversidad genética al simplificar la identificación de las accesiones más contrastantes. La realización de cruzamientos y una correcta verificación de la naturaleza híbrida de los materiales obtenidos garantizan la posibilidad de combinar la variabilidad existente en la colección para su aprovechamiento en programas de mejora genética.
En función del nivel de polimorfismo observado mediante marcadores moleculares AFLP, se considera necesario aumentar sensiblemente el número de combinaciones de cebadores a analizar para una exhaustiva caracterización genética de las accesiones más promisorias, con vistas a la realización de selección asistida por marcadores. Para ello podría ser recomendable también combinar los AFLP con otros tipos de marcadores, de tipo co-dominante, que permitan estimar además el grado de heterosis en las colecciones de ejemplares silvestres.
1. Andrés, A. N. 1986. Variabilidad genética en poblaciones naturales de Agropyron scabrifolium (Doell) Parodi. Tesis MSc, INTA E. E. A. Pergamino. Universidad Nacional de Rosario, Argentina.
2. Annicchiarico, P. 2006. Diversity, genetic structure, distinctness and agronomic value of Italian lucerne (Medicago sativa L.) landraces. Euphytica. 148: 269-282. [ Links ]
3. Bassam, B. J.; Caetano-Anollés, G. 1993. Silver staining of DNA in polyacrylamide gels. Appl. Biochem. Biotechnol. 42: 181-188. [ Links ]
4. Cavagnaro, P. F.; Cavagnaro, J. B.; Lemes, J. L.; Masuelli, R. W.; Passera, C. B. 2006. Genetic diversity among varieties of the native forage grass Trichloris crinita based on AFLP markers, morphological characters, and quantitative agronomic traits. Genome. 49: 906-918. [ Links ]
5. Chiorato, A. F.; Morais Carbonell, A. S.; dos Santos Dias, L. A.; Moura, R. R.; Barbosa Chiavegato, M.; Colombo, C. A. 2006. Identification of common bean (Phaseolus vulgaris) duplicates using agromorphological and molecular data. Genetics and Molecular Biology. 29(1): 105-111. [ Links ]
6. Conceição, L. D.; Belo, G. O.; Souza, M. M.; Santos, S. F.; Cerqueira-Silva, C. B. M.; Corrêa, R. X. 2011. Confirmation of cross-fertilization using molecular markers in ornamental passion flower hybrids. Genetics and Molecular Research. 10: 47-52. [ Links ]
7. Costea, M.; Brenner, D. M.; Tardif, F. J.; Tan, Y. F.; Sun, M. 2006. Delimitation of Amaranthus cruentus L. and Amaranthus caudatus L. using micromorphology and AFLP analysis: an application in germplasm identification. Genet. Resour. Crop Evol. 53: Online-First. DOI:10.1007/s10722- 005-0036-3.
8. Dewey, D. R. 1984. The genomic system of classification as a guide to intergeneric hybridization with the perennial Triticeae. In: Gene manipulation in plant improvement. Gustafson, J. P. (ed.). New York. Plenum Publishing Corporation. p. 209-279.
9. Di Rienzo, J. A.; Casanoves, F.; Balzarini, M. G.; González, L.; Tablada, M.; Robledo, C. W. 2011. InfoStat v2011p. Grupo InfoStat, Facultad de Ciencias Agropecuarias, Universidad Nacional de Córdoba, Argentina. URL http://www.infostat.com.ar
10. Dice, L. R. 1945. Measures of the amount of ecologic association between species. Ecology. 26: 297-302. [ Links ]
11. Doyle, J. J.; Doyle, J. L. 1990. Isolation of plant DNA from fresh tissue. Focus. 12: 13-15. [ Links ]
12. Espósito, M. A.; Martin, E.; Cravero, V. P.; Cointry, R. L. 2011. Uso de marcadores morfológicos, bioquímicos y moleculares SRAP para diferenciar variedades de Cynara cardunculus L. (Asteraceae). Rev. FCA UNCUYO. 43(2): 35-45. [ Links ]
13. Fahleson, J.; Okori, P.; Åkerblom-Espeby, L.; Dixelius, C. 2008. Genetic variability and genomic divergence of Elymus repens and related species. Pl. Syst. Evol. 271: 143-156. [ Links ]
14. Gottlieb, A. M.; Giberti, G. C.; Poggio, L. 2005. Molecular analyses of the genus Ilex (Aquifoliaceae) in southern South America, evidence from AFLP and ITS sequence data. Am. J. Bot. 92: 352-369. [ Links ]
15. Hunziker, J. H.; Tres, L.; De Gregorio, A. 1965. Morphology and meiotic behavior of Agropyron scabrifolium, A. breviaristatum and their hybrid. Kurtziana. 2: 79-89. [ Links ]
16. Larson, S. R.; Palazzo, A. J.; Jensen, L. B. 2003. Identification of western wheatgrass cultivars and accessions by DNA fingerprinting and geographic provenance. Crop Sci. 43: 394-401. [ Links ]
17. Lin, X. C.; Lou, Y. F.; Liu, J.; Peng, J. S.; Liao, G. L.; Fang, W. 2010. Crossbreeding of Phyllostachys species (Poaceae) and identification of their hybrids using ISSR markers. Genetics and Molecular Research. 9: 1398-1404. [ Links ]
18. Maccaferri, M.; Stefanelli, S.; Rotondo, F.; Tuberosa, R.; Sanguineti, M. C. 2007. Relationships among durum wheat accessions. I. Comparative analysis of SSR, AFLP, and phenotypic data. Genome. 50: 373-384. [ Links ]
19. Nuciari, M. 2006. Valor forrajero de Elymus breviaristatus (Hitchc) Á. Löve subsp. scabrifolius y E. scabriglumis (Hack) Á. Löve (Poaceae) del sudeste bonaerense (Argentina) a partir de caracteres histofoliares. Agrociencia. 10: 1-16. [ Links ]
20. Nybom, H. 2004. Comparison of different nuclear DNA markers for estimating intraspecific genetic diversity in plants. Mol. Ecol. 13: 1143-1155. [ Links ]
21. Qi, X.-H.; Yang, J.-H.; Zhang, M.-F. 2008. AFLP-based genetic diversity assessment among Chinese vegetable mustards (Brassica juncea (L.) Czern.) Genet. Resour. Crop Evol. 55: 705-711. [ Links ]
22. Rouf Mian, M. A.; Zwonitzer, J. C.; Chen, Y.; Saha, M. C.; Hopkins, A. A. 2005. AFLP diversity within and among Hardinggrass populations. Crop Sci. 45: 2591-2597.
23. Sasanuma, T.; Endo, T. R.; Ban, T. 2002. Genetic diversity in three Elymus species indigenous to Japan and East Asia (E. tsukushiensis, E. humidus and E. dahuricus) detected by AFLP. Genes Genet. Syst. 77: 429-438.
24. Schut, J. W.; Qi, X.; Stam, P. 1997. Association between relationship measures based on AFLP markers, pedigree data and morphological traits in barley. Theor. Appl. Genet. 95 1161-1168.
25. Seberg, O.; Petersen, G. 1998. A taxonomic revision of the genus Elymus L. s.l. (Poaceae, Triticeae) in South America. Bot. Jahrb. Syst. 120(4): 503-543. [ Links ]
26. Sokal, R. R.; Rohlf, F. J. 1962. The comparison of dendrograms by objective methods. Taxon. 11: 33-40. [ Links ]
27. Suh, J. K.; Hetterscheid, W.; Lee, A. K.; Hong, J.; Roh, M. S. 2011. Identification and evaluation of Forsythia germplasm using molecular markers. Genet. Resour. Crop Evol. 58 (8): 1225-1235.
28. Sun, G. L.; Shee, J.; Salomon, B. 2006. Molecular diversity and relationship among Elymus trachycaulus, E. subsecundus, E. virescens, E. violaceus and E. hyperarcticus (Poaceae: Triticeae) as determined by amplified fragment length polimorphism. Genome. 49: 1160-1169.
29. Szczepaniak, M.; Cieslak, E.; Bednarek, P. T. 2007. Natural hybridization between Elymus repens and Elymus hispidus assessed by AFLP analysis. Acta Societatis Botanicorum Poloniae. 76(3): 225-234.
30. Vos, P.; Hogers, R.; Bleeker, M.; Reijans, M.; Van de Lee, T.; Hornes, M.; Frijters, A.; Pot, J.; Peleman, J.; Kuiper, M.; Zabeau, M. 1995. AFLP: a new technique for DNA fingerprinting. Nucleic Acid Res. 23: 4407-4414.
31. Wang, R. R.-C.; Dong, Y.; Zhou, R. 1993. Resistance to powdery mildew and yellow dwarf in perennial Triticeae species. Genet. Resour. Crop Evol. 40: 171-176.
32. Zabala, J. M.; Tomas, P. A.; Schrauf, G. E.; Giavedoni, J. A. 2009. Seed dormancy in Elymus scabrifolius (Döll) J. H. Hunz. Seed Sci. Technol. 37(1): 241-244.
33. Zabala, J. M.; Taleisnik, E.; Giavedoni, J. A.; Pensiero, J. F.; Schrauf, G. E. 2011. Variability in salt tolerance of native populations of Elymus scabrifolius (Döll) J. H. Hunz from Argentina. Grass Forage Sci. 66(1): 109-122.
34. Zabala, J. M.; Pensiero, J. F.; Rosso, B.; Schrauf, G. E.; Tomas, P. A.; Giavedoni, J. A.; Gutiérrez, H.; Exner, E.; Kern, V.; Bortoluzzi, A.; Fornasero, L.; Toniutti, M.; Marinoni, L.; Cerino, C.; Richard, G. 2011. Programa de documentación, conservación y valoración de forrajeras nativas en la FCA (UNL), Santa Fe, Argentina. VIII Simposio Internacional de Recursos Genéticos para América Latina y El Caribe. 21 al 23 de noviembre, Quito, Ecuador.
35. Zhang, X.-Q.; Salomon, B.; von Bothmer, R. 2002. Application of random amplified polymorphic DNA markers to evaluate intraspecific genetic variation in the Elymus alaskanus complex (Poaceae). Genet. Resour. Crop Evol. 49: 397-407.
36. Zhu, J.; Gale, M. D.; Quarrie, S.; Jackson, M. T.; Bryan, G. J. 1998. AFLP markers for the study of rice biodiversity. Theor. Appl. Genet. 96: 602–611.
Agradecimientos
A los revisores anónimos por su contribución y lectura crítica del manuscrito. Este trabajo fue financiado por el Consejo Nacional de Investigaciones Científicas y Técnicas (CONICET) y el Fondo para la Investigación Científica y Tecnológica (FONCyT; ANPCyT) mediante los proyectos PICT 14119 y PICTO 13244.














