Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista de la Facultad de Ciencias Agrarias. Universidad Nacional de Cuyo
versión On-line ISSN 1853-8665
Rev. Fac. Cienc. Agrar., Univ. Nac. Cuyo vol.46 no.2 Mendoza dic. 2014
ARTICULO ORIGINAL
Caracterización genética de germoplasma de maní cultivado (Arachis hypogaea L.) mediante el empleo de marcadores microsatélites
Genetic characterization of cultivated peanut genetic resources (Arachis hypogaea L.) using microsatellite markers
Florencia Ileana Pozzi 1, Valeria Etchart 2, Daniel Díaz 2, Olegario Manuel Royo 3, Carolina Díaz 4, María Valeria Moreno 1, Jorge Omar Gieco 1
1 Laboratorio de Biotecnología, Estación Experimental Agropecuaria Manfredi, Instituto Nacional de Tecnología Agropecuaria (INTA), Ruta Nacional 9 Km. 636 (5988) Manfredi, Córdoba, Argentina. florenciapoz@hotmail.com
2 Laboratorio de Biología Molecular, Instituto de Genética "Ewald A. Favret", Centro de Investigación en Ciencias Veterinarias y Agronómicas (CICVyA), INTA, Los Reseros y Las Cabañas s/n (1686), Hurlingham, Buenos Aires, Argentina.
3 Área de Cultivos Extensivos, Estación Experimental Corrientes, INTA, Ruta 12 Km. 1008 (3400) Corrientes, Argentina.
4 Área de Estadística, Estación Experimental Agropecuaria Manfredi, INTA, Ruta Nacional 9 Km. 636 (5988) Manfredi, Córdoba, Argentina.
Originales: Recepción: 10/08/2012 - Aceptación: 12/06/2013
RESUMEN
El maní cultivado (Arachis hypogaea L.), es una especie de gran importancia económica, nativo de América del Sur. Se divide en dos subespecies y seis variedades botánicas. Genéticamente es alotetraploide, constituido por dos juegos genómicos duplicados. El empleo de marcadores microsatélites resulta más apropiado para realizar la caracterización genética de esta especie, puesto que permiten detectar un elevado nivel de polimorfismo. El objetivo del presente trabajo fue caracterizar la diversidad genética existente en las entradas de germoplasma de maní cultivado pertenecientes al Banco Activo de Germoplasma de Maní del Instituto Nacional de Tecnología Agropecuaria (INTA). Veinticinco entradas fueron genotipificadas con 23 marcadores microsatélites, de los cuales, 17 resultaron polimórficos. Se observaron 75 fragmentos polimórficos amplificados, con un promedio de 4,41 alelos por locus y un rango de 1 a 9 alelos. El contenido de información polimórfica osciló entre 0,15 y 0,58. El valor de la diversidad genética promedio fue de 0,165. Tanto el análisis de conglomerados como el de coordenadas principales evidenciaron dos grupos, uno formado por los materiales representantes de la subespecie fastigiata y otro por los de la subespecie hypogaea. Los resultados del análisis molecular de la varianza mostraron varianza tanto dentro como entre, las subespecies analizadas.
Palabras clave: Maní cultivado; Germoplasma; Microsatélite; Diversidad genética
ABSTRACT
Cultivated peanut (Arachis hypogaea L.) is a crop species of great economic importance, native to South America. It is divided into two subspecies and six botanical varieties. Genetically is an allotetraploid with two duplicated genomes. For genetic characterization of this specie, microsatellite marker would be the most appropriate. The aim of this study was to characterize the genetic variation among cultivated peanut genetic resources belonging to the Active Peanut Germplasm Bank of the Instituto Nacional de Tecnología Agropecuaria (INTA). Twenty five accessions were genotyped with 23 microsatellite markers, of which 17 were polymorphic. Seventy five polymorphic amplified fragments were observed with an average of 4.41 alleles per locus and a range of 1 to 9 alleles. The polymorphic information content was ranged from 0.15 to 0.58. The value of average genetic diversity was 0.165. Cluster analysis and principal coordinate analysis showed two groups, one for representative accessions of fastigiata subspecie and another of the hypogaea. Analysis of molecular variance results showed variance within and between subspecies.
Keywords: Cultivated peanut; Germplasm; Microsatellite; Genetic diversity
INTRODUCCIÓN
Arachis hypogaea L. (A. hypogaea), conocida comúnmente como maní cultivado, es una especie americana domesticada por el hombre. Según antecedentes previos (11, 12, 33), sería originaria del territorio correspondiente en la actualidad al Sureste de Bolivia y al Noroeste de Argentina. Una vez originada la especie, ésta habría sido distribuida por el hombre hacia todas las partes cálidas y bajas del neotrópico. Posteriormente, fue introducida en África por exploradores portugueses y, en Asia y Europa, por colonizadores españoles (11, 30). A. hypogaea es una especie alotetraploide (2n= 4x= 40 cromosomas), formada por el cruzamiento entre A. ipaënsis (donadora del genoma BB) y A. duranensis (donadora del genoma AA) (22, 27). Krapovickas y Gregory (23) consideraron dos subespecies (subesp.): fastigiata e hypogaea, las cuales a su vez agruparon en seis variedades botánicas para representar la diversidad infra-específica. De este modo, la subesp. fastigiata incluye 4 variedades (var. fastigiata, var. vulgaris, var. peruviana y var. aequatoriana), mientras que la subesp. hypogaea está constituida por 2 variedades (var. hypogaea y var. hirsuta) (23).
El maní es uno de los cultivos de origen sudamericano con mayor impacto a nivel mundial. China e India son sus principales productores y consumidores, siendo cultivada principalmente para el consumo humano (16, 18, 33). Su semilla es una fuente importante de aceite y proteínas, por lo que es utilizada para la producción de aceite refinado, manteca y productos de confitería. La producción argentina de maní se exporta casi en su totalidad, puesto que se caracteriza por una alta calidad e inocuidad de producto. Con respecto a la distribución del cultivo por provincias, en el Centro-Sur de Córdoba se concentra el 96% del total producido nacionalmente, el resto se cultiva en la provincias de Salta, Jujuy, Santa Fe, Corrientes, San Luis, La Pampa, Chaco y Formosa (25).
A. hypogaea presenta una reducida diversidad genética, lo cual se ve reflejado en el bajo nivel de polimorfismo genético detectado en la especie. Esta disminución pudo ser causada por diversos factores, entre ellos: la naturaleza autógama de la especie; el hecho de que se haya originado a partir de uno o pocos eventos de hibridación relativamente recientes en términos evolutivos; la aplicación de esquemas selectivos rigurosos y el apareamiento de individuos superiores genéticamente relacionados y la disminución de diversidad genética presente en especies vegetales silvestres a causa del deterioro de los ecosistemas naturales por la creciente actividad humana (19). Todos los factores mencionados condujeron a un estrechamiento de la base genética del cultivo, dificultando así su mejoramiento (8, 27). La utilidad de una colección de germoplasma depende de la caracterización disponible, la cual permite un mejor aprovechamiento de la diversidad genética y un uso eficiente de los recursos (Tomas et al., 2013). De este modo, a través de la utilización de los recursos genéticos disponibles en los bancos de germoplasma, se podría maximizar el uso de la variabilidad genética existente dado que constituyen un reservorio de genes útiles y deseables para el mejoramiento de la especie. En este sentido, el Banco Activo de Germoplasma de Maní (BAGM) de la Estación Experimental Agropecuaria Manfredi del Instituto Nacional de Tecnología Agropecuaria (INTA EEA Manfredi) almacena una colección de 40 especies de maní silvestre y 3.560 entradas de la especie cultivada (A. hypogaea). Esta colección es de sumo valor debido a su tamaño y a que la mayoría de las entradas fueron recolectadas en los centros de origen y de diversidad de la especie (30).
En el estudio de la variabilidad genética a través de marcadores moleculares, los microsatélites (Single Sequence Repeats: SSR) logran detectar más polimorfismos en maní que otros tipos de marcadores moleculares tales como RFLP (Restriction Fragment Length Polimorphism), RAPD (Random Amplified Polymorphic DNA) y AFLP (Amplified Fragment Lenght Polymorphism) (3, 11).
Objetivos
• Caracterizar la diversidad genética presente en entradas de maní cultivado del BAGM con buen comportamiento agronómico y representantes de las dos subespecies del cultivo mediante el empleo de SSR.
• Determinar la distribución de la variación genética dentro y entre las subespecies en estudio.
MATERIALES Y MÉTODOS
Se seleccionaron 25 entradas de maní cultivado pertenecientes al BAGM de INTA EEA Manfredi (tabla 1, pág. 4).
Tabla 1. Entradas de maní cultivado analizadas en el presente trabajo.
Table 1. Cultivated peanut accessions analyzed in the present study.
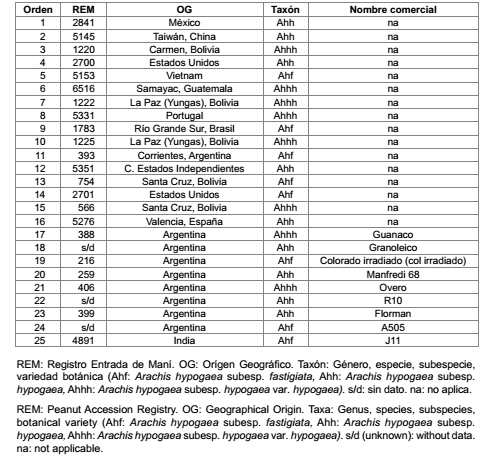
Esta selección se basó en el buen comportamiento agronómico de los materiales frente a Cercospora arachidicola, Cercosporidium personatum y Aspergillus spp., siendo representativas de las dos subespecies del cultivo y provenientes de diversos orígenes geográficos de muestreo (30).
La muestra de tejido vegetal se tomó a partir de 12 individuos de cada entrada analizada, conformando así un bulk por material. El número de individuos utilizados en el muestreo se tomó sobre la base de Fu (2003). Se eligieron aquellas hojas jóvenes sin lesiones, ni signos de ataque por patógenos. Dado que la especie en estudio es de reproducción autógama, se espera un elevado coeficiente de endogamia y por ende un bajo nivel de variabilidad genética entre los individuos de una misma entrada. Por ello, no se estimó la variabilidad interna dentro de los bulks.
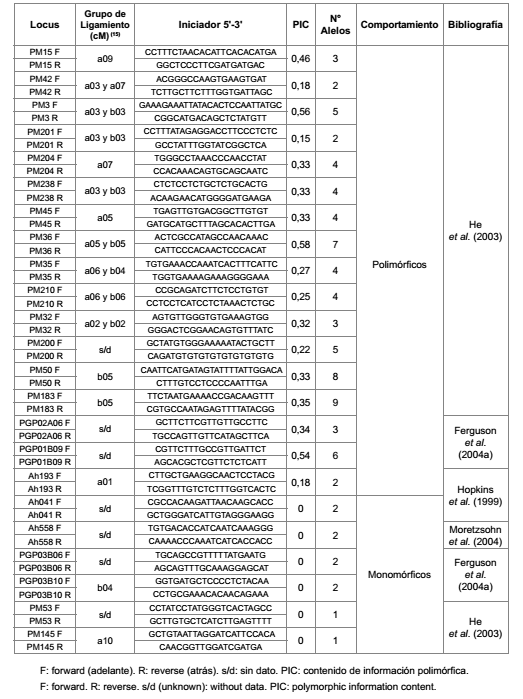
Las hojas frescas fueron liofilizadas y almacenadas en freezer a -80°C hasta su empleo para la extracción de ADN. La misma se realizó mediante el protocolo descripto por Doyle y Doyle (1987) modificado con respecto a la cantidad de material liofilizado a extraer, los tiempos de incubación y centrifugación y al volumen del buffer de extracción utilizado. La extracción de ADN de los materiales analizados fue corroborada mediante geles de agarosa al 0,8% (p/v) y la cuantificación se realizó empleando un fluorómetro Versaflúor (Biorad). Se emplearon 25 marcadores SSR para caracterizar el conjunto de materiales en estudio (tabla 2, pág. 5) (10, 15, 16, 18, 26).
Tabla 2. Descripción de los marcadores SSR utilizados en el análisis.
Table 2. Description of SSR markers used in this analysis.
Las reacciones de amplificación fueron efectuadas en un volumen final de 15 μL, conteniendo 1 μL de ADN (60 ng/μL), 0,5 unidades de Taq DNA polimerasa (Fermentas), 1X buffer de PCR (20 mM Tris-HCl pH 8,4 - 50mM KCl), 1,5 mM MgCl2, 0,2 mM de cada dNTP, 0,75 μL de iniciador forward y 0,75 μL de iniciador reverse en 9,78 μL de agua grado HPLC estéril.
Las amplificaciones fueron efectuadas en un termociclador GeneAmp System 9700 (Applied Biosystems). El programa de amplificación utilizado fue el descripto por He et al. (2003) con modificaciones en el rango de temperatura de 65- 56°C a 61- 56°C y en la rampa de descenso de temperatura durante la hibridación de 0,5°C/ciclo a 1°C/ciclo. Para los pares de iniciadores de PM36, PM42 y PM50, cuya temperatura de hibridación es de 50°C, la rampa de temperatura fue realizada desde 54°C. En los pares de iniciadores de Ah041, Ah193 y Ah558 se utilizó el programa descripto por Hopkins et al. (1999).
Los fragmentos amplificados fueron resueltos por electroforesis vertical en gel de poliacrilamida 6% (p/v) en una cuba Gibco-BRL (modelo S2). Los buffers de corrida utilizados fueron Tris-Borato-EDTA (TBE) 0,5X y TBE 1X, el tiempo de corrida fue de aproximadamente 2 horas a 50 Watts. En cada gel se incluyeron dos patrones moleculares de 10 pb y 25 pb, los geles fueron teñidos con kit de coloración con nitrato de plata de Promega (Madison, WI, EUA).
El análisis de los datos, consistió en el estudio descriptivo de: a) los marcadores SSR, y b) la diversidad genética presente en las entradas evaluadas. El primer análisis se basó en el grado de resolución de las bandas producidas por cada SSR, en el polimorfismo o monomorfismo de las mismas, en la proporción de loci polimórficos, en el número de alelos por locus y su promedio y en su contenido de información polimórfica (Polymorphic Information Content: PIC). El PIC por locus se calculó como:
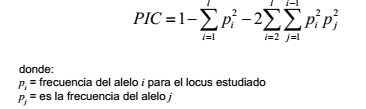
El análisis de la diversidad genética entre las diversas entradas de germoplasma de A. hypogaea se basó en la determinación de la riqueza alélica (R), de la diversidad genética (D) de cada entrada para múltiples loci, del número de alelos exclusivos y condiciones únicas. Éstas corresponden a una combinación alélica única detectada sólo en una entrada para un determinado marcador. La diversidad genética para múltiples loci se calculó mediante la fórmula:

Además se llevó a cabo un análisis de conglomerados basado en las distancias genéticas calculadas como (1-S)1/2 a partir del índice de similitud S de Dice (1946) y se construyó un dendrograma mediante el método de encadenamiento promedio (Unweighted Pair Group Method with Arithmetic Mean: UPGMA). Se realizó un análisis de coordenadas principales (ACoorP) y un análisis molecular de la varianza (AMOVA) a una vía de clasificación (factor subespecie) y empleando 3000 permutaciones para el cálculo del valor p asociado a cada término del modelo (9). Estas estimas fueron calculadas empleando el programa Infogen (2).
RESULTADOS Y DISCUSIÓN
Argentina se consolidó definitivamente como el primer exportador mundial de maní para consumo directo y algunas de sus manufacturas a partir del año 2010, donde el 90% del total exportado se produce y procesa en la provincia de Córdoba. Este avance se logró sobre la base de un proceso de mejoramiento continuo, sistemático y dinámico del cultivo que comenzó a fines de la década del 70 (4). De este modo, el conocimiento de la diversidad genética de las colecciones de germoplasma es de fundamental importancia para el desarrollo eficaz de los programas de mejoramiento, ya que proporciona un sistema de clasificación confiable y facilita la identificación de las entradas (5, 29, 31).
La caracterización de la diversidad genética presente en las entradas de maní cultivado estudiadas se realizó mediante el empleo de 25 marcadores SSR. De este número, dos de ellos (PM188 y PM65) fueron eliminados del análisis, dado que el primero no amplificó luego de tres experimentos sucesivos, mientras que PM65 mostró un patrón altamente confuso. Los 23 marcadores SSR restantes mostraron una resolución de bandas media y alta en gel de poliacrilamida al 6% (p/v). Del total de SSR empleados en la caracterización genética, 17 resultaron polimórficos y 6 fueron monomórficos, con una proporción de loci polimórficos de 73,91%. El total de loci analizados reveló 85 bandas totales de las cuales 75 resultaron polimórficas. De los 75, hubo un total de 15 alelos únicos (20 %).
El promedio de alelos por locus fue de 4,41 y el rango del número de alelos por locus registrado fue de 1 a 9 (tabla 2, pág. 5). Estos resultados fueron consistentes con los valores reportados por He et al. (2003) y por Moretzsohn et al. (2005). Los primeros autores informaron un promedio de alelos por locus de 4,25 y un número de alelos por locus entre 2 y 8; mientras que Moretzsohn et al. (2005) detectaron un promedio de alelos por locus de 5,87 y un número de alelos por locus entre 2 y 12. Estas coincidencias pueden deberse a que en ambos trabajos se utilizaron entradas de las dos subespecies y en número similar al empleado en este estudio.
Los valores de PIC oscilaron entre 0,15 y 0,58, para PM201 y PM36 respectivamente (tabla 2, pág. 5). Al comparar los resultados aquí obtenidos con los de Kottapalli et al. (2007) se observó que estos autores informaron límites superiores mayores para las estimas mencionadas, lo cual podría adjudicarse al análisis de un mayor número de entradas (72) y de SSR (entre 100 y 200). Esto conllevaría a la incorporación de una mayor variabilidad genética. Los marcadores PM50, PM183 y PM36 fueron los que mostraron mayor número de alelos, coincidente con lo informado por He et al. (2003), teniendo en cuenta la similitud del material vegetal de partida y el tipo de marcadores SSR (Serie PM) utilizados en el análisis (tabla 2, pág. 5).
Los marcadores PM36, PM3, PGP01B09 y PM15 fueron los más informativos al evidenciar los valores más elevados de PIC (tabla 2, pág. 5), coincidente con lo informado por Etchart et al. (2012) y Kottapalli et al. (2007). Por lo expuesto, estos 4 SSR deberían incluirse en el conjunto de marcadores a utilizar para un futuro estudio de mapeo por asociación, agregando más marcadores según los requisitos del análisis de estructura genética de la población.
El conjunto de entradas analizadas evidenció una estima de diversidad genética promedio de 0,165 (tabla 3), similar a lo reportado por Kottapalli et al. (2007) (0,18) para una mini colección núcleo de germoplasma de USDA y a Etchart et al. (2012) (0,22).
Tabla 3. Estimas de diversidad genética para todas las entradas de A. hypogaea y para cada subespecie.
Table 3. Summary statistics for all the accessions of A. hypogaea and each subspecies.

Estos resultados fueron consistentes de acuerdo con el tipo de reproducción autógama que caracteriza a la especie en estudio y al hecho de que la misma se haya originado a partir de una o pocas hibridaciones, relativamente recientes en términos evolutivos, erosionando así la diversidad genética del cultivo (28).
Resulta importante destacar que no se detectaron muestras duplicadas, es decir que el perfil genético detectado con cada uno de los marcadores empleados en el análisis fue exclusivo de cada entrada.
En términos de riqueza alélica (R) promedio para múltiples loci se evidenció que ambas subespecies mostraron valores similares (22,23 para hypogaea y 22,37 para fastigiata). En el 62,5% y 41,2% de las entradas de la subesp. fastigiata e hypogaea respectivamente, se registró el máximo valor de R (23 alelos por entrada para los 25 loci analizados).
En cuanto a D y al número de alelos promedio, los valores fueron similares entre ambas subespecies (tabla 3, pág. 8), coincidente con los resultados obtenidos por Kottapalli et al. (2007). Sólo 7 entradas presentaron alelos exclusivos, principalmente en la subesp. hypogaea que mostró un valor 4 veces mayor que la subesp. fastigiata. La misma tendencia fue detectada para las condiciones únicas, donde la subesp. hypogaea mostró un valor 3 veces mayor al de la subesp. fastigiata (datos no mostrados). Estos resultados se atribuyen al hecho de que el número de entradas representantes de la subesp. hypogaea fue mayor y que representó más diversidad de orígenes geográficos que la subesp. fastigiata (tabla 1, pág. 4).
La distancia genética entre pares de entradas calculada utilizando el índice de similitud de Dice varió entre 0,18 y 0,68, con una correlación cofenética altamente significativa (r = 0,955), evidenciando buena representación entre la matriz de similitud de los genotipos y el dendrograma obtenido. Éste mostró una división clara en dos grupos principales, lo cual permitió diferenciar los dos tipos botánicos (subesp. fastigiata y subesp. hypogaea). El grupo I fue conformado por las 8 entradas de la subesp. fastigiata analizadas y una entrada de la subesp. hypogaea (566). La inclusión de la entrada 566 a este grupo fue atribuida a una contaminación mecánica al momento de cosechar las semillas (Royo O. M., 2012, comunicación personal). El grupo II conglomeró las restantes entradas de la subesp. hypogaea (figura 1, pág. 10).
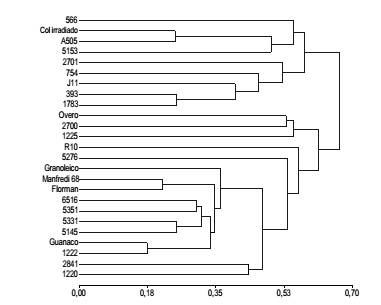
Figura 1. Dendrograma de las entradas analizadas sobre la base de la conversión en distancia del índice de similitud de Dice usando el agrupamiento UPGMA.
Figure 1. Dendrogram of accessions obtained using distances from Dice similarity index and UPGMA clustering.
Esta discriminación entre las dos subespecies fue coincidente con lo reportado por Etchart et al. (2012), Ferguson et al. (2004b), Kottapalli et al. (2007) y Moretzsohn et al. (2004). Se evidenció un alto grado de concordancia entre la clasificación morfológica en subespecies registrada en el Catálogo de Recursos Genéticos de Maní (30) y el agrupamiento basado en los resultados obtenidos de la caracterización genética.
Los resultados obtenidos en el análisis de coordenadas principales (ACoorP) mostraron la misma tendencia que el análisis de conglomerados, es decir, dos grupos claramente definidos sobre la base de la clasificación por subespecie. La coordenada principal 1 (CP1) explicó el 29,7% de la variabilidad total de las entradas estudiadas, evidenciando dos grupos base de eje vertical que separaron a las entradas en función de su clasificación por subespecie. La coordenada principal 2 (CP2) explicó el 12,5% de la variabilidad genética total de las entradas estudiadas. Cabe destacar que la entrada 566 mostró el mismo patrón de comportamiento que en el análisis de grupos, constituyendo parte del conglomerado que agrupó a las entradas de la subesp. fastigiata (figura 2, pág. 10).
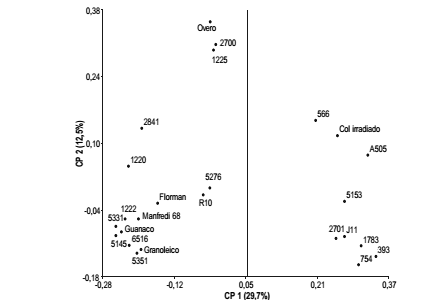
Figura 2. Diagrama de dispersión obtenido a partir del ACoorP utilizando la matriz de similitud de Dice entre las entradas analizadas.
Figure 2. Scatterplot obtained of PCoA analysis using Dice similarity matrix between accessions analyzed.
El porcentaje de variabilidad genética explicada por las dos primeras coordenadas (42,2%) fue similar a lo reportado en otras especies cultivadas (~ 50%) como girasol (14), trigo (1) y arroz (20).
La distinción entre subespecies a nivel morfológico se basa en el hábito de crecimiento erecto o rastrero, la presencia o ausencia de flores en el eje principal, la ramificación alternada o secuencial, la madurez temprana o tardía, los folíolos pequeños con pigmentación clara o folíolos grandes con pigmentación oscura y la ausencia o presencia de dormición en las semillas (11). Los resultados obtenidos en los análisis precedentes avalan la evidente división entre ambas subespecies, posiblemente justificada por la naturaleza autógama del maní que limitaría el flujo génico entre ellas.
El conocimiento de los niveles de variación genética, y su distribución dentro y entre poblaciones, resulta importante para comprender el origen y evolución de las mismas en condiciones naturales (17). Los resultados obtenidos en el AMOVA evidenciaron que existe variabilidad genética tanto, entre las subespecies, como dentro de ellas. La variación entre las subespecies explicó el 42,19% del total, mientras que la variación dentro de las mismas reveló el 57,81% restante, siendo altamente significativa para ambos casos (p<0,0001). El valor del coeficiente PhiST (Phi_subespecie) fue de 0,42, mostrando que a pesar de la variabilidad observada dentro de las poblaciones, el factor subespecie originó un patrón de estructuración genética indicando una mediana diferenciación entre las mismas (tabla 4).
Tabla 4. Resultados del AMOVA para las subespecies hypogaea y fastigiata.
Table 4. AMOVA results for hypogaea and fastigiata subspecies.
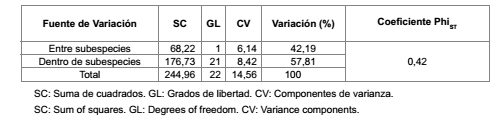
Estos resultados fueron similares a lo reportado por Mace et al. (2007) que informó una variación genética entre subespecies de 46,19% y dentro de ellas de 53,81%, con un PhiST de 0,46. Sobre la base de los resultados obtenidos en el presente estudio, para futuros muestreos se deberá considerar tanto la representatividad de las dos subespecies, como el número de entradas dentro de las mismas.
CONCLUSIONES
El presente estudio muestra la utilidad de los marcadores microsatélites para detectar polimorfismos y analizar las relaciones genéticas entre diversas entradas. De acuerdo con los resultados obtenidos, se concluye que los marcadores PM36, PM3, PGP01B09 y PM15 fueron los más adecuados en cuanto a su nivel de discriminación y detección de polimorfismo para constituir el panel de análisis en un futuro estudio de mapeo por asociación.
Siete entradas presentaron alelos exclusivos, de las cuales 5 pertenecen a la subespecie hypogaea. Se determinó un bajo nivel de diversidad genética promedio presente en las entradas estudiadas coincidente con lo reportado por la bibliografía, posiblemente debido al origen y al tipo de reproducción de la especie. Se detectó alto grado de concordancia entre la caracterización morfológica de las entradas y su respectivo análisis genético, observándose una clara división en dos grupos correspondientes a las subespecies analizadas.
En cuanto a la distribución de la variación genética, se determinó un nivel similar de variación dentro y entre subespecies, evidenciando la importancia de efectuar futuros muestreos que consideren la representatividad y el número de entradas de las dos subespecies estudiadas.
Este trabajo reviste suma importancia dado que constituye el primer paso para el estudio de la diversidad genética con marcadores moleculares en entradas del BAGM del INTA-EEA Manfredi para la futura implementación de mapeo por asociación en maní.
1. Akbari, M.; Wenzl, P.; Caig, V.; Carling, J.; Xia, L.; Yang, S.; Uszynski, G.; Mohler, V.; Lehmensiek, A.; Kuchel, H. 2006. Diversity arrays technology (DArT) for high-throughput profiling of the hexaploid wheat genome. Theoretical and Applied Genetics. 113(8): 1409-1420. [ Links ]
2. Balzarini, M.; Bruno, C.; Peña, A.; Teich, I.; Di Rienzo, J. 2010. Estadística en Biotecnología. Aplicaciones en Info-Gen. Encuentro Grupo Editor. Córdoba, Argentina. 227p. [ Links ]
3. Barkley, N. A.; Dean, R. E.; Pittman, R. N.; Wang, M. L.; Holbrook, C. C.; Pederson, G. A. 2007. Genetic diversity of cultivated and wild-type peanuts evaluated with M13-tailed SSR markers and sequencing. Genetic Resources. 89(2): 93-106. [ Links ]
4. Buteler, M. I. 2012. Cadena de valor del maní: hitos y desafíos. Ciencia y Tecnología de los Cultivos Industriales. 1(3): 195-196. [ Links ]
5. De la Vega, A. J.; De Lacy, I. H.; Chapman, S. C. 2007. Progress over 20 years of sunflower breeding in central Argentina. Field Crops Research. 100: 61-72. [ Links ]
6. Dice, L. R. 1945. Measures of the amount of ecologic association between species. Ecology. 26: 297-302. [ Links ]
7. Doyle, J. J.; Doyle, J. L. 1987. A rapid DNA isolation procedure for small quantities of fresh leaf tissue. Phytochemical Bulletin. 19: 11-15. [ Links ]
8. Etchart, V.; Pozzi, F. I.; Giustetti, M.; Moreno, M. V.; Royo, O.; Díaz, D.; Gieco, J. O. 2012. Estimación de la diversidad genética en germoplasma de maní (Arachis hypogaea) por medio de microsatélites. Ciencia y Tecnología de los Cultivos Industriales. 1(3): 243-249. [ Links ]
9. Excoffier, L.; Smouse, P.; Quattro, J. 1992. Analysis of molecular variance inferred from genetic distances among DNA haplotypes: application to human mitochondrial DNA restriction data. Genetics. 131: 479-491. [ Links ]
10. Ferguson, M. E.; Burow, M.; Schulze, S.; Bramel, P.; Paterson, A.; Kresovich, S.; Mitchell, S. 2004a. Microsatellite identification and characterization in peanut (A. hypogaea L.). Theoretical and Applied Genetics. 108: 1064-1070. [ Links ]
11. Ferguson, M. E.; Bramel, P.; Chandra, S. 2004b. Plant genetic resources: gene diversity among botanical varieties in peanut. Crop Science. 44: 1847-1854. [ Links ]
12. Fernandez, E. M.; Giayetto, O. 2006. Cultivo del maní en Córdoba. Ediciones de la Facultad de Agronomía y Veterinaria, Universidad Nacional de Río Cuarto, Córdoba, Argentina. 50 p. [ Links ]
13. Fu, Y. 2003. Applications of bulking in molecular characterization of plant germplasm: a critical review. Plant Genetic Resources: Characterization and utilization. 1(2-3): 161-167. [ Links ]
14. Garayalde, A. F.; Poverene, M.; Cantamutto, M.; Carrera, A. 2011. Wild sunflower diversity in Argentina revealed by ISSR and SSR markers: an approach for conservation and breeding programs. Annals of Applied Biology. 158: 305-317. [ Links ]
15. Gautami, B.; Foncéka, D.; Pandey, M. K.; Moretzsohn, M. C.; Sujay, V.; Qin, H.; Hong, Y.; Faye, I.; Chen, X.; Prakash, A. B.; Shah, T. M.; Gowda, M. V. C.; Nigam, S. N.; Liang, X.; Hoisington, D. A.; Guo, B.; Bertioli, D.J.; Jean-Francois Rami, JF.; Varshney, R.K. 2012. An international reference consensus genetic map with 897 marker loci based on 11 mapping populations for tetraploid groundnut (Arachis hypogaea L.) PLoS ONE 7(7): e41213. [ Links ]
16. He, G., Meng, R.; Newman, M.; Gao, G.; Pittman, R. N.; Prakash, C. S. 2003. Microsatellites as DNA markers in cultivated peanut (Arachis hipogaea L.). BMC Plant Biology. 3: 3. [ Links ]
17. Hernández Verdugo, S.; González Rodríguez, A.; Sánchez Peña, P.; Casas, A.; Oyama, K. 2006. Estructura y diferenciación genética de poblaciones silvestres y domesticadas de Chile del noroeste de México analizada con isoenzimas y RAPDs. Revista Fitotécnica Mexicana. 29(2): 25-29. [ Links ]
18. Hopkins, M. S.; Casa, A. M.; Wang, T.; Mitchell, S. E.; Dean, R.; Kochert, G. D.; Kresovich, S. 1999. Discovery and characterization of polymorphic simple sequence repeats (SSRs) in peanut. Crop Science. 39: 1243-1247. [ Links ]
19. Iriondo Alegría, J. M. 2001. Conservación de germoplasma de especies raras y amenazadas (Revisión). Invest. Agr.: Prod. Prot. Veg (ES). 16(1): 5-24. [ Links ]
20. Kojima, I.; Ebana, K., Fukuoka, S.; Nagamine, T.; Kawase, M. 2005. Development of RFLP-based rice diversity research set of germplasm. Breeding Science. 55: 431-440. [ Links ]
21. Kottapalli, K. R.; Burow, M. D.; Burow, G.; Burke, J.; Puppala, N. 2007. Molecular characterization of the US peanut mini core collection using microsatellite markers. Crop Science. 47: 1718-1725. [ Links ]
22. Krapovickas, A. 2004. Consideraciones sobre el origen del maní. IV Encuentro Latino americano de Especialistas en Arachis. Brasilia, DF, Brasil. [ Links ]
23. Krapovickas, A.; Gregory, W. C. 1994. Taxonomía del genero Arachis (Leguminosae). Bonplandia. 8(1-4): 1-186. [ Links ]
24. Mace, E. S.; Yuejin, W.; Boshou, L.; Upadhyaya, H.; Chandra, S; Crouch, J. H. 2007. Simple sequence repeat (SSR)-based diversity analysis of groundnut (Arachis hypogaea L.) germplasm resistant to bacterial wilt. Plant Genetic Resources: Characterization and Utilization. 5(1): 27-36. [ Links ]
25. Medrano, M. G. 2008. Caracterización agronómica de germoplasma de maní (Arachis hypogaea L.) con fines de premejoramiento. Tesis de Ingeniería Agronómica, Instituto de Ciencias Básicas y Aplicadas, Universidad Nacional de Villa María, Córdoba, Argentina. 60 p. [ Links ]
26. Moretzsohn, M. C.; Hopkins, M. S.; Mitchell, S. E.; Kresovich, S.; Valls, J. F. M.; Ferreira, M. E. 2004. Genetic diversity of peanut (Arachis hypogaea L.) and its wild relatives based on the analysisof hypervariable regions of the genome. BMC Plant Biology. 4: 11. [ Links ]
27. Moretzsohn, M. C.; Leoi, L.; Proite, K.; Guimarães, P. M.; Leal-Bertioli, S. C. M.; José, A. C. V. F.; Gimenes, M. A.; Martins, W. S.; Valls, J. F. M.; Grattapaglia, D.; Bertoli, D. J. 2005. A microsatellite-based, gene-rich linkage map for the AA genome of Arachis (fabaceae). Theoretical Applied Genetics. 111: 1060-1071. [ Links ]
28. Moretzsohn, M. C.; Leal-Bertioli, S. C. M.; Guimarães, P. M.; Proite, K.; José, A. C. V. F.; Fávero, A. P.; Gimenes, M. A.; Valls, J. F. M.; Bertoli, D. J. 2006. Mapeamiento genético em Arachis. V Encuentro Nacional de Especialistas en Arachis. Córdoba, Argentina. [ Links ]
29. Prada, D. 2009. Molecular population genetics and agronomic alleles in seed banks: searching for a needle in a haystack?. Journal of Experimental Botany. 60(9): 2541-2552. [ Links ]
30. Sanchez, R.; Baldessari, J.; Royo, O. M. 2010. Peanut Genetic Resources Catalogue 2010. Manfredi, Córdoba, Argentina. 150 p. [ Links ]
31. Tanksley, S. D.; McCouch, S. R. 1997. Seed banks and molecular maps: unlocking genetic potential from the wild. Science. 277:1063-1066. [ Links ]
32. Tomas, P. A.; Gottlieb, A. M.; Schrauf, G. E.; Poggio, L. 2013. Utilización de marcadores morfológicos y moleculares AFLP en la identificación de germoplasma nativo y cultivado de Elymus scabrifolius (Poaceae). Revista de la Facultad de Ciencias Agrarias. Universidad Nacional de Cuyo. Mendoza. Argentina. 45(2): 85-100. [ Links ]
33. Williams, D. E. 2006. La historia mundial del maní: ¿cuál será su futuro?. V Encuentro Nacional de Especialistas en Arachis. Córdoba, Argentina. [ Links ]
Agradecimientos
A la Lic. Gabriela Grandón y Lic. Yanina Alarcón por el soporte técnico en el trabajo de laboratorio.
Al Sr. Juan Peroglia por la implantación y mantenimiento de los ensayos de campo.
A la financiación recibida para llevar adelante este trabajo (Proyecto Nacional INTA: PNIND N° 081411).














