Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista FAVE. Sección Ciencias veterinarias
On-line version ISSN 2362-5589
FAVE, Secc. Cienc. vet. vol.15 no.1-2 Santa Fe Dec. 2016
ARTÍCULO ORIGINAL
Discriminación genotípica de serovariedades del serogrupo Icterohaemorrhagiae pertenecientes a Leptospira interrogans (Spirochaetales: Leptospiraceae) y el análisis de distancias génicas mediante coordenadas principales
Grune Loffler S*, Martinez ML, Romero GN, Brihuega BF
INTA Castelar, CICVyA, Instituto de Patobiología, Laboratorio de Leptospirosis
* Correspondencia: Sylvia Grune Loffler, INTA Castelar, CICVyA, Instituto de Patobiología, Laboratorio de Leptospirosis. De Los Reseros y Dr. Nicolás Repetto S/N,CC77 (1708) Hurlingham, Buenos Aires, Argentina.
E-mail: grune.sylvia@inta.gob.ar
Editor: P.M. Beldomenico
doi: https://doi.org/10.14409/favecv.v15i1/2.6243
Recibido: 18 Octubre 2016.
Aceptado: 2 Enero 2017.
Disponible en línea: 18 Enero 2017
RESUMEN
La leptospirosis es una zoonosis de amplia distribución mundial y de importancia en salud pública y veterinaria. El serogrupo Icterohaemorrhagiae toma importancia, ya que ha sido aislado con frecuencia en roedores y en casos clínicos de humanos, caninos, bovinos y porcinos; y se ha logrado aislar a partir de agua del rio Reconquista, provincia de Buenos Aires, Argentina. El objetivo de este estudio fue discriminar molecularmente entre las serovariedades Copenhageni e Icterohaemorrhagiae del serogrupo Icterohaemorrhagiae de importancia en Argentina, utilizando la técnica del análisis de repeticiones en tándem en múltiples locus (MLVA) y analizar las distancias génicas entre los genotipos determinados a partir de cepas aisladas de animales. Para ello, genotipificamos las 4 cepas referenciales del serogrupo Icterohaemorrhagiae de la especie Leptospira interrogans serovariedad Copenhageni cepa M20 y cepa Ictero I y de la serovariedad Copenhageni cepa RGA y cepa Fiocruz L1-130. Para este estudio se incluyeron 24 cepas aisladas de animales domésticos como silvestres. Logramos mediante esta técnica determinar un código numérico para cada serovariedad y así pudimos discriminar entre las serovariedades de este serogrupo. El análisis de coordenadas principales (PCoA) demostró la variabilidad génica existente entre las 4 cepas referenciales pertenecientes al serogrupo Icterohaemorrhagiae, el genotipo con mayor representación fue el serovar Copenhageni cepa Fiocruz L1-130 con un total de 13 cepas aisladas con ese perfil genético. Discriminar las serovariedades de este importante serogrupo nos permitirá en un futuro investigar si existen diferencias entre la sintomatología clínica y/o respuesta al tratamiento en los casos clínicos producidos por las distintas serovariedades del serogrupo Icterohaemorrhagiae discriminadas en este trabajo.
Palabras clave: Leptospira interrogans; Leptospirosis; Icterohaemorrhagiae; Tipificación molecular.
SUMMARY
Genotypic discrimination of serovars belonging to the serogroup Icterohaemorrhagiae of Leptospira interrogans (Spirochaetales: Leptospiraceae) and the analysis of genetic distances using principal coordinates
Leptospirosis is the world`s most widely distributed zoonosis and of great importance in animal health and in public health. The relevance of the serogroup Icterohaemorrhagiae relies on the frequent isolation from rodents and from clinical cases of humans, canines, bovines and swine; and this serogroup was also isolated from water samples of the Reconquista river, Buenos Aires province, Argentina. The objective of this study was to discriminate molecularly between serovars Copenhageni and Icterohaemorrhagiae being of importance in Argentina, using Multiple Locus Variable number tandem repeats analysis (MLVA) and analyzing the genetic distances among genotypes obtained from animal isolates in this study. We genotyped the four referential strains of the serogroup Icterohaemorrhagiae belonging to the species Leptospira interrogans serovar Copenhageni strain M20 and strain Ictero I and of the serovar Copenhageni strain RGA and strain Fiocruz L1-130. In this study we included 24 strains isolated from animals domestic and wildlife. Using this technique we could determine numeric codes for each serovar and in this way, discriminate between serovars of this serogroup. The principal coordinates analysis (PCoA) showed the genetic variability of the four referential strains from the serogroup Icterohaemorrhagiae, the genotype with better representation was serovar Copenhageni strain Fiocruz L1-130 with 13 isolated strains with this genetic profile. Discrimination of the serovars from this important serogroup allows to establish differences between clinical signs and/or response to treatment in the clinical cases produced by different serovars of the serogroup Icterohaemorrhagiae successfully distinguished in this study.
Key words: Leptospira interrogans; Leptospirosis; Icterohaemorrhagiae; Molecular characterization.
Introducción
La leptospirosis es una enfermedad infectocontagiosa de relevante importancia en la salud pública y es considerada la zoonosis de mayor distribución mundial. Es causada por las cepas patógenas del género Leptospira spp., Familia Leptospiraceae, Orden Spirochaetales. Esta enfermedad puede ser trasmitida al hombre principalmente por inadecuado manejo sanitario del medio ambiente contaminado por animales de producción, silvestres y mascotas. El punto de partida para la diseminación de la leptospirosis es la presencia de un portador (Stanchi et al. 2006). Estos, ya sean animales domésticos o silvestres, eliminan Leptospiras por la orina en forma discontinua y por períodos de tiempo variables. De esta manera se efectiviza la infección directa a otros animales, de la misma u otras especie, como así también al hombre (Faine, 1982). No existen datos sobre la incidencia de esta enfermedad en muchos países, siendo poco reconocida y por ende severamente descuidada, estando dentro del grupo de enfermedades olvidadas (Hartskeerl et al., 2011).
En Argentina, se han reportado en el presente año (2016) (hasta la semana epidemiológica 22), 2111 casos notificados y se han confirmado 60 casos de leptospirosis humana, han aumentado un 69.42% los casos notificados con respecto al año 2014 (Ministerio de Salud, 2016). Se debe considerar que esta enfermedad muchas veces es mal diagnosticada y confundida con otras enfermedades febriles como el dengue y la meningitis. En la ganadería esta zoonosis toma relevancia ya que se estima que un 10% de los abortos bovinos anuales en el país son producidos por la leptospirosis. En el año 2009 reportaron la muerte de 100 terneros de recría originado por Leptospira interrogans serovar Pomona (Draghi et al., 2011). En Argentina se han logrado aislar cepas de Leptospira spp. y tipificar como pertenecientes al serogrupo Icterohaemorrhagiae en casos clínicos humanos (Seijo et al., 2002), en casos de abortos en porcinos (Pavan et al., 2011; Rossetti et al., 2002) y de casos clínicos caninos (Grune et al., 2014). Se logró aislar una cepa perteneciente a este serogrupo en el rio Reconquista, Provincia de Buenos Aires, Argentina, zona altamente poblada con el riesgo que esto implica a la población adyacente (Brihuega et al., 2006).
Diferentes especies de roedores han sido descriptas como reservorios de diversas serovariedades de Leptospira spp. ( Hartskeerl et al., 2011; Grune et al., 2014a; Levett et al., 2010; Scialfa et al., 2010). Las ratas son frecuentemente reservorio de las serovariedades Icterohaemorrhagiae y Copenhageni, del serogrupo Icterohaemorrhagiae de la especie Leptospira interrogans, mientras las Leptospiras aisladas de ratones pertenecen al serogrupo Ballum de la especie L. borgpetersenii. Hartskeerl et al., (2011) expone que los roedores son los reservorios principales de las serovariedades patogénicas y altamente virulentas y que es esta la razón por la cual las Leptospiras (y por ende la leptospirosis) no podrán ser erradicadas.
Otros trabajos reportaron leptospirosis en poblaciones de roedores en áreas urbanas y suburbanas de Argentina sin poder discriminar las serovariedades del serogrupo Icterohaemorrhagiae (Scialfa et al., 2010, Grune et al., 2014a). En un trabajo publicado en el 2002 por Seijo, se detallan serovares pertenecientes al serogrupo Icterohaemorrhagiae (sin poder ser discriminados) en caninos, ratas, ratones, armadillos, y porcinos. En el mismo trabajo se presenta un tabla con aislamientos de cepas de Leptospira interrogans del serogrupo Icterohaemorrhagiae desde 19342001, sería de gran utilidad poder aplicar la caracterización molecular por MLVA a estas cepas.
Grune et al., (2014a) tipificaron 22 cepas mediante la técnica del análisis de repeticiones en tándem en múltiples locus (MLVA), encontrando genotipos pertenecientes a L. interrrogans y L. borgpetersenii proveniente de aislamientos de riñón de roedores en Argentina. Los perfiles obtenidos del serogrupo Icterohaemorrhagiae (2,1,13,7,2,0,3) de la especie Leptospira interrogans, no se logró discriminar entre las serovariedades Copenhageni e Icterohaemorrhagiae utilizando los primer propuestos por Majed et al., (2005), flanqueando los loci (VNTR4, VNTR7, VNTR9, VNTR10, VNTR19, VNTR23, VNTR31). Esta dificultad también se presenta utilizando otros métodos de tipificación molecular como es el caso del PFGE, el cual tampoco logra discriminar entre las serovariedades Icterohaemorrhagiae y Copenhageni de L. interrogans (Galloway et al., 2008).
El objetivo de este estudio fue discriminar entre las serovariedades Copenhageni e Icterohaemorrhagiae y analizar las distancias génicas entre los genotipos determinados a partir de cepas aisladas de animales mediante el análisis de coordenadas principales.
Materiales y Métodos
En este trabajo se tipificaron molecularmente las cepas referenciales pertenecientes al serogrupo Icterohaemorrhagiae de la especie Leptospira interrogans presentes en Argentina. Las cepas incluidas en este estudio pertenecen al cepario del Laboratorio de Leptospirosis (Centro de Referencia de la OIE), ubicado en el Instituto de Patobiología, Centro de Investigación en Ciencias Veterinarias y Agronómicas (CICyA), perteneciente al Instituto Nacional de Tecnología Agropecuaria (INTA). Se incluyeron en este estudio, 24 cepas aisladas de animales y pertenecientes al cepario del laboratorio: 19 cepas aisladas de ratas (Rattus rattus) cepas denominadas: Rat1A, Rat2A, Rat3A, Rat5, Rat6, Rat8, Rat9, Rat13, Rat17, Rat101,Rat102, Rat103, Rat298, Rat302, Rat410, Rat451, Rat454, Rat478 y Rat495; 1 cepa denominada ardilla aislada de ardilla (Callosciurus erythraeus), 1 cepa aislada de un canino denominada cepa Avellaneda), 2 cepas aisladas de comadrejas (Didelphis albiventris) denominadas cepa Com3 y Com4 y 1 cepa aislada de ratón (Oligoryzomys nigripes) denominada cepa Mus320. Dentro de la serovariedad Copenhageni, la cepa Fiocruz L1-130 y la cepa M 20; y dentro de la serovariedad Icterohaemorrhagiae se tipificaron la cepa RGA y la cepa Ictero I. Las cepas referenciales fueron mantenidas en medio semisólido Fletcher (Laboratorios Difco, Becton Dickinson Company, NL, Estados Unidos) y mantenidas a 28°C.
Para la extracción de ADN se incubaron alícuotas de 100 μl en un termociclador durante 10 minutos a 100°C. La discriminación de las serovariedades del serogrupo Icterohaemorrhagiae se realizó utilizando los primers propuestos por Salaün et al., (2006) para flanquear los loci:
VNTR4 (4a_bis AAGTAAAAGCGCTCCCAAGA; 4b_bis (ATAAAGGAAGCTCGGCGTTT);
VNTR7 (7a_bis GATGATCCCAGAGAGTACCG; 7b TCCCTCCACAGGTTGTCTTG);
VNTR10 (10a_bis GAGTTCAGAAGAGACAAAAGC; 10b_bis ACGTATCTTCATATTCTTTGCG);
VNTRLb4 (Lb4a AAGAAGATGATGGTAGAGACG; Lb4b ATTGCGAAACCAGATTTCCAC);
y VNTRLb5 (Lb5a AGCGAGTTCGCCTACTTGC; Lb5b ATAAGACGATCAAGGAAACG).
De esta manera y mediante el uso de estos loci (VNTR- 4, -7, -10, -Lb4, -Lb5) se podrá discriminar dentro del serogrupo Icterohaemorrhagiae el perfil genético de las distintas cepas referenciales de Icterohaemorrhagiae.
El volumen final de reacción fue de 50 μl y cada reacción de PCR contenía Buffer (20mM Tris-HCL,pH 8.4;50mM KCL), 200μM dNTPs, 2μM de cada primer, 2mM MgCl2, 1.25U Taq DNA polimerasa (Invitrogen, California, Estados Unidos) y 5μl de templado de ADN. El programa de ciclado fue 94°C durante 5 min, seguido de 35 ciclos de 94°C por 30 segundos, 55°C durante 30 segundos, 72°C durante 90 segundos con una extensión final de 10 minutos a 72°C. Los productos obtenidos (15 μl) se corrieron en un gel de agarosa al 2% en buffer TAE 1X (40mM Trisacetato, 1mM EDTA) y teñido con bromuro de etidio 0,2 μl μg/ml durante 60 minutos a 100V. El tamaño de los amplicones fue visualizado en un transiluminador de luz UV de cabina cubierta (Uvi Tec modelo BTS-20.M, Cambridge, Inglaterra). Para ver el tamaño de las bandas obtenidas se corrió el gel con un marcador de cien pares de base (pb) y se utilizó el programa GelAnalyzer 2010ª (http://www.gelanalyzer.com), para determinar con exactitud los tamaños de las bandas. Una vez obtenidos los tamaños de las bandas de las amplificaciones se debe determinar la cantidad de repeticiones o números de copia presente en la misma. Se aplicó la siguiente fórmula:
Número de repeticiones (pb) = [Tamaño de fragmento (pb) - Regiones de flanqueo (pb)] / Tamaño de repetición (pb)
Una vez obtenidos los cálculos del número de repeticiones de los geles sembrados, se procedió con la determinación de los genotipos del serogrupo Icterohaemorrhagiae obtenidos. Con el número de copias por cada locus (VNTR) flanqueado, se comparó con los perfiles del serogrupo Icterohaemorrhagiae (Tabla 1). Se realizó el análisis de distancia genética utilizando el complemento de Excel GenAlEx 6 (Genetic analysis in Excel. Population genetic software for teaching and research, http://biology.anu.edu.au/GenAlEx/Welcome.html), una vez obtenida la distancia entre los genotipos encontrados en el serogrupo Icterohaemorrhagiae se realizó el análisis de coordinadas principales (PCoA).
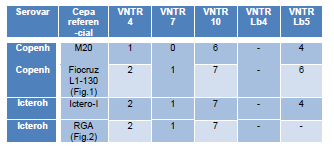
Tabla 1: Repeticiones obtenidas de las cuatro cepas referenciales del serogrupo Icterohaemorrhagiae presentes en Argentina mediante MLVA
El resultado que arroja el PCoA es una representación grafica en cuadrantes de la distribución espacial de los genotipos analizados dependiendo de la variabilidad génica representada en distancias.
Esta matriz de distancia triangular elaborada previamente utilizando el complemento de GenAlEx 6 se procesó con el programa MEGA 5.2 (Molecular Evolutionary Genetics Analysis,
http://www.megasoftware.net/) para la construcción de los árboles filogenéticos. El apareamiento de los genotipos se hizo basado en el método de UPGMA, el cual agrupa los genotipos por afinidad genética y la cercanía entre sí, formando clados que representan las cepas agrupadas con respecto a los genotipos.
Resultados
Con el número de repeticiones o copias determinado (alelos) para cada VNTR flanqueado (4, 7, 10, Lb4, Lb5) se obtuvo un código numérico de cada cepa genotipificada. Este código representa el genotipo de la cepa y corresponde al de una cepa referencial. En este estudio, presentamos 4 códigos que discriminan las cepas referenciales presentes en Argentina pertenecientes al serogrupo Icterohaemorrhagiae (Tabla 1). En la Figura 1, se pueden observar las cuatro figuras con los geles que reflejan el perfil del genotipo serovar Copenhageni cepa Fiocruz L1-130 (Fig.1a), el perfil del genotipo serovar Icterohaemorrhagiae cepa RGA (Fig.1b), el perfil del genotipo serovar Copenhageni M20 (Fig.1c) y el perfil del genotipo serovar Icterohaemorrhagiae Ictero-I (Fig.1d); ambas serovariedades pertenecen la especie L. interrogans. En la Figura 2, se pueden observar los resultados obtenidos del análisis por coordenadas principales, en el cual se reflejan los cuatro genotipos pertenecientes a este serogrupo. El genotipo Fiocruz L1-130 (serovar Copenhageni) se encuentra en el lado izquierdo de la imagen (parte inferior del 1er cuadrante), este genotipo fue el mejor representado con 13 cepas. El segundo genotipo mejor representado es el de Ictero I (serovar Icterohaemorrhagiae) con 10 cepas genotipificadas con este perfil genético. En la parte central del gráfico, se puede observar el genotipo RGA representada con una cepa con este perfil (cepa Avellaneda aislada de un caso clínico canino). No pudimos encontrar cepas con el perfil del genotipo de M20 (serovar Copenhageni) y este genotipo se encuentra muy distante en el cuadrante superior derecho La comparación de los códigos de los genotipos introducidos en este análisis con respecto a su ubicación en el gráfico de coordenadas principales, nos brinda información adicional de relevancia en cuanto a su variabilidad génica. Es decir, los genotipos Fiocruz L1-130 y RGA se encuentran más cercanos a pesar de pertenecer a serovares diferentes, pero cuando observamos los alelos (número de repeticiones o copias por locus flanqueado) en el locus VNTR Lb5, vemos que en el genotipo de Fiocruz existen 6 copias y en el genotipo de RGA 0 copias en el mismo locus. En cuanto al análisis del árbol filogenético por UPGMA (Fig. 3), podemos observar algo similar, en la parte superior del árbol se ubica el serovar Copenhageni representado por la cepa Fiocruz L1-130 y en la parte media del árbol se ubica el genotipo RGA perteneciente al serovar Icterohaemorrhagiae. En la parte inferior del árbol se encuentra el genotipo Ictero I perteneciente al mismo serovar que RGA y el genotipo M20. Este último genotipo perteneciente al serovar Copenhageni representa diferentes números de alelos en los VNTR 4, 7 y 10 con respecto a los otros tres genotipos. Esto nos indica que este genotipo se ubica muy distante al resto (RGA, Ictero-I y Fiocruz L1-1-30) como también nos da información sobre el alto poder discriminativo del VNTR Lb5, ya que con poca varianza en los alelos determinados es capaz de separar los genotipos similares.

Figura 1. Corridas electroforéticas de Leptospira interrogans del serogrupo Icterohaemorrhagiae, de las siguientes procedencias: a) serovar Copenhageni Fiocruz L1-130 (2,1,7,-,6); b) serovar Icterohaemorrhagiae RGA ( 2,1,7,-,-); c) serovar Copenhageni M20 (1,0,6,-,4); d) serovar Icterohaemorrhagiae Ictero-I ( 2,1,7,-,4).
CM= Cien Mark. Los números sobre las barras representan los pares de bases y entre paréntesis se muestran los números de copias.

Figura 2: Análisis de Coordenadas Principales (PCoA) obtenido con los genotipos de las cepas provenientes de aislamientos animales pertenecientes al serogrupo Icterohaemorrhagiae. Cada punto azul representa un genotipo y subrayado se encuentra en nombre del genotipo. En rojo podemos ver las cepas aisladas de ratas, en naranja las cepas aisladas de comadrejas, en violeta la cepa aislada de un canino, en verde la cepa aislada de una ardilla y en celestre la cepa aislada de un ratón.

Figura 3: Árbol filogenético por UPGMA de los genotipos representados por Leptospira interrogans serogrupo Icterohaemorrhagiae serovar Copenhageni Fiocruz L1-130, serovar Copenhageni M20, serovar Icterohaemorrhagiae RGA e Ictero I. Las cepas de referencia se encuentran al principio de cada clado y se presenta entre paréntesis, el número de repeticiones de los VNTRs: (4bis, 7bis, 10bis, Lb4 y Lb5).
Discusión
En los casos clínicos de leptospirosis en los que se logra aislar y tipificar las cepas, en ocasiones, no se logran discriminar los aislamientos pertenecientes al serogrupo Icterohaemorrhagiae y sus serovariedades (Levett et al., 2006; Scialfa et al., 2010; Galloway et al., 2008; Majed et al., 2005; Pavan et al., 2011). En este estudio hemos logrado discriminar entre las serovariedades Copenhageni e Icterohaemorrhagiae flanqueando los VNTR propuestos por Salaün et al., (2006): VNTR4, VNTR7, VNTR10, VNTRLb4, VNTRLb5 y hemos comprobado la variabilidad genética entre las cuatro cepas referenciales mediante el análisis filogenético y el análisis de coordenadas principales. Discriminar las serovariedades y los genotipos de este importante serogrupo nos permitirá en un futuro investigar si existen diferencias entre la sintomatología clínica y/o respuesta al tratamiento en los casos clínicos producidos por las distintas serovariedades del serogrupo Icterohaemorrhagiae discriminadas en este trabajo. Este estudio brinda suficientes herramientas de discriminación como para poder ser aplicadas en trabajos posteriores con cepas de Leptospira aisladas tanto en casos humanos como en casos clínicos animales. Conocer el poder discriminativo de los VNTR elegidos para caracterizar genotípicamente cepas patógenas pertenecientes al género Leptospira spp., es importante ya que puede reducir costos y esfuerzo. Una herramienta útil y veraz de genotipificación se encuentra detallada en este trabajo, ya que una conclusión importante es que el VNTR Lb5 tiene un alto poder discriminativo, ya que es capaz de separar genotipos similares en los alelos obtenidos en los VNTR 4,7 y 10. En un trabajo comparativo sobre VNTRs y MLST (Caimi et al., 2012) se representa como genotipo "a" un aislamiento denominado La Cava 1, el cual se encuentra agrupado con las cepas referenciales Ictero-1, RGA y Fiocruz L1-130. Utilizando los VNTR descriptos originalmente por Majed et al., 2005, no se lograron discriminar entre las serovariedades del serogurpo Icterohaemorrhagiae.
En trabajos recientes, se discute también la posibilidad de que el VNTR Lb5 tenga alto poder discriminativo, coincidiendo con los resultados de este trabajo (Picardeau et al., 2014, Naze et al., 2015). Sin embargo, este método presenta algunas desventajas, una es que no se puede aplicar sobre ADN extraído de muestras clínicas, ya que requiere de ADN de alta calidad y mucha cantidad. Si se aplica sobre cultivos de cepas aisladas, éstas deben cumplir las mismas condiciones. Hasta los momentos, este método es una herramienta molecular con alto poder discriminativo para caracterizar cepas con perfiles genéticos similares y sin el uso de la secuenciación.
Agradecimientos
Este trabajo fue financiado por el Proyecto Nacional de Sanidad Animal (PNSA-1115052). A Carmelo Auteri por su valiosa colaboración en el mantenimiento de las cepas de Leptospira spp.
Bibliografía
1. Brihuega B, Auteri C, Romero G, Samartino L. 2006. Aislamiento de una cepa patógena de Leptospira de un río urbano: Respuesta frente a quinolonas fluoradas. Rev Med Vet. 87: 144-146. [ Links ]
2. Caimi K, Varni V, Melendez Y, Koval A, Brihuega B, Ruybal. 2012. A combined approach of VNTR and MLST analysis: improving molecular typing of Argentinean isolates of Leptospira interrogans. Mem. Inst. Oswaldo Cruz 107: 644-651. [ Links ]
3. Draghi MG, Brihuega B, Benítez D, Sala JM, Biotti GM, Pereyra M, Homse A, Guariniello L. 2011. Brote de leptospirosis en terneros en recría en la provincia de Corrientes, Argentina. Rev. Arg. Microbiol 43: 42-44. [ Links ]
4. Faine S. 1982. Guidelines for the control of leptospirosis, WHO offset publication N° 67. World Health Organization, Geneva, 161 pp. [ Links ]
5. Galloway RL y Levett PN. 2008. Evaluation of a modified pulsed-field gel electrophoresis approach for the identification of Leptospira serovars. Am. J. Trop. Med. Hyg. 78: 628-32. [ Links ]
6. Grune Loffler S, Pavan ME, Vanasco B, Samartino L, Suarez O,Auteri C, Romero G, Brihuega B. 2014a. Genotypes of pathogenic Leptospira spp. isolated from rodents in Argentina. Mem. Inst. Oswaldo Cruz 109:163-167. [ Links ]
7. Grune Loffler S, Passaro D, Samartino L, Soncini A, Romero G, Brihuega B. 2014b. Genotypes of Leptospira spp. strains isolated from dogs in Buenos Aires, Argentina. Rev. Arg. Microbiol. 46: 1-13. [ Links ]
8. Hartskeerl RA, Collares-Pereira M, Ellis WA. 2011. Emergence, control and re-emerging leptospirosis: dynamics of infection in the changing world. Clin. Microbiol. Infect. 17: 494-501. [ Links ]
9. Levett PN D.A. Haake. 2010. Leptospira species (leptospirosis). In: Mandell GL, Bennett JE, Dolin R, editors. Principles and Practice of Infectious Diseases. Philadelphia: Churchill Livingtsone, Elsevier. Pp. 3059-3065 [ Links ]
10. Majed Z, Bellenger E, Postic D, Pourcel C, Baranton G, Picardeau M. 2005. Identification of variable-number tandem-repeat loci in Leptospira interrogans sensu stricto. J. Clin. Microbiol. 43: 539-45. [ Links ]
11. Ministerio de Salud. Presidencia de la Nación. Boletín Integrado de Vigilancia 2016; Nº 312-SE22- Junio 2016. 107 pp. [ Links ]
12. Pavan ME, Cairó F, Pettinari MJ, Samartino L, Brihuega B. 2011. Genotyping of Leptospira interrogans strains from Argentina by Multiple-Locus Variable-number tandem repeat Analysis (MLVA). Comp. Immunol. Microbiol. Infect. Dis. 34: 135-41. [ Links ]
13. Peakall R, Smouse PE. 2006. GENALEX 6: genetic analysis in Excel. Population genetic software for teaching and research. Mol. Ecol. Notes. 6: 288-295. [ Links ]
14. Picardeau M, Bertherat E, Jancloes M, Skouloudis AN, Durski K, Hartskeerl RA. 2014. Rapid tests for diagnosis of leptospirosis: Current tools and emerging technologies. Diagn. Microbiol. Inf. Dis. 78: 1-8. [ Links ]
15. Naze F, Desvaís A, Picardeau M, Bourghy P, Michault A. 2015. Use of a New High Resolution Melting Method for Genotyping Pathogenic Leptospira spp.. PLoSONE 10:e0127430. [ Links ]
16. Rossetti C, Brihuega B, Auteri C, Romero G. 2002. Leptospirosis porcina en la República Argentina: Encuesta serológica y primera comunicación del aislamiento de una cepa de Leptospira interrogans (sg. Icterohaemorragiae) de una cerda abortada. Rev. Vet. Arg. 17:578-582. [ Links ]
17. Salaün L, Mérien F, Gurianova S, Baranton G, Picardeau M. 2006. Application of Multilocus Variable-Number Tandem-Repeat Analysis for Molecular Typing of the Agent of Leptospirosis. J. Clin. Microbiol. 44: 3954-3962. [ Links ]
18. Scialfa E, Bolpe J, Bardon J, Ridao G, Gentile J, Gallicchio O. 2010. Isolation of Leptospira interrogans from suburban rats in Tandil, Buenos Aires. Rev. Arg. Microbiol. 42: 126-128. [ Links ]
19. Seijo A. 2002. Informe sobre Leptospirosis en la República Argentina. Serie Enfermedades Transmisibles. Publicación Monográfica 3. Fundación Mundo Sano. Pp.:7-28. [ Links ]
20. Seijo A, Coto H, San Juan J, Videla J, Deodato B, Cernigoi B, Messina OG, Collia O, de Bassadoni D, Schtirbu R, Olenchuk A, de Mazzonelli GD, Parma A. 2002. Lethal leptospiral pulmonary hemorrhage: an emerging disease in Buenos Aires, Argentina. Emerg. Infect. Dis. 8:1004-1005. [ Links ]
21. Stanchi N, Brihuega B, Gatti E. Leptospirosis. En: Stanchi N, Martino P, Gentilini G, Reinoso Echeverría M, Leardini N, Copes JA. 2007. Microbiología Veterinaria. Intermédica, Buenos Aires, Argentina. Pp. 320-325. [ Links ]
22. Tamura K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S. 2011. MEGA 5 : Molecular Evolutionary Genetics Analysis using maximum Likelihood, Evolutionary Distance, and Maximum Parsimony Methods. Mol. Biol. Evol. 28: 2731-2739. [ Links ]














