Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista FAVE. Sección Ciencias veterinarias
versión On-line ISSN 2362-5589
FAVE, Secc. Cienc. vet. vol.18 no.2 Santa Fe dic. 2019
COMUNICACIÓN CORTA
Comparación de métodos de extracción de ADN simples y económicos para el diagnóstico molecular de leptospirosis animal
Hamer M1*, Saraullo V1,2, Brihuega B1, Watanabe O1, Martinez M1, Grune Loffler S1,2
1 Laboratorio de Leptospirosis, Instituto de Patobiología, CICVyA, Instituto Nacional de Tecnología Agropecuaria (INTA), Argentina.
2 Consejo Nacional de Investigaciones Científicas y Técnicas (CONICET), Argentina.
* Correspondencia: Sylvia Grune Loffler, INTA Castelar, Nicolás Repetto y De Los Reseros s/n, Hurlingham, Argentina.
E-mail: grune.sylvia@inta.gob.ar
Editor: C. Baravalle
doi: https://doi.org/10.14409/favecv.v18i2.8752
Recibido: 28 Agosto 2019.
Aceptado: 20 Noviembre 2019.
Disponible en línea: 3 Diciembre 2019
RESUMEN
La leptospirosis bovina es una importante enfermedad zoonótica cuyo diagnóstico molecular está ampliamente divulgado. Sin embargo, no existe un método único de extracción de ADN para leptospiras patógenas a partir de muestras clínicas. En este trabajo se utilizó orina bovina contaminada con cultivo de L. interrogans serovar Pomona Pomona para analizar el mejor método comparando: M1.) resina Chelex-100, M2.) papel FTA Whatman y M3.) hervido de la muestra (protocolo casero). De estas tres técnicas, la primera (M1) presentó la mayor sensibilidad al realizar la PCR de diagnóstico, detectándose hasta 2x102 leptospiras/mL. La metodología aquí planteada resultó tener buen rendimiento para la detección de leptospiras en muestras clínicas animales, aunque es necesario su validación con mayor número y diversidad de muestras.
Palabras clave: Diagnóstico molecular; Leptospira; Extracción de ADN.
SUMMARY
Comparison of simple and economic DNA extraction methods for molecular diagnosis of leptospirosis in animals
Bovine leptospirosis is an important zoonotic disease whose molecular diagnosis is widely reported. However, there is not a unique method of extraction of DNA for pathogenic leptospires using clinical samples. In this study, bovine urine was contaminated with pure culture of L. interrogans serovar Pomona Pomona in order to compare three of them: M1.) Chelex-100 resin, M2.) FTA Whatman paper and M3.) boiling of the sample (in-house protocol), being the first one the most sensitive when used in diagnostic PCR, detecting up to 2x102 leptospiras/mL. The methodology proposed in this study turned out to have good performance for the detection of leptospires in animal clinical samples, although it should be applied to a greater number of samples and in different stages of the pathology.
Keywords: Molecular diagnosis; Leptospira; DNA extraction.
La leptospirosis es una importante enfermedad infecto-contagiosa que causa pérdidas económicas debido a que produce abortos, muerte perinatal, nacimiento de crías débiles e infertilidad. Por otro lado, los animales infectados de forma crónica actúan como reservorios, liberando por orina leptospiras que contaminan el medio, sobre todo, las fuentes de agua, colaborando en la diseminación de la enfermedad (OIE, 2014). El método gold-standard para su diagnóstico es el Test de Micro-Aglutinación (MAT) que se basa en la detección y titulación de anticuerpos al enfrentar el suero a cultivos de leptospiras patógenas vivas. A pesar de que tiene la ventaja de diferenciar entre los diferentes serovares, es una técnica laboriosa que requiere mantener el cepario continuo y ser realizada por personal altamente calificado. Por otro lado, para detectar Leptospira spp. en orina en casos de infección aguda o portación crónica asintomática (reservorios), se realiza el aislamiento en cultivo, pero estos requieren entre 16 a 26 semanas de incubación y, con ello, el mayor riesgo de contaminación con otras bacterias. Los cultivos deben observarse bajo microscopio de campo oscuro semanalmente para control del desarrollo in vitro, resultando una técnica laboriosa que requiere operadores altamente capacitados. Esto refleja un método de diagnóstico lento, aunque es necesario aislar dicho agente para poder confirmar su presencia y circulación dentro del país.
Actualmente, la Reacción en Cadena de la Polimerasa (PCR), una técnica versátil en cuanto a sus diseños y a las muestras que se pueden emplear, ha permitido el desarrollo de protocolos de diagnóstico con alta sensibilidad y especificidad para diversos patógenos, entre ellos, las leptospiras. La mayor ventaja de la PCR radica en que es posible detectar el ADN incluso cuando las bacterias no son viables. El éxito de esta técnica depende del paso previo de extracción y purificación de ADN a fin de eliminar de la muestra posibles contaminantes e inhibidores de la reacción, manteniendo la calidad e integridad de la secuencia diana a amplificar, permitiendo trabajar con ADN parcialmente degradado o desnaturalizado.
Las técnicas clásicas de extracción se basan en el uso de solventes orgánicos, como fenol/cloroformo, pero son métodos dificultosos y que suponen un riesgo de exposición del operario a dichos solventes y la generación de desechos tóxicos. Otros procedimientos son tan sencillos como hervir la muestra (Olivera et al. 2010). Además, exiten kits comerciales que permiten obtener el ADN diseñados específicamente para un tipo de muestra (Noda y Rodríguez, 2014a), pero, al ser costosos, no todos los laboratorios pueden acceder a éstos.
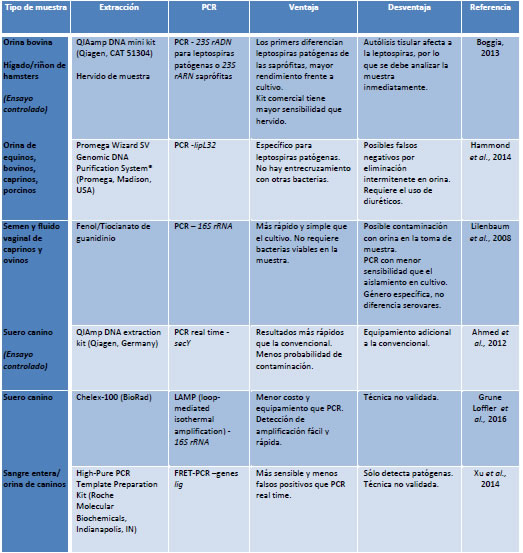
El diagnóstico molecular de leptospirosis se puede aplicar a diferentes tipos de muestra: orina, sangre entera, suero y órganos, sin embargo, no se encuentra establecido un único método de extracción y de PCR gold standard (Tabla 1). Es por esta razón que es necesario buscar métodos que aseguren la extracción de ácidos nucleicos de manera simple, rápida, sin perder sensibilidad, y económica. Siendo éste el objetivo principal del trabajo, se utilizaron tres métodos diferentes de extracción de ácidos nucleicos en un ensayo controlado para evaluar su sensibilidad: M1.) resina Chelex-100, M2.) papel FTA y M3.) hervido.
Tabla 1. Comparación de técnicas de diagnóstico por PCR utilizados en muestras clínicas animales.
La resina Chelex-100 es un quelante de iones metálicos polivalentes, los cuales pueden actuar como cofactores de aquellas enzimas que catalizan la ruptura del ADN a altas temperaturas. Al agregarla a la muestra previo a someterla a calor, este proceso queda impedido, además de eliminar otros inhibidores de la ADN Polimerasa (Noda y Rodríguez, 2014a). De esta manera el material genético obtenido tiene la suficiente integridad y es apto para la amplificación de la secuencia diana en el diagnóstico de leptospirosis.
El papel FTA Whatman contiene impregnado reactivos que permiten la disrupción celular con la consecuente adherencia del material genético, pudiendose sembrar muestras como sangre, orina, material vegetal, tejidos, plásmidos, células bucales, microorganismos (Fata et al. 2009; Wolfgramm et al. 2009), etc. Operariamente, el papel FTA tiene la ventaja de que, una vez sembrada la muestra y seco el papel, éste puede transportarse y guardarse a temperatura ambiente hasta realizar la extracción incluso años luego de recolectada (Osorio et al. 2013) por lo que ha sido ampliamente usado como método de almacenamiento de muestras. No obstante, supone un costo más alto comparado con la resina Chelex. Por otro lado, el hervido (M3) de la muestra, solamente supone el uso de equipamiento de laboratorio, el mismo para los otros dos métodos (centrífuga y termobloque) sin insumos adicionales, aunque carece de un paso de purificación del ADN.
En el ensayo controlado, se contaminó orina bovina con un cultivo puro de L. interrogans serovar Pomona Pomona, cepa de referencia con mayor casuística en el país (Draghi et al. 2011; Pavan et at., 2008). El cultivo utilizado se encontraba, según la escala McFarland, en 2x108 leptospiras por mililitro. Una vez realizadas las diluciones seriadas del cultivo, como se muestra en la Tabla 2, se procedió a la utilización de tres métodos de extracción y purificación de ADN.
Tabla 2: Diluciones del cultivo de leptospira realizadas en el ensayo controlado de orina.
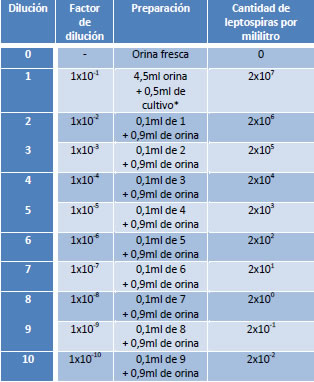
*Cultivo puro de L. interrogans serovar Pomona Pomona, en medio líquido EMJH de ocho días. El mismo se encontraba, según la escala McFarland, en 2x108 leptospiras por mililitro.
Método de extracción 1 (M1): resina Chelex-100 (Bio Rad). La resina fue preparada según las instrucciones del fabricante y se siguió el siguiente protocolo: a) en un tubo de 0,5mL, se pipetearon 150 μl de resina y se añadieron 20μl de orina; b) se incubó a 56ºC durante 20 minutos y 100ºC durante 8 minutos; c) se agitó vigorosamente durante 10 segundos; d) se centrifugó durante 2 minutos a 9,900g.; e) se tomó el sobrenadante como templado para PCR y se preservó a -20ºC para usos posteriores.
Método de extracción 2 (M2): papel FTA Whatman Classic Card (GE Healthcare Life Sciences) según el protocolo "FTA Eluate" modificado: a) se tomaron punchs (discos) de las tarjetas de FTA con Uni-Core de 3mm y se colocaron en tubos de 1,5ml; b) en cada tubo se agregaron 50μl de cada dilución de orina, se dejó reposar por 5 minutos, y luego se descartó el líquido; c) se secaron los punchs 5 minutos a 100°C; d) se añadieron 500μl de agua libre de nucleasas y se agitó usando vortex por 5 segundos; d) se descartó el sobrenadante y se agregaron 30μl de agua; e) se centrifugó 10 segundos; f) se calentó 15 minutos a 100°C; g) se agitó en vórtex 30 segundos; h) se centrifugó 30 segundos; i) se tomó el sobrenadante como templado para PCR y se preservó a -20ºC para usos posteriores.
Método de extracción 3 (M3): Hervido de la muestra (protocolo casero): a) se calentó la orina a 40ºC durante 10 minutos; b) se centrifugó a 700g por 15 minutos; c) se tomaron 100μl del sobrenadante y se colocaron en un nuevo tubo de microcentrífuga; d) se hirvió durante 15 minutos a 100ºC; e) se usó el templado para PCR y preservó a -20ºC para usos posteriores.
Se utilizó la PCR descripta por Gravekamp et al. (1993), empleando los primers G1 (5'-CTG AAT CGC TGT ATA AAA GT) y G2 (5'-GGA AAA CAA ATG GTC GGA AG). El tamaño de amplicón esperado fue de 285 pb correspondiente a un fragmento del gen secY. La mix de reacción fue preparada a un volumen final de 50μl, conteniendo 5ul de buffer 10X, 3mM MgCl2, 1,25U de ADN polimerasa Taq Pegasus (Productos BioLógicos, Argentina), 0,5μM de cada primer, 0,1mM de cada dNTP (Invitrogen, Carlsbad, CA, USA) y 2ul de templado de ADN obtenido según M1, M2 y M3. Las reacciones se llevaron a cabo en el termociclador MyCycler TM (BioRad), siguiendo el esquema: 94º C por 5 minutos, seguido de 35 ciclos de 94°C (1,5 min), 55°C (1 min) and 72°C (2 min), con una extensión final de 7 minutos a 72°C. Como control positivo se utilizó ADN de Leptospira interrogans serovar Pomona Pomona extraído con Chelex a partir del cultivo puro. Los productos de amplificación se analizaron por electroforesis en un gel de agarosa 2% teñido con bromuro de etidio. Se utilizó un transiluminador de luz UV (Uvi Tec transiluminator BTS- 20.M) para visualizar los amplicones. Los tamaños de las bandas se determinaron con un marcador de peso molecular de 100pb (Ladder 100pb precision, Productos BioLógicos, Argentina). Se cuantificó el amplicón utilizando QubitTM Fluorometer (Invitrogen, Carlsbad, CA, USA).
Durante la puesta a punto de la PCR diagnóstica que amplifica el gen secY, se secuenció el amplicón obtenido del control positivo (L. interrogans serovar Pomona Pomona) utilizando el servicio de Macrogen (Corea) obteniendo la siguiente secuencia: 5'-TGC CCA TCC AGC CCA TTC TTG ACT ACT ATT AGA TAA CCA TTG AAT AAT CGT CTG AGG AAA TAA AAT CAA AGA CGA AGC AAA AAT GAT CGG CAT CAC GTT CGC ACC GTT TAC TTT GAA AGG AAT AGA TTG ACT CTT GGC CTG AAC CAT TTT TCT TCC GAC CAT TTG TTT TCC A. Al realizar la búsqueda por BLASTN de la secuencia obtenida, se obtuvo una cobertura del 100% con la secuencia disponible en GenBank.
La sensibilidad de una PCR diagnóstica está dada por el éxito de su amplificación sobre las muestras clínicas, en este trabajo se ve reflejada la sensibilidad de esta PCR sobre un ensayo controlado contaminando orina bovina con un cultivo vivo. Luego de ensayar los tres métodos de extracción de ADN para la detección de leptospiras patógenas en orina contaminada y de realizar la PCR del gen secY, se observó que M1 (Chelex) fue el método con mejores resultados, lográndose detectar ADN leptospiral hasta la sexta dilución (10-6), representando un aproximado de 2x102 leptospiras/mililitro (correspondiente a 1,89μg/ml de ADN) (Figura 1a). Con el método M2, se obtuvieron resultados positivos hasta la dilución 10-3 (2.105 leptospiras/ml) (Figura 1-B). El método M3 arrojó resultados positivos de amplificación hasta la dilución 10-2, representando un aproximado de 2.106 leptospiras/ml. (Figura 1-C).

Figura 1. Resultados de PCR del gen secY utilizando los primers G1/G2. Gel de agarosa al 2% con las amplificaciones obtenidas utilizando como templado ADN obtenido a partir de las extracciones realizadas mediante resina Chelex-100 (A), papel de FTA (B) y hervido (C). La flecha indica hasta que dilución se pudo observar una amplificación por este método de PCR. CM: Marcador de peso molecular. Calles: 0: control negativo. 1-10: diluciones correspondientes. 11: control positivo.
En este trabajo, la resina Chelex tuvo una sensibilidad mayor de extracción en el ensayo controlado de orina, siendo el método que logró detectar la menor cantidad de ADN en comparación con los otros dos métodos cuando se sometió a PCR G1/G2. Se ha descrito el uso de la resina para la extracción de ADN de leptospiras a partir de otros tipos de muestra como sangre (Noda y Rodríguez, 2014a), tejidos humanos embebidos en parafina (Noda et al. 2014b); también para el diagnóstico de otros patógenos, como el Trypanosoma cruzi (López et al. 2014).
En el diagnóstico molecular de leptospirosis, es importante contextualizar el resultado de la PCR para una interpretación adecuada. Es decir, frente a un resultado negativo es necesario considerar desde alteraciones en la conservación de la muestra hasta factores propios de la patología, como el hecho de que es posible detectar la bacteria en sangre sólo en los estadíos agudos de la infección, ya que luego ésta se aloja en los riñones, momento a partir del cual, se pueden detectar las leptospiras en orina. Sin embargo, la eliminación es intermitente, por lo que se propone la administración de diuréticos para lograr una mayor eliminación de la bacteria (OIE, 2014). Por otro lado, en el caso de orinas ácidas de carnívoros, como caninos y humanos, es necesario neutralizar la orina con PBS hasta un pH de 7,6 previo a la extracción, ya que las leptospiras son susceptibles al medio ácido. En resumen, es importante tener en cuenta el origen y la anamnesis de la muestra para poder aplicar el mejor método de extracción y purificación del ADN para emplear una PCR diagnóstica.
Debido a la gran variación de metodologías moleculares para la detección de leptospirosis, en este trabajo se propuso a la resina Chelex como un método óptimo de extracción de ADN en conjunto con PCR para el abordaje del diagnóstico de la enfermedad, quedando pendiente la aplicación de ésta en un mayor número de muestras clínicas y su correlación con la patología.
Agradecimientos
Este trabajo fue financiado por el laboratorio de Leptospirosis del Instituto de Patobiología, CICVyA, CNIA, Instituto Nacional de Tecnología Agropecuaria (INTA). La Bqca. Micaela Hamer tiene una beca doctoral de nivel inicial otorgada por la Agencia Nacional de Promoción Científica y Tecnológica (ANPCYT), encuadrada en el proyecto PICT-Start up (2016-4815).
Bibliografía
1. Ahmed A, Klaasen HLBM, van der Veen M, van der Linden H, Goris MGA, Hartskeerl RA. 2012. Evaluation of real-time PCR and culturing for the detection of leptospires in canine samples. Adv. Mircobiol. 2: 162-170. [ Links ]
2. Boggia V. 2013. Diagnóstico de leptospirosis en bovinos mediante la reacción en cadena de la polimerasa. Tesis en licenciatura en ciencias biológicas. Universidad Nacional de Mar del Plata e Instituto Nacional de Tecnología Agropecuaria. [ Links ]
3. Draghi MG, Brihuega B, Benítez D, Sala JM, Biotti GM, Pereyra M, Homse A, Guariniello L. 2011. Brote de leptospirosis en terneros en recría en la provincia de Corrientes, Argentina. Rev. Arg. Microbiol. 43: 42-44. [ Links ]
4. Fata A, Khamesipour A, Mohajery M, Hosseininejad Z, Afzalaghaei M, Berenji F, Ganjbakhsh M, Akhavan A, Eskandari E, Amin-Mohammadi A. 2009. Whatman Paper (FTA Cards) for storing and transferring leishmania DNA for PCR examination. Iranian J. Parasitol. 4: 37-42. [ Links ]
5. Gravekamp C, van de Kemp, Franzen M, Carrington D, Schoone GJ, van Eys GJJM, Everard COR, Hartskeerl RA, Terpstra WJ. 1993. Detection of seven species of pathogenic leptospires by PCR using two sets of primers. J. Gen. Microbiol. 139: 1691- 1700. [ Links ]
6. Grune Loffler S, Leiva C, Scialfa E, Redondo L, Florin-Chistensen M, Martínez M, Romero G, Brihuega B. 2016. Detection of pathogenic leptospiral DNA traces in canine sera serum samples by Loop Mediated Isothermal Amplification (LAMP). Immunol. Infect. Dis. 4: 39-43. [ Links ]
7. Hamond C, Martins G, Loureiro AP, Pestana C, Lawson-Ferreira R, Medeiros MA, Lilenbaum W. 2014. Urinary PCR as an increasingly useful tool for an accurate diagnosis of leptospirosis in livestock. Vet. Res. Commun. 38: 81-85. [ Links ]
8. Lilenbaum W, Varges R, Brandao FZ, Cortex A, deSouza SO, Brandao PE, Richtzenhain LJ, Vasconcellos SA. 2008. Detection of Leptospira spp. in semen and vaginal fluids of goats and sheep by polymerase chain reaction. Theriogenology 69: 837- 842. [ Links ]
9. López M, Rivera MG, Viettri M, Lares M, Morocoima A, Herrera L, Ferrer E. 2014. Comparación de dos protocolos de extracción de ADN de Trypanosoma cruzi cultivados en medio axénico. Rev. Peru. Med. Exp. Salud Publica 31: 222-227. [ Links ]
10. Noda AA, Rodríguez I. 2014a. DNA isolation by Chelex-100: an efficient approach to consider in leptospirosis early stages. J. Coast. Lif. Med. 2: 501-504. [ Links ]
11. Noda AA, Rodriguez I, Rodríguez Y, Govín A, Fernández C, Obregón AM. 2014b. High sensitive PCR method for detection of pathogenic Leptospira spp. in paraffin-embedded tissues. Inst. Med. Trop. Sao Paulo. 56: 411-415. [ Links ]
12. OIE (World Organization for Animal Health). 2014. Leptospirosis. En: Manual of diagnostic tests and vaccines for terrestrial animals. OIE, Paris. [ Links ]
13. Oliveira ST, Messick JB, Biondo AW, Santos AP, Guimaraes AMS, Mohamed AS, Pires Neto JAS, Dalmolin ML, Gonzalez FHD. 2010. Serum and urinary C-reactive protein concentractions in dogs with leptospirosis. Acta Scientiae Veterinariae 38: 245-249. [ Links ]
14. Osorio J, Pachajoa H, Hurtado P. 2013. Concentración y pureza del ADN de muestras sanguíneas en papel Whatman FTA almacenadas entre 1 a 3 años. Rev. Estomatol. Salud 21: 35-38. [ Links ]
15. Pavan ME, Cairó F, Brihuega B, Samartino L. 2008. Multiple-locus variable-number tandem repeat analysis (MLVA) of Leptospira interrogans serovar Pomona from Argentina reveals four new genotypes. Comp. Immunol. Microbiol. Infect. Dis. 31: 37-45. [ Links ]
16. Wolfgramm EV, Carvalho FM, Aguiar VRC, Sartori MPN, Hirschfeld-Campolongo GC, Tsutsumida WM, Louro ID. 2009. Simplified buccal DNA extraction with FTA Elute Cards. Ferensic Sci. Int. Genet. 3: 125-127. [ Links ]
17. Xu C, Loftis A, Ahluwalia SK, Gao D, Verma A, Wang C, Kaltenboeck B. 2014. Diagnosis of canine leptospirosis by a highly sensitive FRET-PCR targeting the lig genes. PLoS ONE 9: e89507. [ Links ]














