Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista de Ciencia y Tecnología
versión On-line ISSN 1851-7587
Rev. cienc. tecnol. no.17 Posadas ene./jun. 2012
GENÉTICA-BIOLOGÍA
Amplificación interespecífica de microsatélites de Astyanax abramis, utilizando cebadores para la especie emparentada Astyanax fasciatus
Interspecific amplification de Astyanax abramis microsatellites using primers for the related astyanax fasciatus
Alejandra Paola Ojeda, Lourdes M. Hirt, Pedro Darío Zapata.
1. Laboratorio de Biotecnología Molecular
2. Laboratorio de Biología General. Facultad de Ciencias Exactas, Químicas y Naturales. Universidad Nacional de Misiones. Posadas, Misiones, Argentina. Email: biotecmol2010@gmail.com
• Lourdes Hirt2
Es Licenciada en Genética por la UNaM y Doctorado en Ciencias Biológicas en la UNNE. Actualmente es Profesora Adjunta Regular de la Cátedra de Biología General de las carreras de Bioquímica, Farmacia y Genética desde el año 1992. Es profesora del Doctorado en Ciencias Aplicadas y de la Maestría en Gestión Ambiental de la FCEQyN. Se desempeña como Investigadora y Directora de Proyectos de investigación relacionados con la ecología y los recursos pesqueros, habiendo obtenido diversas financiaciones de agencias nacionales a modo de subsidios. Dirige auxiliares, becarios de grado y posgrado. Posee publicaciones en revistas indexadas y presentaciones a congreso.
• Pedro Darío Zapata1
Es Doctor por la Universidad de Alcalá de Henares. Se desempeña actualmente como Profesor Regular Adjunto en las Cátedras de Biología Celular y Molecular (Bioquímica), Genética Molecular (Lic. en Genética), Biología Celular (Lic. en Genética) y Biotecnología Molecular (Ingeniería Química, Bioquímica y Farmacia). Ha sido beneficiario de subsidios de agencias gubernamentales. Dirige auxiliares, becarios de grado y posgrado. Posee publicaciones en revistas indexadas nacionales e internacionales y presentaciones a congresos nacionales e internacionales.
• Alejandra Paola Ojeda1
Es Licenciada en Genética, egresada de la Universidad Nacional de Misiones. Integrante de algunos proyectos relacionados con la ecología y las aplicaciones la biología molecular a este campo, especialmente la diversidad de peces. Actualmente desarrolla estudios de posgrado en Brasil.
Resumen
La reserva de la Biosfera Yabotí se localiza en el centro-este de la Provincia de Misiones (Argentina) y cubre un amplio sector que comprende el arrojo Yabotí y sus efluentes. Astyanax es un importante eslabón en la cadena trófica y en el mantenimiento de las comunidades ícticas naturales. Los estudios poblacionales son datos ecológicos que permiten el análisis sobre el manejo sustentable del recurso y las regiones microsatélites representan marcadores de gran utilidad en este sentido. Sin embargo aun no existen datos genómicos de estas regiones en esta especie, siendo conocidos únicamente SSR para la especie emparentada Astyanax fasciatus. El objetivo de este trabajo fue lograr la amplificación interespecífica de regiones de microsatélites en Astyanax abramis utilizando cebadores diseñados para A. fasciatus. Se extrajo DNA de tejido muscular del pedúnculo caudal de 13 individuos. Se estandarizaron las condiciones para amplificar 4 regiones microsatélites en A. abramis: Abral, Abra2, Abra3 y Abra4, de los cuales 3 fueron secuenciados y registrados en Genebank. Nuestros hallazgos confirman la transferibilidad de los cebadores utilizados, reconociendo secuencias conservadas y permitiendo establecer las primeras secuencias microsatélites de la especie estudiada.
Palabras clave: Astyanax abramis; SSR; Amplificación interespecífica.
Abstract
The Yabotí biosphere reserve is located in the center-east of Misiones (Argentina) and it covers a vast area of the basins of Yabotí streams, tributaries of Alto Uruguay River. Astyanaxis important in the trophic level and in the maintenance of the natural fish communities, since it is food for the last links of the food chain. Population analysis are important ecological data for supporting the reasonable use of the resource. Futhermore, there are no yet specific data for SSR regions in Astyanax abramis, only for Astyanax fasciatus. The objective of this work was to cross-amplify the microsatellites regions of in Astyanaxabramiswith specific primers designed for A. fasciatus. DNA was extracted from the caudal fin peduncle from 13 specimens. The PCR conditions were standardized to extend 3 microsatellite regions in A. abramis: Abral, Abra2, Abra3 y Abra4. Three of these were sequenced and registered in the Genebank database. Although our findings are preliminary, they confirm the transferability of the use of primers, identifying sequences preserved and allowing us to establish the first microsatellite sequence of the species under study.
Key words: Astyanax abramis; SSR; Cross-amplification.
Introducción
La Reserva de la Biosfera Yabotí (RBY) se localiza en el centro-este de la Provincia de Misiones (Argentina) entre las coordenadas E 53° 40', W 54° 18', N 26° 37' y S 27° 12' y cubre un amplio sector de las subcuencas de los arroyos Yabotí, tributarios del Alto Uruguay, todavía cubierto de selvas en un buen estado de conservación. Entre los cursos fluviales de la zona se encuentran el arroyo Yabotí o Pepirí-Miní integrado por 2 grandes cursos paralelos que se unen a unos 10 km al norte de su desembocadura en el río Uruguay: el Yabotí-Guazú al oeste y el Yabotí Miní al este, poseedores de una vasta red imbrífera que actúa como columna vertebral de la reserva que merecidamente lleva este nombre (Yabotí en lengua tupí significa Tortuga). Hirt y col. [1] efectuaron una evaluación de la fauna íctica del arroyo Yabotí, siendo los resultados de este trabajo un aporte para el conocimiento de la biodiversidad de los cuerpos de agua de la RBY. Desde el punto de vista de la composición y diversidad, se reconocieron 40 especies agrupadas en 4 órdenes y 12 familias. Tanto en términos de diversidad taxonómica como abundancia, el orden Characiformes fue el grupo más representativo, con la especie Astyanax abramis (mojarra) ocupando el noveno lugar en abundancia. La importancia en el estudio de la especie radica en ser un importante eslabón en la cadena trófica y en el mantenimiento de las comunidades ícticas naturales [2], siendo alimento de los últimos eslabones tales como el dorado (Salminus brasiliensis), la tararira (Hoplias malabaricus) y los surubíes (Pseudoplatystoma coruscans y P fasciatum) [3].
El orden Characiformes (peces con escamas) incluye a la familia Characidae, donde se encuentra el género Astyanax caracterizado por un cuerpo comprimido, colorido y más o menos largo [4]. De amplio hábito alimentario, pudiendo ser zooplanctívoro, insectívoro, omnívoro y de carácter oportunista [3, 5].
Astyanax abramis posee cuerpo alto, cabeza pequeña y ojos grandes (Figura 1). Escamas más bien grandes, regularmente imbricadas en el dorso y en los flancos. Mancha humeral alargada horizontalmente y con una estría oscura extendiéndose hacia arriba y adelante. Línea lateral con 38-49 escamas. Aleta anal con 22-34 radios. Pre-maxilar con 4 a 5 dientes en la fila externa, maxilar sin dientes. Con una banda plateada bien desarrollada, una mancha caudal marcada, angosta sobre los radios caudales medios y prolongada hacia delante pero muy fina y poco visible.

Figura 1: Características morfológicas de Astyanax abramis.
Los marcadores moleculares son herramientas de la biología molecular que facilitan el estudio de poblaciones, especialmente aquellas que muestran una gran variabilidad, como son las regiones microsatélites (SSR). Los micro-satélites constituyen marcadores moleculares basados en la presencia de secuencias repetitivas de DNA, representan una proporción importante del genoma de organismos eu-cariotas y son ampliamente usados en la caracterización de poblaciones por ser codominantes, multialélicos, altamente polimórficos y por la facilitad técnica con la que pueden analizarse utilizando cebadores específicos complementarios a las secuencias únicas que lo flanquean y visualizándoselos mediante electroforesis en geles de agarosa o de poliacrilamida [6]. Existen trabajos que describen el uso transespecífico de marcadores microsatélites entre grupos taxonómicos relacionados a distintos niveles [7].
Esto agrega un valor potencial a las características de estos marcadores ya que al trabajar con microsatélites en una especie en particular, pueden ser usados en taxa relacionados. Algunas especies del género Astyanax han sido estudiadas utilizando marcadores moleculares como microsatélites y RAPD en otras regiones de Brasil y México. [8, 9, 10, 11]
Strecker y col. identificaron 6 microsatélites para Astyanax fasciatus y los utilizaron para la caracterización de poblaciones, demostrando que su validez como herramientas moleculares para analizar la variabilidad genética intra e ínterpoblacional de esta especie [11]. Sin embargo, son escasos los estudios con especies autóctonas de la zona mencionada, en la región del arroyo Yabotí, existiendo como antecedente el trabajo de Amarilla y col., 2008, quienes estandarizaron técnicas de extracción de DNA y con una metodología similar lograron la amplificación cruzada de microsatélites para Astyanax saguazu [12].
Partiendo de la característica de transferibilidad de estos marcadores y la descripción de 6 microsatélites y sus correspondientes cebadores para A. fasciatus por Strecker y col. [11] en este trabajo se evalúa la transferibilidad de estos marcadores microsatélites a través del uso de los cebadores descriptos para A. fasciatus para el estudio de microsatélites de A. abramis.
Materiales Y Métodos
Área de estudio, obtención de ejemplares e identificación taxonómica de Astyanax abramis
Los individuos provienen de capturas realizadas en el arroyo Yabotí Guazú en 3 sectores: la cuenca alta (26° 50'11"S, 54° 07'50,7"W, media (27° 03'29"S, 53° 53'03"W) y desembocadura (27° 08'29"S, 53° 55'50"W), por el grupo que integra el proyecto "Una alternativa turística para la reserva Yabotí: la fauna íctica y su uso potencial en la pesca deportiva con devolución. La comunidad íctica del arroyo Yabotí". Los ejemplares fueron capturados vivos con redes de pesca caladas per-pendicularmente a la dirección de la corriente del arroyo, procediéndose a su eutanasia con cloroformo y posterior conservación en frascos con etanol 95% hasta la identificación taxonómica donde con claves, instrumentos de disección y ópticos [9]. Esta clasificación fue confirmada por investigadores del ILPLA, Instituto de Limnología de La Plata (Bs. As). Para los estudios moleculares se realizaron cortes transversales y longitudinales de tejido muscular de pedúnculo caudal según Strecker y col. [11].
Extracción de DNA
Se realizó a partir de tejido muscular de pedúnculo caudal del ejemplar a través de cortes transversales y longitudinales [11, 13]. Para la extracción y purificación se utilizó el protocolo desarrollado por el Dr. Alejandro D'Anatro (comunicación personal) para Plagioscion ternetzi (corvina), modificación del método Salting Out de Miller y col. [14]. Veinte mg de tejido fueron digeridos a 55°C durante toda la noche con buffer de lisis (50 mM de Tris HCl, 50 mM de NaCl, 50 mM EDTA ph 8) conteniendo 1% de dodecilsulfato de sodio (SDS) y 10 de proteinasa K 1 mg/ml. Posteriormente el DNA fue purificado con NaCl 5 M y precipitado con isoporpanol en proporción 1:1. El DNA fue lavado con etanol 70%, disuelto en agua libre de DNasas y RNasas y conservado a -20°C hasta su utilización. La concentración y pureza del DNA se cuantificó por espectrofotometría. La concentración de DNA de cada aislamiento se calculó a través de la absorbancia a 260 nm. (A260) mediante la relación: [AcNu]= 50 ng/^l x D x A260, donde D representa el factor de dilución y 50 ng/^l es la concentración de DNA de doble cadena. El índice de pureza se calculó a través de la relación A260/A280 [15]. La integridad del DNA se verificó en geles de agarosa al 1%. El DNA obtenido se diluyó para obtener una concentración de trabajo de aproximadamente 10 ng/^l.
Amplificación por PCR
Para estandarizar las condiciones de PCR para A. abramis se procedió a realizar un panel de reacciones, iniciándose las mismas por las condiciones descriptas previamente por Strecker y col. [11] para A. fasciatus utilizando los pares de cebadores descriptos para Ast1, Ast2, Ast4 y Ast10 (Tabla1).
Tabla 1: Características de los cebadores utilizados.

La PCR se desarrolló sobre 100 ng de DNA en un volumen final de 12,5 ul de Buffer de Taq Polimerasa (Fermentas ®) conteniendo 0,3 U de Taq polimerasa, 2 mM de MgCl2, 200 uM de dNTPs y 0,5 pmol de cada cebador. Las condiciones de ciclado utilizadas por este grupo fueron: desnaturalización previa a 95°C por 1 min, seguidos de 40 ciclos de desnaturalización a 95°C por 15 s, hibridación a temperatura locus específico por 30 s y extensión a 72 °C por 30 s. Los cebadores Ast1, Ast2, Ast4 y Ast10 hibridan en esta especie a 55 °C, 50°C, 55 °C y 50°C respectivamente. Las reacciones fueron realizadas en un termociclador Life Cycler (Labnet). Los productos de la amplificación fueron separados mediante electroforesis en geles de agarosa al 2%, teñidos con bromuro de etidio (10mg/ml, Promega) y corridas a 100V por 30 minutos con buffer TBE. Posteriormente, fueron visualizados en un transiluminador de luz UV (MUV21-312-220) y fotografiados con cámara digital ACER (CS-5531). Como marcador de tamaño molecular fue utilizado el 100 pb Ladder de Promega.
Secuenciación de las regiones de microsatélites amplificadas
La finalidad de la secuenciación fue la confirmación de la identidad de las regiones encontradas y el inicio de la caracterización genómica de la misma. Para ello se seleccionaron individuos homocigotos para cada uno de los loci, los amplicones se purificaron y enviaron a secuenciar. Los resultados de la secuenciación se analizaron con el programa Chromas Lite, el BLAST (Basic Local Alignment Search Tool) y el Find Tandem Repeats (http:// www.biophp.org/minitools/microsatellite_repeats_finder) disponibles on line, y una vez confirmadas fueron registradas en GenBank.
Resultados y discusión
Identificación taxonómica de Astyanax abramis
De los 13 ejemplares de A. abramis identificados, 2 integran la colección íctica del Proyecto de Investigación citado y los 11 ejemplares restantes fueron utilizados para este trabajo. Astyanax es uno de los géneros más comunes en Sudamérica con un número incierto de especies [16]. Garutti y Britski [17] consideraron la existencia de 100 especies nominales, mientras que lo estimado por Bertaco y Malabarba [18] es de 74 especies y subespecies. Desde el punto de vista taxonómico es un taxón complejo ya que presenta características muy similares entre sus especies lo que dificulta su identificación. Se lo define tradicionalmente sobre caracteres morfológicos, como patrón de coloración, número de radios en aletas y especialmente el patrón de dentición [16, 17].
Amplificación de regiones microsatélites
Los microsatélites presentan varias ventajas como herramienta, pero admiten una característica como valor agregado, la posibilidad de utilizar los cebadores desarrollados para una especie en otras emparentadas, esto disminuye el esfuerzo temporal y económico que involucra la construcción de cebadores específicos mediante la elaboración de librerías genéticas [11, 19, 20, 21]. Se lograron productos de amplificación utilizando 4 pares de cebadores de los 6 descriptas previamente para A. fasciatus: Ast1, Ast2, Ast4 y Ast10 [11], aunque fue necesario reajustar las temperaturas de hibridación por tratarse de una especie diferente (Ast1, Ast2, Ast4 y Ast10 hibridaron en esta especie a 55 °C, 50 °C, 55 °C y 50 °C respectivamente). Luego de los ensayos de prueba se definieron las condiciones de ciclado. Como los cebadores corresponden a otra especie del mismo género se ensayaron temperaturas de hibridación de locus específico distintas a las descriptas anteriormente, aunque el número de ciclos fue el mismo. (Tabla 2)
Tabla 2: Comparación de las condiciones de amplificación para los cebadores analizados entre las especies A. fasciatus y A. abramis.
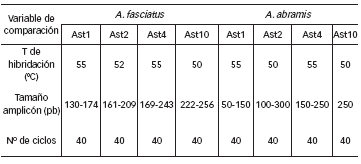
Las condiciones de PCR para A. abramis difieren de las establecidas para A. fasciatus [11] dado que se necesitó incrementar los tiempos de desnaturalización y adicionar un ciclo de extensión final, coincidiendo con las características de ciclado descriptas por Ivanova y col. [19] al trabajar con numerosas taxa de peces. Al comparar las temperaturas de hibridación vemos que los cebadores Astl hibridan a menor temperatura que en A. fasciatus. Cuando se utilizaron los cebadores Ast2 y Ast4 fue necesario un aumento de la temperatura de hibridación debido a los productos inespecíficos obtenidos a temperaturas menores; sin embargo, Ast10 amplifica en ambas especies a la misma temperatura. Los mejores resultados se obtuvieron con una disminución en el número de ciclos.
Por otro lado, para los cebadores Astl, Ast2, Ast4 y Ast10 Strecker y col. [11] describe amplicones en los siguientes tamaños en pares de bases: 130-174; 161-209; 169-243 y 222-256 respectivamente; mientras que en los individuos analizados en este trabajo encontramos amplicones en los siguientes tamaños en pares de bases: 50-150; 100-300; 150-250 y 250 para los mismos cebadores (Figura 2). Estos rangos no son exactamente coincidentes y esta diferencia se explica al considerar que se trata de secuencias heterólogas, descriptas para otra especie.

Figura 2: Amplificación de microsatélites obtenidos para A. abramis. Los productos de PCR fueron separados mediante electroforesis en agarosa al 2% teñida con bromuro de etidio. Los carriles muestran la amplificación para los 11 individuos analizados. Las flechas indican los productos de amplificación que fueron tomados como específicos. Carril 1: Control negativo. Carril 2 al 12: individuos 1 al 11. Carril 13: marcador de peso molecular.
Secuenciación y análisis de las regiones de microsatélites amplificadas
Los amplicones obtenidos para los pares de cebadores Astl, Ast4 y Ast10 fueron secuenciados para corroborar la presencia de regiones microsatélites, para ello se seleccionaron individuos homocigotos. Estas secuencias fueron registradas en la base de datos GenBank del NCBI (National Center for Biotechnology Information) y analizadas con la herramienta bioinformática BLAST (Basic Local Alignment Search Tool) disponible on line en el NCBI. [20, 21].
Los resultados bioinformáticos indican que el amplicón obtenido con los cebadores Ast4 muestra una secuencia microsatélite rica en regiones (CA)n mostrando más de un 90% de identidad con otras regiones microsatélites pertenecientes a especies de peces (Cyprinus carpio, Glyptosternon maculatum, Astatotilapia burtoni) y a especies no emparentadas como el molusco Oncomelania hupensis, el crustáceo Penaeus monodon, el cérvido Cer-vus nippon, la especie vegetal Arenaria grandiflora y el hongo Phytophthora cinnamomi. Esto indica que se trata de un microsatélites nuevo para la especie en estudio por lo que se lo registró con el nombre de Abra1 bajo el código GeneBank GU942469.3.
Para el caso del amplicón obtenido con los cebadores Ast10 la alineación la secuencia correspondiente de A. fasciatus (AY178852.1) y el resultado bioinformático indica una concordancia con el patrón de secuencia microsatélite propuesto por Strecker y col [11], (GT)2GC(GT)14. Además, mostró identidad con regiones microsatélites pertenecientes a trucha común (Salmo trutta) y al árbol Cercis canadensis, mostrando cierta similitud con algunos genes mitocondriales pertenecientes al citocromo b. Esta secuencia fue registrada con el nombre de Abra3 bajo el código GeneBank JF714418.1.
Por otro lado, la secuencia Abra2, registrada con el código de acceso GeneBank JF312216.1 y que fuera obtenida con el uso de los cebadores Astl no muestra identidad con ninguna secuencia microsatélite conocida, aunque el análisis bioinformático indica que podría tratarse de una secuencia microsatélite, por lo que deberá profundizarse en su estudio, ya que el resultado de la secuenciación fue sumamente corto, siendo en este caso conveniente la clonación del fragmento.
Nuestros hallazgos confirman la transferibilidad de los cebadores Astl, Ast4 y Ast10 utilizados en este trabajo, reconociendo secuencias conservadas y permitiendo establecer las primeras secuencias microsatélites de la especie A. abramis.
Si bien es necesario aumentar el número de muestras para alcanzar resultados válidos desde el punto de vista poblacional, en este describen nuevos marcadores Abral, Abra2 y Abra3 que pueden ser utilizados con este fin sobre A. abramis. Igualmente, es destacable que con un bajo número de muestras tanto Abral como Abra2 mostraron un interesante número de individuos heterocigotos. Los estudios de variabilidad genética en el género Astyanax del arroyo Yabotí representan un dato importante para determinar el manejo del recurso pesquero en esta cuenca. A. abramis se encuentra dentro de especies consideradas vulnerables por Gómez y col, [22], por ser una especie ornamental, objeto de comercialización sin claras regulaciones ni controles adecuados. Estas especies son también utilizadas como carnada de especies de gran porte como el dorado (Salminus brasiliensis), la tararira (Hoplias mala-baricus) y los surubíes (Pseudoplatystoma coruscans y P. fasciatum), tanto en la pesca deportiva como comercial, al constituir el alimento principal de estas especies carnívoras. Este es un trabajo original, de importancia regional, donde se identificaron por primera vez marcadores moleculares de A. abramis que pueden ser usados en estudios futuros sobre el cauce del arroyo Yabotí. Diversos estudios demuestran que este tipo de marcador es ideal para el estudio de la diversidad genética en organismos acuáticos por su alto nivel de variabilidad alelica y de esta manera servir como un soporte molecular para el desarrollo de la acuicultura [22, 23], para el análisis poblacional y para la conservación de la biodiversidad [24, 25].
1. Hirt, L., Araya, P., Flores, S., López, G., Ojeda, P., Serrano, M. y Amarilla, L., Una alternativa turística para la Reserva Yabotí: la fauna íctica y su uso potencial en la Pesca Deportiva con devolución. INFORME ETAPA 6. Convenio 11/06 SCTIP-UNaM. 2009. [ Links ]
2. Vazzoler, A., Agostinho, A. y Segatti Hahn, N., A Planicie de Inundagäo do Alto Rio Paraná. Ed. EDUEM, Brasil. 460p. 1997. [ Links ]
3. Hahn, N. S., Fugi, R., Almeida, V. L. L, Russo, M. R. & Loureiro, V. E. 1997. Dieta e atividade alimentar de peixes do reserva-tório de Segredo. Pp. 141-162. In: Agostinho, A. A. & Gomes, L. C. (Eds.). Reservatório de Segredo: bases ecológicas para o manejo. EDUEM, Maringá, 387p.
4. Baird, S. y Girard, C., Descriptions of some new fishes from the river Zuni. Proc. Acad. Nat. Sci. Phyla. USA, 6: p 368-369. 1854.
5. Arcifa, M. y Meschiatti, M., Distribution and feeding ecology of fishes in a tropical Brazilian reservoir. Soc.Ciec. Nat.48:p 302-313. 1993.
6. Posso Duque y Ghneim Herrera, Uso de marcadores micro-satélites para la estimación de diversidad genética en plantas. Manual de laboratorio. Caracas, Venezuela. 48p. 2006.
7. Wang M.L., Barkley, N.A., Yu, J.K., Dean, R.E., Newman, M.L., Sorrells, M.E. y Pederson, G.A., Transfer of simple sequence repeat (SSR) markers from major cereal crops to minor grass species for germplasm characterization and evaluation. Plant Genetic Res. 3: p 45-57, 2005.
8. Prioli, S.M.A.P., Prioli A.J., Julio Jr., H.F., Pavanelli, C.S., de Oliveira, A.V., Carrer, H., Carraro, D.M. y Prioli, L.M., Identification of Astyanax altiparanae (Teleostei, Characidae) in the Iguacu River, Brazil, bases on mitochondrial DNA and RAPD markers, Gen. Mol. Biol. 25: p421-430, 2002.
9. Bachair Moysés, C.B. y de Almeida-Toledo, L., Restriction fragment length polymorphisms of mitochondrial DNA among five freshwater fish species of the genus Astyanax (Pisces, Characidae), Gen. Mol. Biol. 25:p 401-407, 2002.
10. Papa-Leuzzi, M.S., Simoés de Almeida, F., Orsi, M.L., Koelblinger-Sodré, M., Analysis by RAPD of the genetic structure of Astyanax altiparanae (Pisces, Characiformes) in reservoirs on the Paranapanema River, Brazil, Gen. Mol. Biol. 27:p 355-362, 2004.
11. Strecker, u, Polymorphic microsatellites isolated from the cave fish Astyanax fasciatus. Mol. Ecol. Notes 3: p150-151. Hamburg, Germany. 2003.
12. Amarilla, L.; Hirt, L. y Zapata, P., Caracterización de marcadores microsatelitales en Astyanax saguazu (Pisces, Characiformes) del Arroyo Yaboti Guazú (Misiones, Argentina). 13er Congreso latinoamericano de Genética, VI Congreso de Genética. Lima. Perú. 2008.
13. Wasco, A. P., Martins, C., Oliveira, C. y Foresti, F., Non destructive genetic in fish. An improved method for DNA extraction from fish and scales. Hereditas 138, p161-165, 2003.
14. Miller, S. Dykes, D. y Polesky, F., A Simple salting out procedure for extracting DNA from human nucleated cells. Nucl. Ac. Res. 16: p1215. 1988.
15. Cariaga Martinez, A. y Zapata, P., El laboratorio de biología molecular. Editorial Universitaria de Misiones. Posadas. 104p. 2005.
16. Casciotta, J., Almirón, A. y Azpilicueta, M., A new Species of Astyanax from Río Uruguay Basin, Argentina (Charciformes: Characidae). Ichthyol. Explor. Fres-hwaters.14: p 329-334. 2003.
17. Garutti, V y Brisky, H., Descrigäo de uma Espécie Nova de Astyanax altiparanae (Teleostei, characidae) da Bacia do alto do rio Paraná e Concideragoes sobre As Demais Espécies do Genero na Bacia. Comum. Mus. Cienc. Tecnol. PUCRS 13:p 65-88. 2000.
18. Bertaco, V. y Malabarba, R., Description of two new Species of Astyanax (Teleostei:Characidae) from Headwater Stream of Southern Brazil, with Components on the A. scabripinis Species Complex. Ichthyol. Explor. Freshater.12:p 221-234, 2001.
19. Ivanova, N., Zemelak, T., Hanner, R. y Hebert, P., Universal primer for fish DNA barcoding. Mol. Ecol. Notes 7:p 544-548, 2007.
20. Spooner, D., Van Treuren, R. y De Vicente, M.C., Molecular markers for genbank management. IPGRI Technical Bulletin. 126 p. 2005.
21. Ferreira, M. y Grattapaglia, D., Introducao ao uso de marcadores moleculares em analisis genetico. EMBRAPA. 220 p. 1996.
22. Gomez, S.E. y Chevez, J.C., Peces de la Provincia de Misiones. Capítulo 4 en: J. C. Chebez, Fauna Misionera. Catálogo Sistemático y zoogeogràfico de los vertebrados de la Provincia de Misiones (Argentina). L.O.L.A., Buenos Aires: p 38-70. 1996.
23. Bruford, M. W. y Wayne, R. K., Microsatellites and their application to population genetic studies. Curr. Op. Genet. Develop., 3:p 939-934. 1993.
24. Yan, J., Liu, S., Sun, Y., Zhang, C., Luo K. y Liu., Y., RAPD and microsatellite analysis of diploid gynogens from allo-tetraploid hybrids of red crucian carp (Carassius au-ratus) Xcommon carp (Cyprinus carpio). Aquaculture 243:p 49-60. 2005.
25. Romana-Eguia, M.R.R., Ikeda, M., Basiao Z.U. y Taniguchi. N., Genetic diversity in farmed Asian Nile and red hybrid tilapia stocks evaluated from microsatellite and mitocondrial DNA analysis. Aquaculture 236:p 131-150. 2004.
Recibido: 07/05/2012
Aprobado: 02/07/2012














